| 크리스퍼 유전자가위와 인공유전자회로를 이용한 질병의 진단과 치료 | |
|---|---|
| Date 2018-04-15 15:51:15 |
|

선임연구원
생명공학연구원 바이오합성연구센터
dhlee@kribb.re.kr
1. 서론
합성생물학은 2000년대 초 인공유전자회로에 대한 두 편의 논문이 발표되면서 시작되었다. 두 개의 인공유전자회로는 유전자 토글 스위치(genetic toggle switch)와 유전자 발현 오실레이터(gene expression oscillator, repressilator)이다. 이러한 인공유전자회로는 발전을 거듭하여 의약품 전구체, 산업 바이오소재 등을 생산하는 대사공학에 성공적으로 활용되어 왔다. 최근에는 제3세대 유전자가위인 크리스퍼 유전자가위 기술과 접목하여 외부 신호를 감지하여 세포 대사를 제어하려는 연구가 많이 이루어지고 있다. 이러한 인공 유전자회로를 통해 외부 신호를 감지하고, 감지된 신호에 의해 세포 제어가 시도되고 있는 대표적인 연구 분야는 인간 마이크로바이옴 엔지니어링이다. 인간 마이크로바이옴 엔지니어링은 우리의 건강에 매우 중요한 역할을 하는 마이크로바이오타로부터 유익한 미생물(probiotics)을 발굴하거나 이러한 미생물의 생육에 유리한 환경을 조성하는 연구가 주를 이루고 있었다. 그러나 최근에는 적극적으로 질병의 진단과 치료를 위해 합성생물학의 최신 기술을 도입하여 인간 마이크로바이오타를 제어하려는 연구가 시작되고 있다. 이에 본 기고에서는 질병의 진단과 치료를 위한 인공유전자회로와 크리스퍼 유전자가위의 합성생물학적 활용에 관한최신 연구 동향을 소개하고자 한다.
2. 본론
2.1 크리스퍼 유전자가위를 이용한 유전체 편집 및 유전자 발현 조절
제3세대 유전자가위 기술은 미생물의 적응 면역(adaptive immunity)의 일종인 크리스퍼(clustered regularly interspaced short palindromic repeats, CRISPR)-카스(CRISPR-associated, Cas) 시스템으로부터 유래했다. 크리스퍼-카스(CRISPR-Cas)는 바이러스 감염 시 숙주 세포 게놈에 바이러스 DNA 일부를 저장하고, 이 저장된 DNA 서열로부터 crRNA를 발현하여 특정한 카스 단백질과 결합한 후 바이러스 DNA 혹은 RNA를 파괴하는 시스템이다. 현재 크리스퍼-카스 시스템은 계통학적으로 2개의 클래스(class)와 6개의 유형(type)으로 분류되고 각 유형은 다양한 구조와 기능을 갖고 있지만 여기에서는 합성생물학 분야에서 가장 많이 사용되는 2형 크리스퍼-카스9(CRISPR-Cas9) 시스템 중심으로 소개하겠다[1].
크리스퍼-카스9 시스템은 카스9 단백질과 sgRNA (single guide RNA)로 구성된다. 카스9 단백질에는 DNA를 절단할 수 있는 endonuclease 활성이 존재하고 sgRNA는 목적 DNA에 결합할 수 있는 20 bp 정도의 스페이서(spacer)서열이 존재한다. 이 스페이서를 원하는 목적 DNA 서열에 상보적으로 디자인하면, 카스9/sgRNA 복합체가 목적 DNA에 결합하여 절단하는 크리스퍼 유전자가위가 제작된다. 크리스퍼 유전자가위에 의해 동물 세포 게놈에서 이중가닥 절단(double strand break)이 발생하면 비상동 말단 연결(non-homologous end joining, NHEJ)에 의해 절단된 DNA가 수선되면서 변이가 발생한다. 만일 절단된 위치의 서열과 상동성을 갖는 주형 DNA(template DNA)가 존재하면 상동 의존 수리(homology-directed repair, HDR)를 통해 수선될 수 있다. 이 현상을 이용하면 원하는 유전자를 결손(knock-out)하거나 다양한 유전자를 원하는 위치에 삽입(knock-in)하는 등과 같은 유전체 편집(genome editing)이 가능하다. 카스9 단백질의 생화학적 특성 및 모듈성(modularity)을 이용하면 유전체 편집 뿐만 아니라 유전자 발현 조절, 후성 유전체 편집, 단일 염기 편집 등이 가능하다. 카스9 단백질의 endonuclease 활성 도메인인 RuvC와 HNH의 중요 아미노산에 변이를 도입하면 DNA 절단 기능이 불활성화된 디카스9(catalytically dead Cas9,dCas9)이 생성된다. 디카스9은 sgRNA의 스페이서 서열에 상보적인 위치에 결합하지만 DNA를 절단하지는 않는다. 이 디카스9 단백질에 전사 활성화 도메인(transcriptional activation domain), 전사 억제 도메인(transcriptional repression domain), 히스톤 아세틸화 도메인(histone acetyltransferase, HAT)과 같은 기능 도메인(effector domain)을 결합시키면 유전체 서열의 변화 없이 기능 도메인의 작동에 따라 유전자의 발현을 조절할 수 있다. 또한 다양한 유전병은 시토신(cytosine)이 자연적으로 탈염기화(deamination) 되어 티민(thymine)으로 바뀌면서 발생하는데 이러한 질병을 치료하고자 디카스9에 아데닌 디아미네이즈(adenine deaminase)를 결합시킨 크리스퍼 염기교정 유전자가위(base editor)를 제작하여 A→T, G→C로 치환하는 연구도 보고되었다[2]. 크리스퍼 염기교정 유전자가위는 기존의 비상동 말단 연결에 의한 변이 유도에 비해 훨씬 정밀하게 목적 유전자를 교정할 수 있기 때문에 치료제로서 크게 각광받고 있다.
2.2 크리스퍼 유전자가위를 이용한 신호 저장 장치
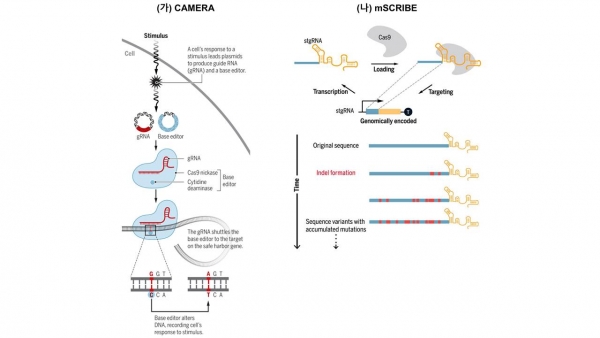
세포 분화, 노화, 암 발생 과정 동안 세포 상태의 변화를 기록하여 관찰하는 것은 생명현상에 대한 이해와 치료제 개발에 매우 중요하다. 크리스퍼-카스9의 유전체 편집 기능을 활용하면 세포가 받는 다양한 외부 혹은 내부 신호에 대한 기록을 DNA에 지속적으로 저장할 수 있다. 크리스퍼-카스9을 이용한 저장 방법으로 Tang 그룹은 대장균 및 동물 세포에서 2종류의 카메라(CRISPR-mediated analog multi-event recording apparatus, CAMERA) 시스템을 선보였다[3]. 카메라1 시스템은 대장균에서 작동하는 장치로 하나의 세포 내에 크리스퍼-카스9 시스템과 크리스퍼-카스9에 의해 절단되지 않는 플라스미드 R1, 절단되는 플라스미드 R2가 동시에 존재한다. 항생제나 영양소와 같은 외부 신호가 발생할 때 크리스퍼-카스9 시스템이 플라스미드 R2를 절단하여 플라스미드 R1과 R2의 비율을 변화시킨다. 이 비율 변화를 판독하면 여러 외부 신호의 세기 및 지속 시간을 측정할 수 있다. 카메라2 시스템은 크리스퍼 염기교정 유전자 가위를 이용한 저장 장치로 다양한 외부 신호에 대한 기록이 C.G에서 T.A로 치환된 DNA 서열로 기록된다(그림 1).
그림 1. 크리스퍼-카스9 시스템을 이용한 기록 장치. CAMERA 시스템: 외부 자극 시 크리스퍼 염기교정 유전자가위가 발현되어 세포염색체의 시토신을 티민으로 변이시킨다. 이 변이 서열을 분석하면 외부 자극의 유무를 판독할 수 있다[5]. mSCRIBE 시스템: 외부 자극에 의해 stgRNA와 카스9 단백질이 발현되면 카스9/stgRNA 복합체가 자신의 stgRNA를 절단한다. 이때 비상동 말단 연결에 의한 복구 기작에 의해 stgRNA 영역에 변이가 발생한다. 자극이 지속되면 상기 과정이 반복되어 변이가 계속 축적된다[4].
예를 들면 빛, 바이러스 감염 등과 같은 다양한 신호를 처리하였을 때 대조군 세포에 비해 신호를 받은 세포에서 DNA변이가 가속화되었다. 또한 인간의 질병과 연관되어 있고 배 발생(embryonic development) 과정에서 중요한 Wnt 신호를 감지하는 카메라2 시스템을 제작할 경우 Wnt 신호를 활성화시키는 물질을 처리하였을 때 세포의 게놈에 변이가 도입되어 신호 발생 유무를 판단할 수 있었다. 이렇듯 카메라 시스템은 신호에 대한 기록이 DNA에 저장되기 때문에 염기 서열 분석을 통해 변이의 빈도 판독이 가능하고 신호의 유무 및 지속 시간 등에 대한 정보를 알 수 있다. 한편, Perli 그룹은 크리스퍼-카스9 시스템에 의해 DNA가 절단 시 비상동 말단 연결에 의해 변이가 발생하는 현상을 이용한 기록 장치를 개발하였고 이를 mSCRIBE (mammalian synthetic cellular recorders integrating biological events)라 명명하였다(그림 1) [4]. 이 기록 장치는 카스9/sgRNA 복합체가 자신의 sgRNA를 절단(self-targeting guide RNA, stgRNA)하여 변이를 발생시키고 이 과정은 자극이 지속되는 동안 반복되어 stRNA의 스페이서에 변이가 축척된다. 따라서stgRNA 스페이서 부위의 서열 변이를 분석하면 세포가 받는 자극의 세기 및 지속 시간 등에 대한 정보를 얻을 수 있다. 일례로, 쥐에서 LPS (lipopolysaccharide)에 의한 염증 유도 시 mSCRIBE를 갖고 있는 기록 세포가 염증의 유무 및 염증 유도제의 처리 빈도를 성공적으로 기록하였다.
2.3 크리스퍼 유전자가위를 이용한 질병의 진단
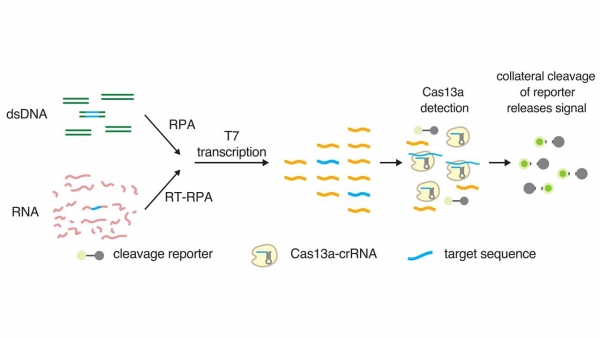
크리스퍼-카스9 시스템은 유전체 편집 뿐만 아니라 암, 바이러스 감염과 같은 질병을 조기에 진단하는 데에도 사용될 수 있다. Lee 그룹은 다양한 암을 조기에 진단하기 위하여 CUT-PCR (CRISPR-mediated, ultrasensitive detection of target DNA-PCR) 방법을 고안하였다[6]. 이 방법은 혈액에 존재하는 암세포로부터 유래한 미량의 순환종양 DNA (circulating tumor DNA, ctDNA)를 중합효소 연쇄 반응(polymerase chain reaction, PCR)으로 검출하는 방법으로 정상세포로부터 유래된 다량의 DNA가 크리스퍼-카스9에 의해 제거되기 때문에 순환 종양 DNA가 선택적으로 증폭된다. 이 방법을 대장암 환자의 혈액에 적용한 결과 암과 연관된 변이를 효과적으로 검출할 수 있었다. 최근에는 제6형 크리스퍼-카스13a/C2c2 시스템을 이용한 DNA/RNA 검출 방법인 셜록(specific high-sensitivity enzymatic reporter unlocking, SHERLOCK)이 등장하였다(그림 2) [7].
그림 2. 셜록 시스템. 극미량의 바이러스 혹은 암 유래의 DNA/RNA 샘플을 RPA/RT-RPA로 증폭한 후 T7 RNA 중합효소로 전사시킨다.
전사된 RNA에 카스13a가 결합할 수 있는 목적 RNA가 존재하면 카스13a가 활성화된다. 활성화된 카스13a는 주변의 리포터 RNA를 무작위로 절단하여 형광 신호를 나타낸다 [7].
셜록은 시료에 포함된 목적 DNA 혹은 RNA를 RT-RPA (reverse transcription-recombinase polymerase amplification) 및 T7 RNA 중합효소로 증폭하는 단계와 크리스퍼-카스13a/C2c2 시스템에 의해 증폭된 목적 RNA가 존재할 때 주변의 리포터 RNA (reporter RNA)가 절단되어 형광 신호를 방출하는 단계로 구성된다. 이는 제6형 크리스퍼-카스13a/C2c2 시스템이 제2형 크리스퍼-카스9 시스템과 다르게 DNA 대신에 RNA를 절단하고, 또한 목적 RNA를 절단한 후에 카스13a가 활성화되어 비특이적으로 주변의 리포터 RNA를 절단하는 특성이 존재하기 때문이다. 셜록은 아토몰 (10-18 M) 수준의 극미량으로 존재하는 바이러스를 검출할 수 있으며 단일 핵산염기 다형현상(single nucleotide polymorphism, SNP)을 구분할 수 있기때문에 다양한 전염병 및 암을 진단하는 장치로 활용될 것으로 전망되고 있다.
2.4 인간 마이크로바이옴 엔지니어링을 이용한 질병의 진단
인간 마이크로바이옴 엔지니어링을 통한 특정 질병의 진단 및 치료를 위해서는 우선 질병의 지표 물질을 감지하는 인공유전자회로가 장착된 미생물을 제작해야 한다. 일반적으로 야생형 미생물의 경우 지표 물질을 감지할 수 없다. 따라서 일반적으로 전사조절인자(transcriptional factors)를 이용하여 질병의 지표 물질을 감지하고, 이 때 발생한 신호에 의해 리포터 유전자의 발현을 유도하는 인공유전자회로를 이용해야 한다. 최근, Pamela Silver 연구팀은 메모리 디바이스(memory device)를 제작하여 쥐의 장내에서 공생하는 대장균에 도입하였다. 이 대장균은 쥐에서 장내 염증이 발생했을 경우 염증의 지표 물질인 사티온산염(tetrathionate)을 감지하고, 감지된 신호를 지속적으로 기억하여 장기간 장내 정착하면서 염증을 진단할 수 있었다[8, 9]. 이때 개발한 메모리 디바이스는 일시적인 외부 신호를 장기간의 세포 반응으로 전환하기 때문에 입력 신호가 사라져도 입력된 신호를 기억할 수 있게 한다. 이 메모리 디바이스는 두개의 유전자회로로 구성되어 있는데, 하나는 외부 환경의 신호에 반응하는 유도 회로(trigger element)이고 다른 하나는 입력된 신호를 저장하는 기억 회로(memory element)이다. 기억 회로는 외부 환경의 신호가 없을 경우 전사조절단백질인 cI의 발현을 통해 하위에 위치한 다른 전사조절단백질인 Cro 및 lacZ 리포터 유전자의 발현을 억제함으로써 “OFF” 상태를 유지한다. 이때, 장내 염증 지표 물질 중 하나인 사티온산염과 같은 외부의 특정 감지 물질이 감지되면 유도 회로에 의해 Cro 단백질이 발현되고, Cro 단백질은 “OFF” 상태를 유지하는 전사조절단백질 cI의 발현을 억제함으로써 Cro 단백질과 lacZ 리포터 유전자의 발현을 유도한다. 한번 유도된 Cro 단백질과 lacZ 리포터 유전자는 신호 물질이 없어도 지속 가능한 “ON” 상태를 유지한다(그림 3). 개발된 기술을 이용하여 제작한 장 정착성 대장균은 비침습성 리포터(noninvasive reporters)로써 장내 염증이 유도된 쥐에 적용한 결과 6개월 이상 지속적으로 염증을 진단할 수 있었다.
그림 3. 외부의 특정 신호를 인식 후 입력된 신호를 장기간 기억할 수 있는 기억 장치. 외부 환경의 특정 신호가 없을 경우 기억 회로(memory element) 상의 전사조절단백질 cI에 의해 하위의 다른 전사조절단백질 Cro 및 lacZ 리포터 유전자의 발현이 억제된다(Memory “OFF” 상태). 사티온산염(tetrathionate)과 같은 외부의 특정 신호에 의해 유도 회로(trigger element)의 Cro 단백질이 발현되면 “OFF” 상태를 유지하고 있던 cI 단백질의 발현을 억제함으로써 유도 회로의 Cro 단백질 및 lacZ 리포터 유전자의 발현이 유도된다(Memory “ON” 상태). 한번 유도된 Cro 단백질 및 lacZ 리포터 유전자의 발현은 지속적인 외부의 신호 없이도 지속가능한 “ON” 상태를 유지할 수 있다[8, 9].
2.5 마이크로바이옴 엔지니어링을 이용한 질병의 치료
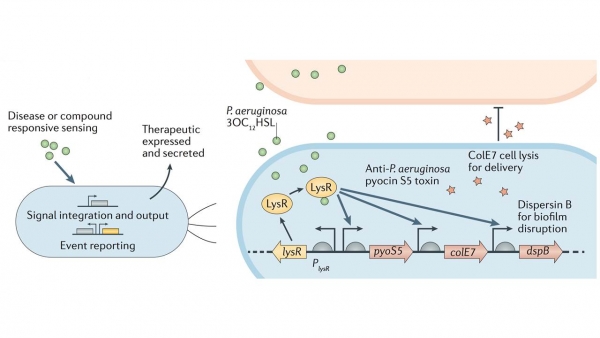
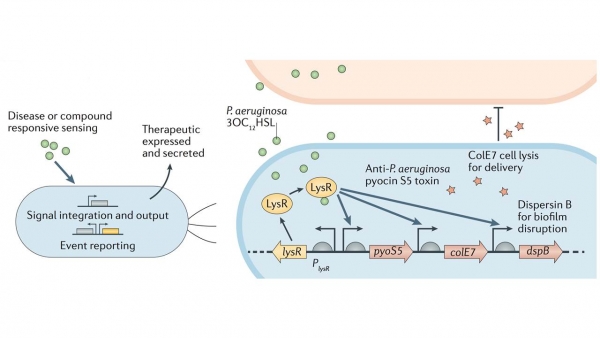
앞서 설명된 인공유전자회로를 이용한 질병의 진단 외에도 설계된 미생물이 질병의 지표 물질을 감지한 후, 이 신호에 의해 질병을 치료할 수 있는 물질을 생산한다면 질병의 진단과 치료가 동시에 가능해진다. 최근 Matthew Chang연구팀은 녹농균(Pseudomonas aeruginosa)에 의한 세균성 감염 치료를 위해 프로바이오틱스 대장균을 재설계하여 사용하였다[10]. 녹농균에 대한 특이적 감지 신호로 녹농균에서 유래한 정족수 감지 물질(quorum sensing molecule)인 N-acyl homoserine lactone(3OC12HSL)을 이용하였다. 정족수 감지는 미생물이 자기와 동종 혹은 다른 종의 군집밀도를 감지하는 기작으로, 미생물이 분비하는 특정한 신호 물질이 미생물의 밀도가 증가하여 일정 농도이상 축적되면, 축적된 신호 물질을 감지한 전사조절단백질에 의해 내부의 유전자 발현이 조절된다. 이 녹농균의 정족수 감지 기작을 이용하여 상기 연구팀은 프로바이오틱스인 대장균 니즐 1917(E. coli Nissle 1917)에 도입하여 녹농균(P.aeruginosa)으로부터 유래한 정족수 감지 신호(quorum signal)에 반응하는 치료용 대장균을 개발하였다. 이 대장균은 녹농균의 정족수 감지 신호 물질인 3OC12HSL을 인식하여 이에 대한 반응으로 자가 세포 용해 단백질인 ColE7을 생산한다. 동시에 항생물막(anti-biofilm) 단백질인 Dispersin B와 항균 단백질인 S5 pyocin을 생산하여 녹농균의 생물막을 파괴하고 궁극적으로는 녹농균을 사멸시켜 녹농균의 감염을 치료할 수 있게 된다(그림 4).
그림 4. 녹농균(Pseudomonas aeruginosa)에 의한 세균성 감염을 치료할 수 있는 치료용 대장균 니즐 1917(Escherichia coli Nissle 1917).
녹농균의 정족수 감지 물질(quorum sensing molecule)인 3OC12HSL을 특이적 감지 신호로 인식하여 자가 세포 용해 단백질ColE7, 항생물막 치료 단백질 Dispersin B, 항균 단백질 S5 pyocin을 동시 생산하여 감염 부위에 확산시켜 녹농균의 사멸을 유도한다[9, 10].
2.6 재설계된 미생물의 제어를 통한 생물안전(biosafety) 및 생물학적 봉쇄(biocontainment)
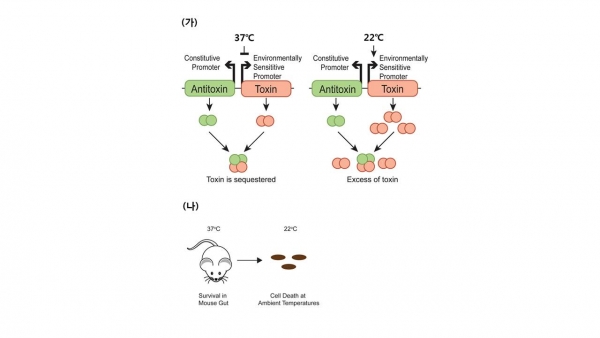
합성생물학 기술에 의해 재설계된 미생물을 질병의 진단 및 치료 분야에 적용하기 위해서는 생물안전(biosafety)과 생물학적 봉쇄(biocontainment)에 대한 기술 개발이 중요하다. 이에 관한 다수의 선행 연구가 주목받고 있는 가운데 최근 세포 사멸 스위치(kill switch) 시스템이 보고되었다[11]. 이 시스템은 미생물 주위의 온도 변화에 반응하는 프로모터(promoter)와 type Ⅱ toxin-antitoxin 시스템을 이용한다. 체내 온도인 37℃에선 미생물의 사멸을 유도하는 독성단백질(toxin protein)인 CcdB의 발현이 억제되어 미생물의 생존이 가능하지만 미생물이 체외로 배출되었을 경우 환경의 온도가 체내 온도보다 낮기 때문에(22℃ 이하) 낮은 온도에서 작동하는 프로모터에 의한 CcdB의 발현이 높아져서 세포 사멸이 유도되게 된다(그림 5). 이러한 시스템을 통해 진단 및 치료용 미생물이 체외로 배출되었을 경우, 사멸 스위치에 의해 미생물의 사멸이 유도되어, 재설계된 미생물이 자연의 미생물 군집으로 유입되거나 혹은 개인 간에 이동될 가능성을 줄여주는 생물학적 봉쇄가 가능하게 된다.
그림 5. 생물안전(biosafety) 및 생물학적 봉쇄(biocontainment)를 위한 세포 사멸 스위치(kill switch). (가) 미생물 주위의 온도 변화에 반응하는 프로모터(promoter) 및 type Ⅱ toxin-antitoxin 시스템을 이용한 세포 사멸 스위치 시스템. (나) 장내(37℃)에선 미생물의 생존이 가능하지만 체외(22℃)에선 독성 단백질(toxin protein)인 CcdB에 의해 미생물의 사멸이 유도된다[11].
참고 문헌
1. Koonin EV, Makarova KS, Zhang F: Diversity, classification and evolution of CRISPR-Cas systems. Current Opinion in Microbiology 2017, 37:67-78.
2. Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR: Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 2017, 551:464-471.
3. Tang W, Liu DR: Rewritable multi-event analog recording in bacterial and mammalian cells. Science 2018.
4. Perli SD, Cui CH, Lu TK: Continuous genetic recording with self-targeting CRISPR-Cas in human cells. Science 2016, 353:.
5. Cohen J: ‘CAMERA’ records cell action with new CRISPR tricks. Science 2018, 359:728.
6. Lee SH, Yu J, Hwang GH, Kim S, Kim HS, Ye S, Kim K, Park J, Park DY, Cho YK et al: CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene 2017, 36:6823-6829.
7. Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, Verdine V, Donghia N, Daringer NM, Freije CA et al: Nucleic acid detection with CRISPR-Cas13a/C2c2. Science 2017, 356:438-442.
8. Riglar, D. T. et al. Engineered bacteria can function in the mammalian gut long-term as live iagnostics of inflammation. Nature Biotechnology 2017, 35:653.
9. Riglar, D. T. et al. Engineering bacteria for diagnostic and therapeutic applications. Nature Reviews Microbiology 2018, 16:214-225.
10. Hwang, I. Y. et al. Engineered probiotic Escherichia coli can eliminate and prevent Pseudomonas aeruginosa gut infection in animal models. Nature Communications 2017, 8:15028.
11. Stirling, F. et al. Rational design of evolutionarily stable microbial kill switches. Molecular Cell 2017, 68:686-697.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update