| 합성생물학의 바이오메디컬 응용: 마이크로바이옴 엔지니어링 | |
|---|---|
| Date 2018-04-15 16:50:50 |
|

교수
서울대학교 화학생물공학부
swseo@snu.ac.kr
1. 서론
인간은 장내를 비롯한 다양한 기관계에 미생물과 공생관계를 이루며 그 상호작용에 기반한 생명 활동을 한다. 1인의 장 내에 분포하는 미생물은 마이크로바이옴(microbiome)이라 불리며, 인체의 한 기관계로 분류되기도 하는 이 장 내 세균은 장내에서 하나의 생태계(ecosystem)를 가진다. 또한 다방면의 측면에서 인체의 건강과 유효한 상관관계를 가지며, 그 수는 인체를 구성하는 사람 세포의 10배를 훨씬 웃도는 세포 수를 가지는 것으로 추정된다. 최근에는 장 내 환경뿐만 아니라 인간의 면역계 및 뇌와 같은 신경계에까지 영향을 미친다는 연구 결과들을 통해 인체에 미치는 높은 영향력이 입증되어 왔다. 더 나아가 복잡한 상호 진화 과정(co-evolution)을 거쳐 그상관관계는 지속적으로 확장되고 있다고 보고 있다[1]. 특히 현대인의 건강에 적신호인 당뇨, 비만 및 고혈압과 같은 대사증후군(metabolic syndrome)을 비롯하여 사망률 1위인 암과 같은 질환들이 장 내 건강 및 미생물 분포도와 상관관계를 가짐이 관찰됨에 따라, 장 내 미생물을 활용한 질병의 예방 및 치료에 대한 연구가 활발히 이루어지고 있다.
최근 급속도로 발전하는 합성생물학의 기술은 이러한 장내 야생형(wild type) 미생물의 내재적인 질병 예방 기능뿐만 아니라, 더 나아가 미생물의 공학적인 재조합을 통해 질병의 진단 및 치료까지 가능하게 한다는 점에서 바이오 메디컬 분야에 접목될 잠재성이 제시된다. 본 리포트는 다양한 합성생물학의 응용 분야 중 체내 바이오센서 기술에 주목하여 서술하고, 대표적인 사례들을 소개하고자 한다. 바이오 센서 기술은 전기회로에서 착안하여 외부의 환경을 인풋으로 주어진 자극에 적응 혹은 환경을 변화시키기 위한 아웃풋을 생성하는 유전자 회로(genetic circuit)의 구축을 기본 개념으로 갖는다. 이는 장내 환경을 감지하고 특정 질환의 자체적인 진단을 통해 증상 완화 혹은 치료를 수행하는 공학적 재조합 미생물을 만드는 데에 적합한 기술로 여겨진다.
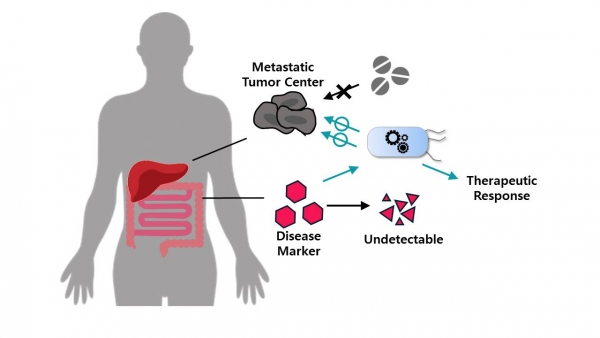
체내에서 직접 질병의 진단 및 치료를 수행하는 미생물 치료제는 기존의 비(非)생물 치료(non-living therapeutics)가 갖는 한계들을 보완한다는 점에서 의의를 갖는다. 혈관 혹은 상부 위장관(uppergastrointestinal tract)에서 빠르게 분해되는 약물의 전달(therapeutic delivery)의 경우에는 장내 미생물을 통해 on-site로 생산하는 것이 훨씬 더 효율적이다[2]. 또한 결장(colon)이나 종양의 중심부(centre of tumours)와 같이 기존의 경구 및 비경구 투여로 전달되기 어려운 취약 부위에 미생물을 통해 약물의 전달을 할 수 있다는 점에서 기존의 한계를 극복할 가능성이 있다. 이에 최근 암세포의 증식 세포 및 보조 세포로의 이분화 이론이 인정받으면서, 보조 세포에 둘러싸인 증식 세포를 제거하기 위해 혈중산소결핍(hypoxic) 상태의 종양 중심부에서 서식 가능한 대표적인 미생물 Salmonella entrica subsp. Enterica serovar Typhimurium(이하 S.Typhimurium)을 재조합하는 암 치료제가 개발 중에 있다[3]. 약물의 전달(therapeutic delivery) 뿐 아니라 질병 진단(disease diagnosis)에 있어서도 재조합 미생물 치료제가 갖는 장점이 있다. 장 내 환경은 다양한 효소 및 생명활동에 따라 매우 활발하게 대사 물질의 분해, 변형,흡수가 이루어진다. 이러한 역동적인(dynamic) 장내 환경에 의해 장에서 분리되기 전 빠르게 분해, 변형 및 흡수될 수 있는 질병 지표들을 장 내에서 유전자회로로 구축된 바이오 센서를 통해 직접 감지함으로써 더욱 정확하고 정밀한 질병 진단이 가능해진다. 즉, 기존의 비생물적 치료의 한계를 극복하고 역동적인 장 내 환경에서 신속하게 질병을 진단하고 직접적으로 치료를 수행하는 장내 재조합 미생물을 개발하는 것은 미래 의학에 있어 지속적으로 발전할 전망이다(그림 1).
그림 1. 재조합 미생물 치료제의 의의
2. 본론
2.1 장내 환경 감지 바이오 센서의 개발
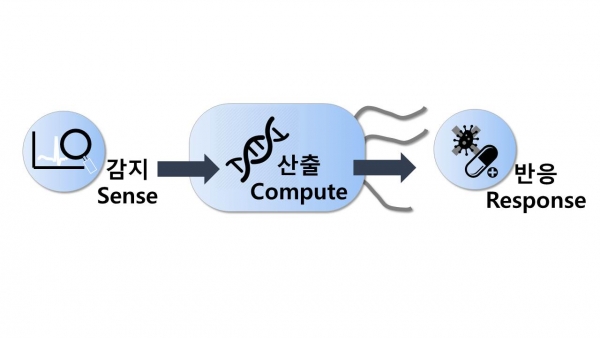
바이오 센서 적합의 재조합 미생물은 감지(sense), 산출(compute), 그리고 반응(respond)의 세 단계로 그 역할을 수행한다(그림 2).
이전부터 연구되어 온 재조합 미생물로부터의 제약 단백질 및 펩타이드 생산은 ‘반응(respond)’ 단계에 접목되며, 본조에서 다룰 바이오 센서 기능은 ‘감지(sense)’ 단계에서 응용된다.
미생물을 통해 구현하는 바이오 센서는 다양한 요소들(components)로 구성된다. 첫 번째 요소는 자극을 감지하고 결과를 도출하는 유전자회로이다. 기존의 생물체는 무수히 많은 유전자 발현 제어 시스템으로(expression regulatory system) 생명활동을 한다. 이러한 제어 시스템을 합성생물학 기술을 통해 자체적으로 응용 혹은 외래 유전자 회로로 구축하여 표적 미생물에 도입함으로써, 특정 유전자의 발현을 자극하는 선별된 인자를 사용할 수 있다. 미생물 감지의 기작은 일성분계(一成分系, one-component systems 이하 OCSs)와 이성분계(二成分系, two-component systems 이하 TCSs)의 두 종류로 설명 가능하다. OCSs는 자극 인자가 직접적으로 표적 유전자의 발현을 촉진 혹은 억제하는 것으로, 대표적으로 lac 프로모터가 있다. TCSs는 자극 인자가 특정 단백질의 발현을 촉진하거나 인산화(phosphorylation)시킴으로써 그 단백질이 표적 유전자의 발현을 조절하는 두 단계의 제어 시스템이라는 점에서 차이를 갖는다. OCSs는 TCSs에 비하여 더 단순하고 조작하기 쉽다는 장점이 있으나, 미생물의 세포막(membrane)을 통과하지 못하는 화학적 질병 지표는 감지하기 어렵다는 점에서 제한이 있다. 그리고 TCSs가 단백질의 인산화 단계를 거치기 때문에 유전자 발현량의 미세 변이에 대항하기에 OCSs에 비하여 상대적으로 견고한 시스템으로 알려져 있다[4]. 세부적으로는 메모리 회로(memory circuit), 논리 게이트(logic gate), 쿼럼 센싱(quorum sensing) 그리고 킬 스위치(kill switch)와 같이 기존의 생물체가 가지고 있는 다양한 유전자 발현 제어 시스템을 장 내 미생물에 도입한 연구들이 이루어졌다.
바이오 센서 재조합 미생물의 구축에 있어 고려해야 할 또 다른 주요 요인은 숙주 미생물 선별(chassis selection)이다. 합성생물학의 기술이 적용되기 위해서는 기본적으로 ‘유전적으로 다루기 쉬운(genetically tractable)’ 미생물이어야 한다. 바이오 센서라는 복잡한 유전 체계를 도입하기 위해서는 프로모터, RBS (Ribosome Binding Sites) 등의 라이브러리를 통해 발현량을 수치적으로 제어 가능한 데이터 구축이 필수적이다. Voigt 교수 연구진은 이러한 필요성을 반영하여 기존에 많이 개발된 E. coli 뿐만 아니라, 장내 미생물로 알려진 B. thetaiotaomicron 에 대하여 20배의 발현량 차이를 보이는 프로모터 라이브러리를 설계하였다[5]. 이러한 합성생물학 데이터를 기반으로, 본 전략은 체내 공생미생물의 거처(niche)가 어디냐에 따라서 표적으로 하는 질환과 효과를 달리한다. 예를 들어, 궤양성 대장염(ulcerative colitis)의 경우 결장(colon)에 장시간 서식하는 미생물을 선별하고, 크론병(Crohn’s disease)의 경우 소장 및 대장(intestine)에서 장시간 서식하는 미생물을 선별하는 것이 타당하다. 더 나아가 거처에 따른 일차적 선발 후, 숙주 미생물이 해당 거처에서 얼마나 군집(colony)을 용이하게 형성하는지가 자극을 감지하는 감도에 영향을 준다. 이는 세부적인 질병 치료 전략을 결정 짓는 요인이 된다. 장내 미생물인 Lactobacillus lactis와 Escherichia coli Nissle 1917(EcN1917)은 자연적으로 군집화(colonization)를 잘 이루지 않는 특징을 보이므로, 기존 장 내 미생물 분포에 영향을 주지않고 체내 부담을 최소화하기 위한 전략에 응용될 수 있다[6]. 위와 같은 요인들을 조직적으로 구성하여 의학과 합성생물학을 융합한 바이오 의학 연구들이 최근 급속도로 개발되었다. 이제 그 대표적인 사례들에 대해 소개하고자 한다.
2.2 Escherichia coli
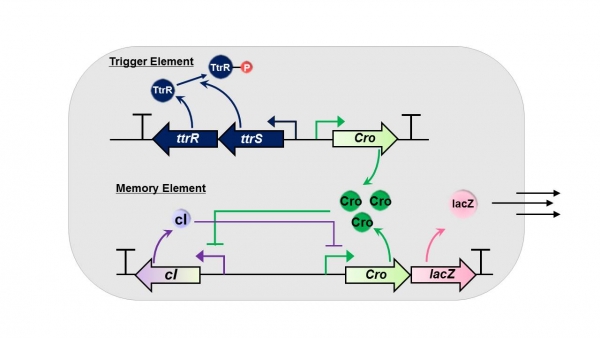
하버드 의과 대학 연구진은 박테리오파지의 cI-Cro 스위치를 합성생물학 기술 기반으로 대장균에 도입하여, lacZ 를 표지 유전자로 하는 기억 회로(memory circuit)를 구축하고, 체내 염증 반응의 산물인 tetrathionate를 감지하도록 하였다(그림 3) [7].
그림 3. E. coli cI-Cro bistable trigger switch 기반 유도인자 및 기억인자 개발 연구
대표적인 TCSs인 cI-Cro 스위치는 박테리오 파지가 용원환(溶原環, lysogenic cycle)에서 용균 회로(溶菌回路, lytic cycle)로 유전자 발현 주기를 변화시키는 것을 제어하는 시스템으로 외부 자극을 인풋으로 감지하여 장기적인 아웃풋으로 유지시킨다는 특징을 갖는다. cI 단백질은 Cro의 발현을 억제하고, Cro 단백질은 cI의 발현을 억제한다. 즉, 기존에는 안정적인 cI 발현 시스템으로 유지되다가 특정 자극에 의해 한계점 이상으로 Cro 단백질이 생성되면 cI의 발현이 억제되는 반대의 안정적인 시스템으로 전환이 일어나는 것이다[8]. 이 자연적 유전자 발현 제어 스위치를 합성생물학적 기억 회로로 구축하기 위해서는 유도 인자(trigger element)와 기억 인자(memory element) 가 필요하다. 유도 인자로는 tetrathionate에 의해 성장을 촉진시키는 S. Typhimurium 의 PttrBCA 프로모터를 사용하였다. Tetrathionate의 인산화(phosphorylation)에 의해 양성 피드백을 받는 PttrBCA 하에 Cro 단백질 유전자를 설계하였다. 그리고 기억 인자로는 cI-Cro 스위치를 설계하고 Cro 프로모터 하에 Cro 단백질 대신 표지단백질인 lacZ를 도입하였다. Cro 시스템의 유지는 유도 인자에서 생성되는 Cro 단백질로도 충분히 신호를 유지할 수 있기 때문이다. In vivo 실험을 통해, 선별 표지용 항생제를 재조합 미생물의 초기 투여 단계에서 일시적으로 투여하고 미생물의 군집화(colonization) 정도를 살펴본 결과, 약 200일 이상까지 재조합 미생물이 항생제 없이 장 내에서 생존함이 관찰되었다.
즉, 장 내에서 장기간 서식하며 일시적인 질병 지표를 인지하며, 그 신호를 장기적으로 유지하여 시간적인 유예를 갖는 유전자 발현 제어 전략이 성공하였다. 또한 항생제 선별 표지 없이 쥐 동물실험에서 장시간 경쟁적 성장을 보인 재조합 미생물의 가능성을 관찰한 첫 연구라는 점에서 본 연구는 의의를 갖는다.
장 내 환경을 감지하는 진단 뿐 아니라, 간암의 전이 정도를 파악하는 재조합 대장균 진단제에 대한 연구도 이루어졌다. MIT의 Bhatia 교수 연구진은 암세포에 군집화(colonization)를 이루면서 인체 무해성 질병 지표를 생산하는 재조합 대장균 EcN 1917을 개발하였다[9]. 산소 결핍의 장 내에서 활발하게 활동하는 장 내 미생물은 그 특성에 따라 종양의 환경에서도 군집하는 경향을 가진다고 알려져 있다[10]. 이에, 혈관에서 생존하기 위해 고농도로 투입되어야 했던 기존의 세포 치료제의 문제점을 해결하기 위해 저농도의 무해성 미생물을 구강으로 섭취하여 간정맥을 통해 장에서 바로 간으로 도달하는 전략을 시험하였다. 그 결과, 다양한 미생물 선별 표지를 IVIS 이미징 기술을 통하여 관찰함으로써 간암의 전이 정도와 미생물의 군집 정도가 비례함을 보였다. 더 나아가 재조합 미생물이 생산하는 lacZ enzyme의 발현 정도에 따라 소변의 색이 유의미하게 변하는 것을 통해, 미생물을 구강 섭취하여 소변의 색으로 암을 진단하는 손쉬운 방법의 개발 가능성을 보였다.
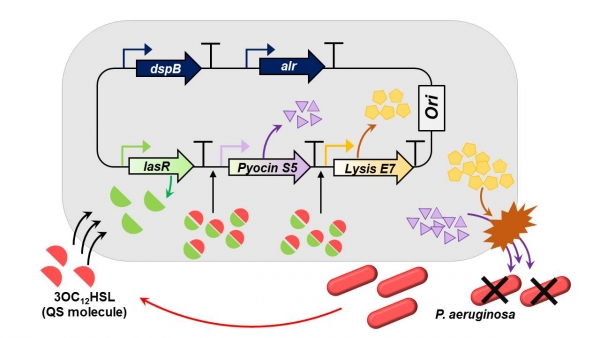
싱가포르 국립대학교의 장욱 교수 연구진은 병원균의 자연적인 쿼럼 센싱(Quorum sensing)을 EcN의 쿼럼 센싱의 매개로 활용하여 ‘Sensor-Kill’ 바이오 센서 회로로 구축하였다(그림 4) [11].
그림 4. P. aeruginosa QS의 E. coli Nissle 1917에 도입을 통한 병원성 균의 성장 억제 연구
병원성 균이 자체적으로 생산하여 내보내는 3OC12HSL이 세포막을 통과하면, 기저 발현되는 lasR 단백질과 결합하여 luxR 프로모터의 작동을 유도한다. 두 개의 luxR 프로모터의 하위에는 각각 Pyocin S5와 Lysis E7 합성 유전자가 있는데, 그 역할은 다음과 같다. S5 단백질은 P. aeruginosa 병원성 균의 생존을 억제하는 역할을 하며, E7단백질은 장내 미생물의 세포막을 파괴하여 S5 단백질이 병원성 균에 효과적으로 도달할 수 있도록 보조하는 역할을 한다[12]. 연구진은 이러한 기작을 도입하여, 병원성 균에 감염된 꼬마선충에서 두 유전자를 회로에 넣었을 경우에만 유효한 감염 저하 기능을 확인하였다. 본 연구를 통해 장내 미생물이 쿼럼 센싱의 매개가 되어, 병원균의 자체적인 기작을 활용하여 세포 용해를 촉진, 병원성 감염의 억제능을 선보였다.
2.3 Salmonella Typhimurium
앞서 언급했던 살모넬라 균 역시 암의 치료를 위한 공학적 재조합 기술을 거치는 또 하나의 대표적인 균주이다. 기존의 화학적 항암 치료 요법의 한계는 바로 자체적인 독성으로 인해 암세포가 아닌 건강 세포에도 악영향을 줄 수 있다는 것이다. 살모넬라 균주는 다른 일반적인 기관에 비하여, 암 조직에서 약 10,000 배 높은 군집률을 보인다는 특징을 보이고 있다[13]. 또한 산소 결핍 환경인 암 조직 내부를 자유롭게 통과할 수 있는 균이라는 점에서 in situ 의 항암 물질을 생산하기에 적합한 치료 균으로 여겨진다. 더 나아가, 합성생물학적으로 재조합된 살모넬라의 선별적 성장은 암세포에 대하여 특이적으로 작동하면서 정상 세포에의 악영향을 최소화할 수 있기 때문에, 기존의 항암 치료 기법을 넘어서는 혁신적인 암 치료법으로 대두되고 있다.
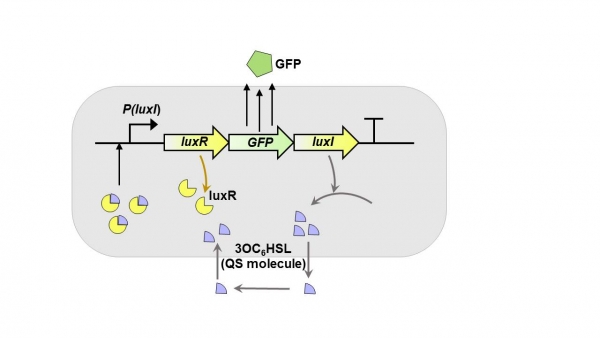
미국의 Forbes 교수 연구진은 살모넬라 균의 자체적인 쿼럼 센싱 기작을 이용하여, 암세포에서 높은 군집률을 보이는 살모넬라 균의 내재적 생명 활동을 활용하였다(그림 5) [14].
그림 5. S. typhimurium의 자체 QS 기반 군집도에 따른 reporter 단백질의 발현 연구
QS 스위치라고 불리는 기작은 다음과 같다. 살모넬라 단일 균이 근접 거리에서 동일 종의 균이 분비하는 3OC6HSL 단백질을 감지하면서 이를 특정 유전자 발현의 유도 지표로 사용한다. LuxR 프로모터가 3OC6HSL 단백질로 인하여 유도되며, 하위 오페론의 단백질을 발현시킨다. 연구진은 LuxR 프로모터의 하위에 GFP 단백질을 도입하여 lux QS 시스템이 암 조직과 정상 조직에서 유의미한 발현 차이를 보이는지 관찰하였다. 또한, tumor-on-a-chip 시스템을 사용하여, 인지 가능한 GFP 발현을 촉진하기 위해 필요한 암세포의 밀도를 측정하였다. 연구에 사용된 비(非)병원성 살모넬라는 약 155mm 이하의 근접 거리에서 동종 균의 3OC6HSL 단백질을 인지하여 유도체로 사용함이 관찰되었다. 또한, 유효 GFP 발현을 위해서는 최저 108 cfu/mL의 세포 밀도를 가져야 함이 관찰되었다. 더 나아가, GFP 발현 정도가 암 조직과 정상 간 조직과 유의미한 차이를 보였다. 이를 통해, 의도하지 않은(off-target) 조직에서의 유전자 발현 유도를 최소화 할 수 있는 시스템을 개발한 연구라고 할 수 있다.
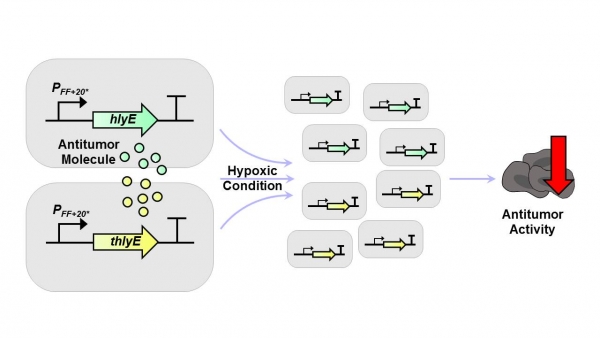
2009년 영국에서는 비 산소(hypoxic) 환경에서 성장이 촉진되는 살모넬라의 특징을 활용하여, 비 산소 환경에 의해 발현이 제어되는 시스템을 개발하였다(그림 6) [15].
그림 6. S. typhimurium의 자체 Hypoxic inducible system 기반 종양 중심의 산소 결핍 환경에서 항암 물질의 발현 촉진 연구
연구진은 비산소 유도성 프로모터(hypoxia-inducible promoter) FF20을 더 보완한 FF+20* 프로모터를 사용하여 암 조직에서 lac Z의 발현을 통해, 암 조직 특이적인 항암물질 생산 가능성을 보였다. 위의 연구와 마찬가지로 살모넬라가 종양과 정상 간 조직에서 약 10000:1의 비율로 서식한다는 선행 연구 결과가 있었으나[16], 정상 조직에서 살모넬라의 항암 작용 가능성을 배제할 수 없었다는 것이 기존의 견해였다. 본 연구진은 산소 결핍 환경에만 특정 유전자의 발현이 촉진되도록 하는 유전자회로를 설계하였고, 약한 -10 서열 프로모터를 갖는 기존의 자연적 pepT 프로모터를 합성생물학 기반의 site-directed mutagenesis 기술을 통해 보완하여무산소 환경에서 생성되는 FNR(Fumarate and Nitrate Reduction regulator) 단백질에 대한 의존성이 더 강해졌음을 관찰하였다. 또한 기존의 화학적 항암 요법이 암 조직의 비 산소성 부위를 더욱 축적시키는 기작임을 고려하여, 기존의 항암 요법과 병행 시에 더 높은 효과를 얻을 수 있음을 제시하였다.
2.4 그 외 기타
그 외에도 기존에 유익 물질 생산에 가장 보편적으로 사용되는 Lactococcus lactis 는 바이오 의학의 질병 치료 균주로 구축하기 위한 공학적 재조합 시도들이 진행되어 왔다. 2011년 펜실베니아에서 당뇨 환자의 장 내에서 nisin을 감지하여 생물학적으로 활성화 상태의 인슐린 유사체(analog)를 생산하는 L. lactis NZ9000이 개발, 연구되었다[17]. L.lactis 는 다세대를 거쳐 유제품 산업에 활용도가 높은 균으로, 위의 강한 산성에도 저항성을 갖는다는 것이 밝혀지면서[18], 구강 섭취 가능한 점막 전달 매개체(mucosal stomach delivery)로 주목 받고 있다. 백신을 비롯하여 다양한 체내 유익 물질을 생산하는 재조합 L. lactis가 개발되어 왔으며, 최신 바이오 센서 기술들을 접목하여 구강 세포 치료제로서의 발전 가능성이 높은 균이다.
장 내에서 가장 많은 군집 종으로 알려진 Bacteroides 속에 해당하는 B. thetaiotaomicron 도 마찬가지로 세포 치료 균주로서 최근 연구가 활발히 이루어지기 시작하였다. MIT의 Voigt 및 Lu 교수 연구진은 B. thetaiotaomicron 을 합성생물학적 플랫폼 균주로 활용하기 위해 프로모터, RBS과 같은 회로의 구성을 발현량을 기준으로 선별하는 라이브러리를 구축하였다[5]. 또한, 쥐의 장 내에서 유도성 CRISPRi (inducible CRISPR interference)와 재조합효소(recombinase) 시스템을 활용하여 B. thetaiotaomicron의 유도성 유전자 발현의 가능을 파악하였다. 연구진은 회로구성 요소들의 최적화 과정을 거치면서 장내 미생물인 B. thetaiotaomicron 이 디지털 논리 게이트, 기억 회로와 같은 더 복잡한 바이오 센서 기작을 소화할 수 있을 것이라는 전망을 보였다.
3. 결론
복잡한 생명 활동의 집합체인 인체 내에서 질환을 즉각적으로 감지하고 이를 치료하기 위해서 기존의 in vitro 진단방법으로는 한계가 있다. 장 내에서 공생관계를 이루며 인체와 무수히 많은 상호관계를 구축하는 장내 미생물에 합성생물학 분야의 유전자 재조합 기술을 접목한다면, 질환을 in vivo로 감지하여 더 나아가 치료까지 즉각적으로 수행하면서 기존 의학 기술의 한계를 보완하는 미생물 치료제의 개발이 가능하다. 다양한 사례들을 통해 현재 미생물 치료제에 대한 활발한 연구 및 개발이 외부 자극을 감지하는 바이오 센서 회로의 구축에 기반하고 있으며, 급속도로 발전하고 있음을 살펴보았다. 하지만 GMO(Genetically Modified Organism)의 개발이라는 점에서 이에 따르는 여러 사회적인 장벽을 넘기 위한 관련 법규의 제정과 논의가 필요하다. 현대인의 대사 질환과 암을 비롯한 질환 치료에 있어서 미생물 치료제가 사회에 기여하는 바가 더욱 커지기를 기대하는 바이다.
참고문헌
1. Ley, R. E., Lozupone, C. A., Hamady, M., Knight, R., & Gordon, J. I. (2008). Worlds within worlds: evolution of the vertebrate gut microbiota. Nature Reviews Microbiology, 6(10), 776.
2. Landry, B. P., & Tabor, J. J. (2017). Engineering Diagnostic and Therapeutic Gut Bacteria. Microbiology spectrum, 5(5).
3. Danino, T., Lo, J., Prindle, A., Hasty, J., & Bhatia, S. N. (2012). In vivo gene expression dynamics of tumor-targeted bacteria. ACS synthetic biology, 1(10), 465-470.
4. Shinar, G., Milo, R., Martinez, M. R., & Alon, U. (2007). Input.output robustness in simple bacterial signaling systems. Proceedings of the National Academy of Sciences, 104(50), 19931-19935.
5. Mimee, M., Tucker, A. C., Voigt, C. A., & Lu, T. K. (2015). Programming a human commensal bacterium, Bacteroides thetaiotaomicron, to sense and respond to stimuli in the murine gut microbiota. Cell systems, 1(1), 62-71.
6. Schultz, M., Watzl, S., Oelschlaeger, T. A., Rath, H. C., Gottl, C., Lehn, N., ... & Linde, H. J. (2005). Green fluorescent protein for detection of the probiotic microorganism Escherichia coli strain Nissle 1917 (EcN) in vivo. Journal of microbiological methods, 61(3),389-398.
7. Riglar, D. T., Giessen, T. W., Baym, M., Kerns, S. J., Niederhuber, M. J., Bronson, R. T., ... & Silver, P. A. (2017). Engineered bacteria can function in the mammalian gut long-term as live diagnostics of inflammation. Nature biotechnology, 35(7), 653.
8. Svenningsen, S. L., Costantino, N., & Adhya, S. (2005). On the role of Cro in λ prophage induction. Proceedings of the National Academy of Sciences of the United States of America, 102(12), 4465-4469.
9. Danino, T., Prindle, A., Kwong, G. A., Skalak, M., Li, H., Allen, K., ... & Bhatia, S. N. (2015). Programmable probiotics for detection of cancer in urine. Science translational medicine, 7(289), 289ra84-289ra84.
10. Toso, J. F., Gill, V. J., Hwu, P., Marincola, F. M., Restifo, N. P., Schwartzentruber, D. J., ... & Freezer, L. J. (2002). Phase I study of the intravenous administration of attenuated Salmonella typhimurium to patients with metastatic melanoma. Journal of clinicaloncology, 20(1), 142-152.
11. Hwang, I. Y., Koh, E., Wong, A., March, J. C., Bentley, W. E., Lee, Y. S., & Chang, M. W. (2017). Engineered probiotic Escherichia coli can eliminate and prevent Pseudomonas aeruginosa gut infection in animal models. Nature communications, 8, 15028.
12. Saeidi, N., Wong, C. K., Lo, T. M., Nguyen, H. X., Ling, H., Leong, S. S. J., ... & Chang, M. W. (2011). Engineering microbes to sense and eradicate Pseudomonas aeruginosa, a human pathogen. Molecular systems biology, 7(1), 521.
13. Forbes, N. S., Munn, L. L., Fukumura, D., & Jain, R. K. (2003). Sparse initial entrapment of systemically injected Salmonella typhimurium leads to heterogeneous accumulation within tumors. Cancer research, 63(17), 5188-5193.
14. Swofford, C. A., Van Dessel, N., & Forbes, N. S. (2015). Quorum-sensing Salmonella selectively trigger protein expression within tumors. Proceedings of the National Academy of Sciences, 112(11), 3457-3462.
15. Ryan, R. M., Green, J., Williams, P. J., Tazzyman, S., Hunt, S., Harmey, J. H., ... & Lewis, C. E. (2009). Bacterial delivery of a novel cytolysin to hypoxic areas of solid tumors. Gene therapy, 16(3), 329.
16. Ryan, R. M., Green, J., & Lewis, C. E. (2006). Use of bacteria in anti-cancer therapies. Bioessays, 28(1), 84-94.
17. Ng, D. T., & Sarkar, C. A. (2011). Nisin-inducible secretion of a biologically active single-chain insulin analog by Lactococcus lactis NZ9000. Biotechnology and bioengineering, 108(8), 1987-1996.
18. Klijn, N., Weerkamp, A. H., & De Vos, W. M. (1995). Genetic marking of Lactococcus lactis shows its survival in the human gastrointestinal tract. Applied and environmental microbiology, 61(7), 2771-2774.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update