| 미생물로부터 지놈마이닝에 의한 신규이차대사산물 발굴 | |
|---|---|
| Date 2018-10-06 16:10:59 |
|

교수
건국대학교 의생명공학과
hkang@konkuk.ac.kr
1. 서론
현재 임상에서 사용되고 있는 다수의 항생제 및 항암제들은 미생물의 이차대사산물로부터 유래되어왔다(그림 1). 따라서 미생물의 이차대사산물은 전통적으로 신약개발에 있어서 중요한 자원으로 이용되어 왔다[1,2]. 미생물로부터 신약후보물질을 찾아내는 전통적인 방법은 균주를 실험실 내에서 배양하여 이로부터 유기용매 추출에 의한 추출물을 만들고 이들의 활성을 평가하여 추출물 내에 활성물질의 존재 유무를 판단하는 것이다. 추출물 내에 활성물의 존재가 확인되면 이로부터 다양한 크로마토그래피 방법을 이용하여 활성을 나타내는 순수한 물질을 분리하고 이들의 화학적 구조 및 생물학적 활성을 조사하여 신약 후보물질로서의 가능성을 판단하게 된다.
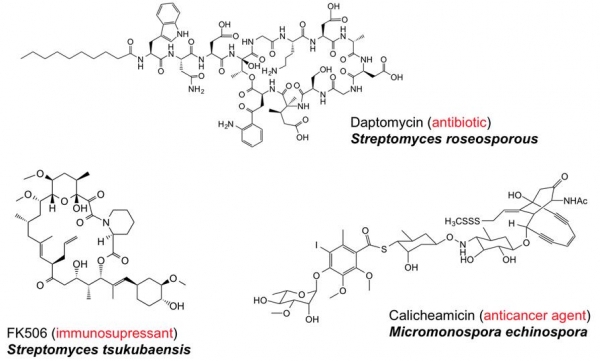
그림 1. 임상에서 사용되고 있는 미생물의 이차대사산물로부터 유래한 대표적인 항생제, 면역억제제와 항암제의 예.
지난 한 세기 동안 이러한 전통적인 방법에 의하여 많은 신약 후보물질들이 미생물로부터 발굴되어 왔지만 많은 반복적인 활성 스크리닝 과정 그리고 신규 미생물 균주의 발굴이 점차 어려워지면서 미생물로부터 신규화합물을 발굴하는 빈도 또한 급격히 줄어들고 있다. 이로 인하여 많은 다국적 제약회사에서는 미생물유래 이차대사산물을 회사의 신약개발 파이프라인 과정에서 철수시켜 왔다[3].
차세대 시퀀싱 기술의 발달은 하나의 미생물의 지놈 전체를 시퀀싱하기 위해 필요한 시간 및 비용을 획기적으로 줄였으며 따라서 현재까지 많은 수의 미생물에 대한 지놈 시퀀싱이 이루어졌다. 바이오인포메틱스 기술을 이용하여 미생물의 지놈시퀀스를 분석한 결과 의외로 하나의 미생물 지놈 안에는 많은 수의 이차대사산물의 생합성과 관련된 유전자 클러스터가 발견되었으며 이들의 대부분은 미생물에는 실험실 환경에서 발현되지 않는다는 사실을 확인하였다[4].
따라서 실험실 환경에서 발현되지 않아 기존의 전통적인 방법으로는 발굴이 어려웠던 미발현 생합성 유전자 클러스터들로부터 대사산물의 생산을 유도하고 이를 신약개발에 응용하려는 연구가 활발히 진행되고 있다. 이에 본 글에서는 지놈마이닝 방법에 의해 미생물로부터 신규이차대사산물을 발굴하는 내용에 대한 최근 연구동향을 소개하고자 한다.
2. 메타지노믹스 방법에 의한 미배양 미생물로부터 신규이차대사산물 발굴
기존의 연구에 의하여 자연환경에 서식하는 대다수 미생물들은 실험실 배양조건에서 증식하지 않는다는 사실이 확인되었으며 이러한 미배양 미생물들은 신규 이차대사산물 발굴에 있어서 중요한 자원으로 각광받고 있다. 토양이나 환경에서 수집한 시료로부터 순수한 미생물을 분리하여 배양하지 않고 바로 DNA만을 추출한 것을 eDNA 또는 메타지놈이라고 하며 이를 벡터에 클로닝 하여 라이브러리를 만들면 이로부터 유용한 유전자를 발굴하는 것이 가능하다.
하나의 이차대사산물 생합성에 관련된 유전자는 미생물의 지놈 안에 클러스터링 되어 있으며 이러한 생합성 유전자클러스터는 사이즈가 작게는 수십 kb에서 크게는 100 kb이상으로 매우 크다. 따라서 이러한 생합성 유전자클러스터를 탐색하고 클로닝하기 위해서 약 40 kb의 eDNA 조각을 높은 효율로 클로닝할 수 있는 cosmid 벡터를 이용하여 라이브러리를 제작하게 된다[5]. 이렇게 제작된 라이브러리는 이차대사산물 생합성 유전자를 선택적으로 증폭할 수 있는 degenerate 프라이머를 이용하여 스크리닝하고 이를 시퀀싱하여 증폭된 amplicon 시퀀스와 기존에 알려진 동일 생합성 유전자 시퀀스를 가지고 phylogenetic tree를 만든다(그림 2). 이 tree는 라이브러리로부터 신규 생합성 유전자 클러스터를 탐색하기 위한 가이드로 사용된다[6]. Phylogeny 분석을 통하여 amplicon시퀀스가 신규 생합성 유전자 클러스터로부터 유래되었다고 판단되면 이를 amplicon시퀀스에 결합하는 프라이머를 제작하여 라이브러리로부터 표적분리한다.
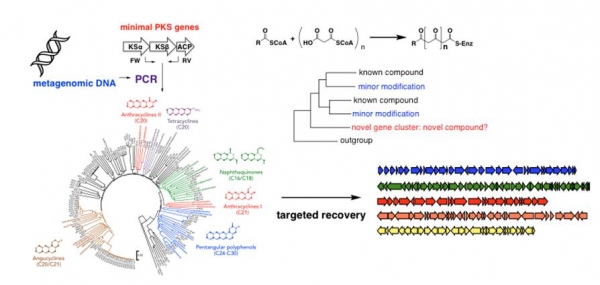
그림 2. Phylogenetic tree 분석을 이용하여 메타지놈으로부터 이차대사산물 생합성에 관련된 유전자클러스터를 탐색하는 방법
일반적으로 생합성 유전자클러스터는 사이즈가 40kb 이상이므로 2개에서 3개의 서로 겹치는 코스미드에 걸쳐 분포하며 따라서 표적 분리된 코스미드를 효모에서 상동재조합을 이용하여 하나의 DNA로 접합하여 최종적으로 BAC 벡터에 클로닝하게 된다[7]. 이렇게 메타지놈에서 클로닝 된 생합성 유전자 클러스터는 실험실 배양조건에서 잘 증식하는 이종호스트의 지놈에 삽입하여 발현을 유도하고 HPLC를 통한 메타볼롬 분석을 통하여 이로부터 생산되는 대사산물을 표적 분리한다.
현재까지 메타지노믹스 방법은 구조적으로 신규성이 뛰어난 이차대사산물 보다는 그림 3에서 보는바와 같이 기존에 알려진 임상적으로 효능이 검증된 이차대사산물의 유사체를 탐색하는 데에 주로 활용되어 왔다[8,9]. 그 이유는 메타지놈은 그 규모가 방대하기 때문에 메타지놈 전체를 시퀀싱하는 것이 불가능하기 때문에 생합성 유전자 클러스터 전체를 분석하기 보다는 증폭된 특정 생합성 유전자의 amplicon시퀀스만을 이용하여 클로닝 하려는 생합성 유전자 클러스터를 탐색하기 때문에 아직까지는 구조적으로 신규성이 우수한 이차대사산물의 발굴에 적용하는 데 있어서는 한계점을 드러내왔다.
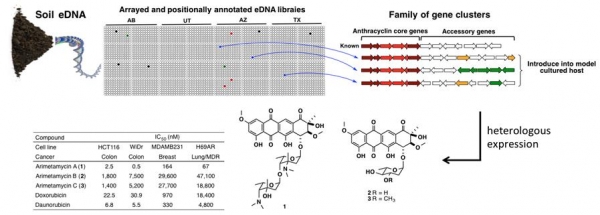
그림 3. 사막의 토양에서 분리된 메타지놈으로부터 임상에서 항암제로 사용되는 doxorubicin과 구조적으로 유사한 신규 anthracycline 계열의 화합물을 발굴한 예.
3. 지노믹스 방법에 의한 배양 미생물로부터 신규이차대사산물 발굴
실험실 조건에서 배양이 가능한 미생물의 경우에도 이차대사산물의 생합성에 관련된 유전자 대다수는 실험실 배양 조건에서는 발현되지 않는다. 따라서 아직도 미생물의 지놈 안에는 많은 수의 대사산물이 확인되지 않은 미발현 생합성 유전자 클러스터가 존재하며 이들은 신규 이차대사산물을 생산할 수 있는 잠재성을 가지고 있다. 차세대 시퀀싱 기술의 발달로 인하여 미생물의 지놈 전체를 빠르고 적은 비용으로 시퀀싱 하는 것이 가능하다. 지노믹스 기반 신규 이차대사산물 발굴연구에서는 먼저 미생물의 지놈을 시퀀싱 한 후 이를 바이오인포메틱스 기법을 이용하여 이차대사산물 생합성에 관여하는 유전자 클러스터를 탐색하고 클러스터내 존재하는 생합성 유전자를 분석하여 이의 대사산물의 신규성을 다각적으로 판단한다[10]. 이를 바탕으로 신규 이차대사산물을 생산할 것으로 판단되는 생합성 유전자 클러스터가 존재하면 이를 클로닝 한다. 클로닝과정은 위에서 언급한 메타지놈으로부터 이차대사산물 생합성 유전자 클러스터를 클로닝하는 방법과 유사하다(그림 4). 또한 최근에는 cosmid 라이브러리를 구축하지 않고 효모의 강력한 상동재조합 기능을 이용하여 genomic DNA로부터 생합성 유전자 클러스터를 바로 BAC 벡터에 클로닝하려는 시도가 이루어져 왔다[11]. 이 방법이 확립되기까지는 기술적으로 극복해야할 몇 가지 요소가 있지만 이 기술은 향후 생합성 유전자클러스터의 클로닝과정을 획기적으로 단축시킬 수 있을 것으로 생각된다.
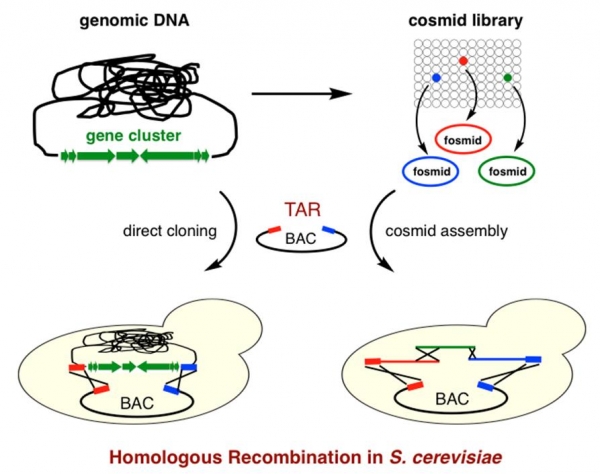
그림 4. 미생물의 지놈으로부터 이차대사산물 생합성에 관여하는 유전자클러스터를 클로닝하는 과정
이렇게 클로닝 된 생합성 유전자 클러스터는 이차대사산물의 생산이 최적화되었으며 또한 실험실 배양조건에서 안정적으로 증식하는 이종호스트의 지놈에 삽입하여 대사산물의 생산을 유도한다[12]. 동종호스트에서는 엄격한 전사조절시스템에 의하여 전사가 조절되어 실험실 배양조건에서는 미발현되던 생합성 유전자들이 이종호스트에서는 전사조절시스템이 작동하지 않아 발현이 유도되는 경우가 존재한다. 생산된 대사산물은 외부의 생합성 유전자 클러스터를 가지고 있는 이종 호스트와 가지고 있지 않은 이종 호스트 간에 메타볼롬(metabolome)을 HPLC를 이용하여 비교분석하여 확인할 수 있다.
4. 미발현 생합성 유전자클러스터의 활성화 전략
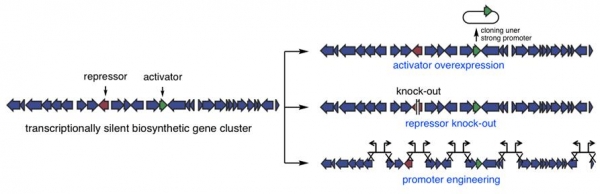
클로닝된 생합성 유전자 클러스터를 이종호스트의 지놈에 삽입하여도 발현되지 않는 경우가 많이 존재하며 대부분의 경우에 이는 생합성 유전자의 전사가 비활성화되어 있기 때문이다. 이종호스트에서 전사가 비활성화되는 이유는 여러 가지가 있을 수 있다. 첫 번째로는 전사조절시스템이 이종호스트에서도 작동하여 특정 조건에서만 활성화되기 때문일 가능성이 있고, 두 번째로는 삽입된 생합성 유전자의 프로모터를 이종호스트의 시그마인자(sigma factor)가 인지하지 못하기 때문일 가능성도 있다. 이러한 경우에는 몇 가지 분자생물학적 방법을 시도해 볼 수 있다(그림 5).
그림 5. 전사 비활성화 되어 있는 생합성 유전자 클러스터를 활성화시키기 위해 사용되는 분자 생물학적 방법의 예
이차대사산물의 생합성 유전자클러스터 내에 존재하는 오페론의 전사는 활성인자(activator) 또는 억제인자(repressor)로 구성된 전사조절인자(transcription factor)에 의해 엄격하게 조절되며 이러한 전사조절인자는 대부분의 경우 생합성 유전자와 함께 클러스터링 되어있다. 이러한 전사인자에 의해 엄격히 조절되는 경우 전사가 활성화되는 배양조건을 찾아내는 것은 매우 어려우며 따라서 분자생물학적 방법인 활성인자 과발현 또는 억제인자 녹아웃에 의해 생합성 유전자를 포함하는 오페론의 전사를 유도하는 방법을 시도해 볼 수 있으며 여러 성공사례가 보고되어왔다[13,14].
이러한 방법에 의해서도 생합성 유전자가 발현되지 않을 경우에는 유전자 클러스터 내에 존재하는 전사조절시스템을 완전히 비활성화시키기 위하여 프로모터 엔지니어링 방법을 시도해 볼 수 있다[15]. 프로모터 엔지니어링 방법은 유전자 클러스터 내에 존재하는 엄격한 전사조절을 받는 프로모터를 인위적으로 전사조절이 가능한 잘 알려진 프로모터로 대체하는 방법이다. 하지만 보통 이차대사산물 생합성 유전자 클러스터는 다수의 오페론으로 구성되어 있어 이들의 프로모터를 모두 대체하는 것은 상당한 시간이 걸리는 비효율적인 작업이었다. 하지만 최근 크리스퍼 기술과 효모의 상동 재조합 기술을 결합하여 다수의 프로모터를 동시에 대체할 수 있는 멀티플렉스 방법이 개발되어 생합성 유전자클러스터의 프로모터 엔지니어링에 걸리는 시간을 상당히 단축시킬 수 있게 되었다[16].
5. 결론 및 전망
앞에서 서술한 바와 같이 메타지노믹스 또는 지노믹스 방법은 차세대 시퀀싱 기술과 결합하여 이전의 전통적인 방법으로는 접근이 불가능했던 미생물 지놈 안에 존재하는 미확인 생합성 유전자 클러스터(cryptic biosynthetic genecluster)로부터 신규 이차대사산물을 발굴하는 것을 가능하게 하였다. 지난 수년간의 바이오인포메틱스 기반의 연구에 의해 이러한 미확인 생합성 유전자클러스터의 수는 현재까지 알려진 이차대사산물의 수보다 훨씬 많다는 것이 확인되었으며 이들은 신약개발에 있어서 중요한 자원으로 각광받고 있다. 미생물의 지놈 또는 메타지놈에 존재하는 미발현생합성 유전자 클러스터로부터 대사산물의 생산을 유도하고 이들의 생리활성을 분석하는 것은 아직도 많은 기술적 어려움이 존재하지만 최근 합성생물학의 급속한 발달로 기술적 어려움이 점차 극복되고 있다. 따라서 향후 수년 안에 이들로부터 생산되는 이차대사산물을 신속하게 탐색하고 생리활성을 테스트하여 신약으로서의 잠재성을 판단할 수 있는 플랫폼이 개발되고 활성화되어 신약개발 파이프라인에서 미생물 유래 이차대사산물이 다시 중요한 역할을 하는 것을 기대해 본다.
참고문헌
1. Demain AL, Sanchez S. 2009. Microbial drug discovery: 80 years of progress. J Antibiot. 62:5-16.
2. Dias DA, Urban S, Roessner U. 2012. A historical overview of natural products in drug discovery. Metabolites. 2:303-336.
3. Carter GT. 2011. Natural products and pharma 2011: strategic changes spur new opportunities. Nat Prod Rep. 28:1783-1789.
4. Cimermancic P, Medema MH, Claesen J, Kurita K, Brown LCW, Mavrommatis K, Pati A, Godfrey PA, Koehrsen M, Clardy J, Birren BW, Takano E, Sali A, Linington RG, Fischbach MA. 2014. Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters. Cell. 158:412-421.
5. Courtois S, Cappellano CM, Ball M, Francou FX, Normand P, Helynck G, Martinez A, Kolvek SJ, Hopke J, Osburne MS, August PR, Nalin R, Guerineau M, Jeannin P, Simonet P, Pernodet JL. 2003. Recombinant environmental libraries provide access to microbial diversity for drug discovery from natural products. Appl Environ Microbiol. 69(1):49-55.
6. Kang HS. 2017. Phylogeny-guided (meta)genome mining approach for the targeted discovery of new microbial natural products. JInd Microbiol Biotechnol. 44(2):285-293.
7. Kallifidas D, Brady SF. 2012. Reassembly of functionally intact environmental DNA-derived biosynthetic gene clusters. Methods Enzymol. 517:225-39.
8. Kang HS, Brady SF. 2013. Arimetamycin A: improving clinically relevant families of natural products through sequence-guided screenign of soil metagenomes. Agew Chem Int Ed Engl. 52(42):11063-11067.
9. Kang HS, Brady SF. 2014. Mining soil metagenomes to better understand the evolution of natural product structural diversity:pentangular polyphenols as a case study. J Am Chem Soc. 136(52):18111-18119.
10. Doroghazi JR, Albright JC, Goering AW, Ju KS, Haines RR, Tchalukov KA, Labeda DP, Kelleher NL, Metcalf WW. 2014. Aroadmap for natural product discovery based on large-scale genomics and metabolomics. Nat Chem Biol. 10(11):963-968.
11. Yamanaka K, Reynolds KA, Kersten RD, Ryan KS, Gonzalez DJ, Nizet V, Dorrestein PC, Moore BS. 2014. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A. Proc Natl Acad Sci USA. 111(5):1957-62.
12. Ongley SE, Bian X, Beilan BA, Muller R. 2013. Recent advances in the heterologous expression of microbial natural product biosynthetic pathways. Nat Prod Rep. 30(8):1121-38.
13. Kallifidas D, Kang HS, Brady SF. 2012. Tetarimycin A, an MRSA-active antibiotic identified through induced expression of environmental DNA gene clusters. 134(48):19552-5.
14. Gottelt M, Kol S, Gomez-Escribano JP, Bibb M, Takano E. 2010. Deletion of a regulatory gene within the cpk gene cluster reveals novel antibacterial activit yin Streptomyce scoelicolor A3(2). Microbiology 156:2343-53.
15. Montiel D, Kang HS, Chang FY, Charlop-Powers Z, Brady SF. Yeast homologous recombination-based promoter engineering of the activation of silent natural product biosynthetic gene clusters. Proc Natl Acad Sci USA. 112(29):8953-8.
16. Kang HS, Charlop-Powers Z, Brady SF. 2016. Multiplexed CRISPR/Cas9- and TAR-mediated promoter engineering of natural product biosynthetic gene clusters in yeast. ACS Synth Biol. 5(9):1002-10.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update