| In vivo 단백질 진화 – 차세대 단백질 진화법 | |
|---|---|
| Date 2019-04-08 17:32:30 |
|

교수 박사과정
서울대학교 화학생물공학부
byungkim@snu.ac.kr jinyohg@gmail.com
1. In vitro 단백질 진화법
In vitro 실험을 통한 단백질 진화는 PCR[1]이 소개된 이후로 지속적으로 개발 및 발전되어 왔다. Error-prone PCR, DNA shuffling 등의 기술을 통해 유전자에 random으로 돌연변이를 도입하거나(단백질에 있어서 잔기의 변환의 결과를 가져옴), 같은 기능을 하는 유전자들의 단편들을 무작위로 섞는 방법(단백질에 있어서 도메인 혹은 모티프 전체를 변환하는 결과를 가져옴)을 통해 돌연변이 유전자를 만들고, 이를 돌연변이 유전자로부터 연구자가 필요로 하는 특성을 띄는 유전자를 골라내는 방법이 주된 연구방법이었다[2-5]. 그러나 이러한 무작위 돌연변이법은 고효율의 선별법이 반드시 필요했다. 왜냐하면, 무작위 돌연변이법은 수많은 활성(혹은 특성) 감소 돌연변이(deleterious mutation)를 생산해내는 특징이 있었고, 또한 천문학적인 숫자의 돌연변이를 만들어내므로, 이와 같은 천문학적인 숫자의 활성 감소 돌연변이들 중에 숨어있는 활성(혹은 특성) 증진 돌연변이(beneficial mutation)를 찾아내기 위해서는 고효율의 선별법이 반드시 필요하였다. 이를 위해 1) microplate 상에서 각각의 돌연변이주에 대해 반응을 수행하여, 반응이 수행되었을 경우 반응혼합물의 색깔이 변화하도록 하여 이를 관찰 혹은 분석하는 방법, 2) 목표화합물을 유일한 영양소로 제공하고 이를 활발히 활용하는 돌연변이를 포함하는 세포가 잘 자라는 것을 활용한 세포성장 기반 선별법(예, enrichment culture), 3) 항생제 저항성을 획득하는 돌연변이 단백질을 개발하기 위하여 항생제를 포함하는 agar plate 상에서 세포의 사멸 혹은 생존을 통해 활성 돌연변이를 선별하는 방법, 혹은 4) FACS와 같은 고효율 선별 기술을 통해 선별하는 등의 고효율 선별법 등이 동원되어 왔다(표 1 참조). 이런 기술만으로도 천문학적인 돌연변이 라이브러리로부터 활성 돌연변이를 찾아내는 것이 쉽지 않았기 때문에, 연구자들은 라이브러리의 숫자를 줄이는 기술 또한 개발하여 왔다. 이의 한 예가 smart library 구현이었고, 생물정보학적 분석 혹은 분자모델링 기법을 사용하여 목적 단백질 내의 중요한 모티프 혹은 잔기에 대해서만 돌연변이를 일으키는 방법을 통해 라이브러리 숫자를 줄이려는 노력이 행하여져 왔다[6-8]. 이러한 노력의 가장 극단적인 경우가 de novo 단백질 디자인인데, 이는 단백질 시퀀스 분석이나 구조분석 없이 활성 단백질이나 안정한 단백질 scaffold를 만들어내는 노력이었다[9]. 또한, 무작위 돌연변이법과 de novo 단백질 디자인법을 혼합한 hybrid method 또한 행하여져 왔는데, 이는 최근 들어서 가장 많이 적용되는 단백질 진화법이다[10]. 이에 비하여 in vivo 진화법은 돌연변이 유발과 활성이 증진된 돌연변이 균주 혹은 단백질을 세포 안에서 동시에 생성할 수 있는 장점 덕분에 주목을 받게 되었다. 이러한 in vivo 돌연변이법에는 무작위 in vivo 돌연변이법 및 target 특이적 in vivo 돌연변이법의 두 가지 접근법이 존재한다. 그림 1에서는 현재까지의 in vitro 진화법과 in vivo 진화법의 개발의 시간 순서로 소개되어 있다.
표 1. 빈번하게 사용되는 고효율 선별법 및 단백질 진화 적용
Methods |
Method summary |
Examples |
References |
Microplate assay |
Selection of mutants by assaying activity, cofactor consumption, etc |
Measured NAD(P)H consumption and H2O2 formation to screen for P450 BM3 mutants; Colorimetric assay to find mutants with increased activity of α2,3/2,6-sialyltransferase; Colorimetric assay to select CYP153A35 mutants with enhanced ω-hydroxylation of palmitic acid |
[11-13] |
Antibiotics |
Mutant selection via cell live or death under antibiotics |
Selection of trimethoprim-resistant mutants; Selection of spectinomycin-resistant mutants; Selection of trimethoprimresistant mutants |
[14-16] |
Enrichment culture |
Selection of mutants by differing cell growth rate |
Selection of mutants of AlkB and CYP153A6 with increased terminal alkane hydroxylation activity via enrichment culture in alkane medium; Selection of ω-transaminase mutants with increased resistance to product inhibition via enrichment culture in aliphatic ketone medium |
[17, 18] |
FACS |
Sorting of mutant cells by differing fluorescence |
Selection of Cre recombinase mutants with differing loxP specificity; Selection of TEV protease mutants with change in substrate specificity; Selection of sialyltransferase mutants with increased catalytic efficiency |
[19-21] |
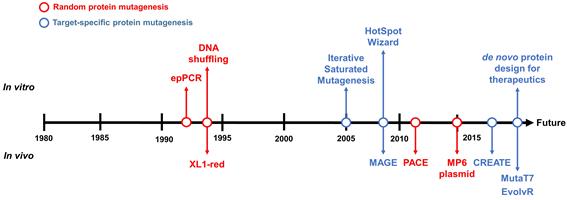
그림 1. In vitro 진화법과 in vivo 진화법의 개발 역사
2. 무작위 in vivo 돌연변이법
최근 들어, 돌연변이 유발과 활성 돌연변이 선별을 모두 세포 안에서 한꺼번에 수행하고자 하는 연구가 소개되고 진행되고 있다. 무작위 in vivo 돌연변이 방법들 중 가장 활발하게 이용되고 있는 방법은 XL-1 Red 대장균 균주를 사용하는 방법이다. Greener 등은 DNA 수복(修復)에 관여하는 세 개의 유전자에 돌연변이를 도입함으로써 대장균이 DNA 복제 시에 스스로 돌연변이를 많이 유발하도록 하였다[22]. 이 균주는 mutS (DNA mismatch repair protein), mutT(8-oxo-dGTP diphosphatase), mutD (DNA pol III exonuclease)에 활성 감소 돌연변이(deleterious mutation)가 도입되어 있어(그림 1A), 야생주 대장균보다 약 5000배가량 많은 돌연변이가 발생한다(표 2 참조). Ehrig 등은 XL1-red strain을 사용하여 green-fluorescent protein(GFP)의 random library를 만들었고 기존과는 다른 파장의 빛을 받아들여 들뜬상태가 되는 돌연변이 단백질을 찾아내었다[34]. 또 다른 예는 University of Manchester의 Nicholars Turner 그룹에 의해 보고된 monoamine oxidase의 광학선택성을 증가시킨 것이다[35]. Turner 그룹에서는 XL-1 r e d 균주를 사용하여 (S)-광학이성질체에 대한 선택성이 높은 돌연변이 효소를 찾음으로써 (S)-α-methylbenzylamine에 대한 높은 광학선택성을 보이는 효소를 개발하였다. XL-1 red 균주는 Lactobacillus gasseri 유래 β-glucuronidase (GusA)의 sub-optimal pH에서의 활성을 높이는 연구에도 적용되었다. Callanan 등은 돌연변이 효소 라이브러리 중 pH 5에서 가장 높은 활성을 보이는 돌연변이 효소를 찾아내었는데, 이 돌연변이 효소는 중성 pH에서 야생주 효소보다 더 높은 활성을 보여주었다[36].
대부분의 무작위 in vivo 돌연변이 라이브러리 구축 시스템은 XL-1 red 균주를 사용하는 방법보다 더 좋은 결과를 보여주지는 못하였다. 그러나 2015년에 하버드대의 David Liu 그룹은 무작위 in vivo 돌연변이 제작법에서 좋은 결과를 보여주었는데, 그들은 넓은 돌연변이 범위와 빈도(~ 6.2×10-6 μbp)를 보여주는 mutator plasmid인 MP6를 개발하였다[37]. MP6 mutator plasmid는 원래 phage-assisted continuous evolution (PACE)에서 돌연변이 비율을 높이기 위해 개발되었으나 이의 성능을 더욱 높인 것이다. Arabinose에 의해 발현유도 되는 PBAD 프로모터를 사용하여 발현을 조절 하에, 활성이 떨어지는 돌연변이를 함유하고 있는 DNA pol III (dnaQ926 ), DNA methylase (dam ), hemimethylated GATC-binding domain (seqA ), uracil-DNA glycosylase inhibitor (ugi ), cytidine deaminase (cda1 ), transcription repressor for intracellular dNTP pool (emrR )들을 발현하여, 야생주 대장균 chromosome에 322,000배가량 증가된 돌연변이 비율을 구현하여, 항생제 내성 균주, T3 프로모터에서 작동할 수 있는 T7 RNA polymerase 개발 등 다양한 돌연변이 균주 혹은 단백질을 매우 빠른 시간 안에 개발할 수 있는 시스템을 개발하였다(그림. 2B). 또한, Liu 그룹은 이 방법을 사용하여 CRIPSR-Cas 시스템 상에서 Cas9 효소가 다양한 종류의 PAM 서열(NG, GAA, GAT 등)을 인지할 수 있도록 돌연변이 효소를 만들었다[38,40]. 이러한 예들은 무작위 돌연변이 유발과 이중 활성(혹은 특성)이 증진된 돌연변이 효소(혹은 단백질)를 동시에 그리고 빠른 속도로 개발할 수 있는 가능성을 보여주었다. 무작위 in vivo 돌연변이 방법인 MP6 플라스미드와 PACE 등이 개발되어 사용되었으나, 이러한 무작위 in vivo 돌연변이법은 off-target 효과가 많이 일어나는 단점이 있다. 이러한 off-target 효과는 세포를 사멸로 이끌 수 있기 때문에, 현재의 대부분의 방법은 target 특이적인 in vivo 돌연변이법에 더욱 주목하고 있다.
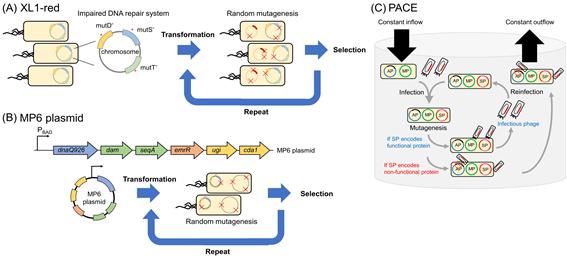
그림 2. 무작위 in vivo 진화법의 개요. (A) XL1-red 균주의 개요. XL1-red는 세 개의 DNA 수복(修復) 효소(mutD, mut S, mutT )의 기능이 정상 작동하지 않음으로 인해 돌연변이를 스스로 유발할 수 있는 대장균 균주이다. (B) MP6 플라스미드의 개요. MP6는 inducer로 발현 유도되는 돌연변이 유발 효소를 포함하는 플라스미드이다. 이것은 넓은 범위의 돌연변이 종류와, Pol III의 돌연변이와 망가진 수복(修復) 기능으로 인한 높은 돌연변이 비율을 유발하는 특징이 있다. (C) PACE의 개요. PACE는 phageassisted continuous evolution 법으로, 대장균 내의 유전자의 돌연변이를 유발하는 매개법으로써 M13 phage의 생존을 활용하는 방법이다.
표 2. 단백질에 대한 돌연변이 유발법 및 그의 응용
Methods | Method summary | In vivo or in vitro? | Specificity | Mutation rate (μbp)* | Applications | References |
epPCR | A PCR-based mutagenesis method that utilizes an errorprone DNA polymerase to insert errors randomly | In vitro | Random | N/A | Improved proteolytic activity of subtilisin BPN’ in cold temperatures; Improved Coprinus cinereus (CiP) heme peroxidase stability and activity under laundry condition | [23-25] |
DNA shuffling | A PCR-based method that utilizes DNase to cut DNA in fragments then allow them to prime each other for random combinatorial library | In vitro | Random | N/A | Increased resistance of TEM-1 β-lactamase against cefotaxime; Increased substrate specificity of β-galactosidase toward nitrophenyl fucopyranoside; Improved fluorescence signal of GFP; Increased resistance of ars operon against arsenate | [26-29] |
Site-saturated mutagenesis | A mutagenesis method that substitutes a specified amino acid to any of 19 other amino acids | In vitro | Targetspecific | N/A | Substrate specificity change for catechol 1,2-dioxygenase; Improved uridine diphosphogalactose 4-epimerase activity on tagatose; Increased lactose phosphorylase activity of cellobiose phosphorylase from Cellulomonas uda | [30-33] |
XL1-red | an E. coli mutator strain with three repair pathways inactivated | In vivo | Random | ~10-6 | Modified fluorescence of GFP; Improved enantioselectivity of monoamine oxidase; Improved activity of â-glucuronidase in neutral pH | [22, 34-36] |
MP6 plasmid | a small-molecule inducible mutagenesis plasmidthat offers broad mutational spectra andhigh mutational rates by using mutated Pol III and impaired repair pathways | In vivo | Random | ~6.2x10-6 | Increased resistance to number of antibiotics; Evolution of Cas9 to engineer PAM compatibility and DNA specificity | [37, 38] |
PACE | a phage-assisted continuous evolution method that utilizes the survival of M13 phage to mutate genes in E. | In vivo | Random | ~10-7 | Modified specificity of T7 RNA polymerase toward T3 promoter; Modified protein-protein interaction of Bt toxin to bind a novel receptor; Modified specificity of TEV protease toward a totally different sequence; Combined with AND gate to improve cytosolic expression of misfolded antibodies and the rat APOBEC1 | [39-42] |
MAGE | a multiplex genome engineering method that utilizes singlestranded DNAs and recombination to mutate multiple loci in genome | In vivo | Target-specific | N/A | Optimized metabolic flux of DXP pathway to overproduce lycopene in E. coli; Improved the production of indigo and indirubin; Reassignment of 314 stop codons in E. coli to measure individual recombination frequencies and viabilities | [43-45] |
MutaT | a method utilizing a chimeric protein of T7 RNA polymerase and cytidine deaminase to specifically mutate genes downstream of T7 promoter | In vivo | Target-specific | N.D. | Improved resistance of dihydrofolate reductase to trimethoprim | [16] |
EvolvR | a method utilizing a chimeric protein of error-prone Pol I and nicking variant of Cas9to specifically mutate genes targeted by gRNA | In vivo | Target-specific | 10-5 ~ 10-6 | Increased resistance of ribosomal protein subun it to spectinomycin | [14] |
CREATE | a multiplex genome engineering method that utilizes CRISPRCas9 with barcodetracking of mutations | In vivo | Target-specific | N/A | Improved resistance of dihydrofolate reductase to trimethoprim; Metabolic engineering of E. coli to produce isopropanol; Increased NADPH pool with increased 3-hydroxypropionic acid production as well | [15, 46, 47] |
* Mutation rate (μbp)는 mutation/bp/generation의 rate와 같다. N/A: not applicable. N.D. :not determined.
3. 목표 특이적 in vivo 돌연변이법
차세대 시퀀싱(Next generation sequencing (NGS))이 발전하고, DNA 합성 가격이 큰 폭으로 내려감에 따라, in vivo 진화법을 위한 최신의 기술들이 가능해지게 되었다. 하버드대의 George Church 그룹은 자동 다중 유전체 엔지니어링 기술법(automatic, multiplex genome engineering (MAGE))을 개발하였는데, 이 방법은 단일 가닥의 DNA(single-stranded oligonucleotides (ssOligo))와 lambda Red recombinase (λ-Red)의 β protein을 사용하여 대장균 유전체의 목표 부위를 특이적으로 변화시키는 방법이다[43]. Church 그룹은 이 방법을 통해 세포 배양, β protein 발현, 목표부위 DNA 서열에 돌연변이 혹은 염기 삽입/제거를 포함하는 ssOligo 삽입, 세포 재건을 자동적으로 수행하는 방법을 개발하여 유전체에 돌연변이를 유발하는 방법을 완전 자동화 및 여러 돌연변이를 동시에 수행하는 방법을 개발하였다(그림 3A). 이렇게 개발된 MAGE법 시험을 위해, 대장균에서 lycopene을 더 많이 생산할 수 있도록 DXP 생합성 경로를 개량하여 metabolic flux를 최적화하였다. 이를 위해 isoprenoid 생산 유전자의 ribosome binding site를 개량하여 최종적으로 lycopene 생산이 증진된 균주를 만들었다[43]. 또한 Church 그룹은 Cos-MAGE법을 개발하였는데, 이 방법은 돌연변이를 더 잘 보유할 수 있는 돌연변이주를 더 많이 포함하도록 하여 돌연변이 라이브러리 크기를 더욱 크게 하는 방법이다[44]. 이러한 Cos-MAGE법을 사용하여 indigo와 indirubin 생산 유전자의 upstream에 T7 프로모터를 삽입하고 최종적으로 8.6 mg g-1 dry cell weight의 indigo를 생산할 수 있는 대장균 돌연변이주를 제작하였다. 이에 더하여 Isaacs 등은 MAGE법을 활용하여 hierarchical conjugative seembly genome engineering (CAGE) 법을 개발하였는데, 이 방법은 대장균 유전체에 대하여 MAGE를 사용하여 돌연변이를 유발하고, 이들 각각의 대장균 돌연변이를 gene transfer를 사용하여 하나로 합치는 방법이다[45]. 이 연구를 통해 연구진은 314개의 TAG stop codon을 모두 TAA stop codon으로 바꾸는 것을 성공적으로 수행하였다. 그 결과로, 각각의 mutation이 cell viability와 recombination frequency에 미치는 영향을 평가할 수 있었다.
또 다른 목표 특이적 in vivo 돌연변이 라이브러리 구축법은 T7 RNA polymerase와 base editing 능력이 있는 cytidine deaminase를 활용하는 방법이다. Moore 등은 MutaT7이라는 chimera 효소를 개발하였는데, 이 효소는 T7 프로모터에 결합하여 T7 프로모터가 관할하는 유전자에 특이적으로 돌연변이를 유발하는 방법이다(그림 3B). 연구진은 이 방법을 사용하여 trimethoprim에 대한 dihydrofolate reductase (DHFR)의 저항성을 높이는 연구를 수행하였다[16].
CRISPR-Cas9 시스템의 발견은 유전체 편집에 있어 획기적인 전기를 마련하였다. 이 시스템은 유전체 편집에 사용되지만, in vivo 돌연변이 유발에 응용되기도 한다. CRISPR-Cas9 시스템은 guide RNA와 Cas9 endonuclease가 사용되는데, Cas9이 위치특이적으로 유전체를 자르게 되면 잘려진 유전체는 homologous recombination(HR)이나 non-homologous end joining (NHEJ)를 통해 복구된다. 이러한 복구과정을 활용하여 유전체에 돌연변이를 유발하는 방법이 개발되기도 하였다. Halperin 등은 EvolvR법을 개발하였는데, 이 시스템은 guide RNA, Cas9 효소의 돌연변이인 nickase, 에러를 유발하는 DNA polymerase I를 활용하는 방법이다. nickase와 Pol I이 pTarget 플라스미드에 결합하여 지속적으로 돌연변이를 유발하는 방법이다(그림 3C). 이 방법은 더 보완되었는데, thioredoxin-binding domain을 활용하고, off-target effect를 감소시켜 불완전한 퓨전단백질의 발현을 감소시켰다. 이 방법을 응용하여 spectinomycin에 저항성이 우수한 대장균을 개발하였다[14].
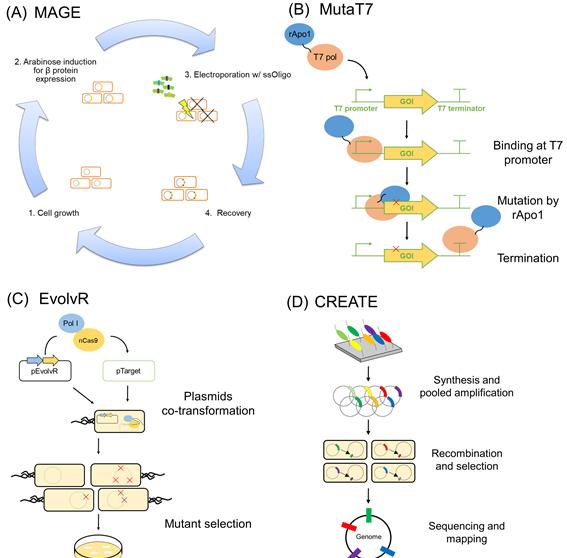
그림 3. 목표 특이적 in vivo 돌연변이법의 개요. (A) MAGE법의 개요. MAGE는 목표부위에 대한 돌연변이를 포함하는 단일가닥 DNA를 주입하여 여러 부위에 대해 동시에 재조합을 일으킴으로써 유전체에 대한 다중 돌연변이를 유발하는 방법이다. (B) EvolvR법의 개요. EvolvR은 에러를 유발하는 Pol I의 chimeric 단백질과 Cas9의 돌연변이 효소인 nickase를 활용하여 guide RNA가 목표로 하는 부분에 특이적으로 돌연변이를 일으키는 방법이다. (C) MutaT7법의 개요. MutaT7은 T7 RNA polymerase의 돌연변이 효소와 cytidine deaminase를 사용하여 T7 프로모터에 의해 전사되는 유전자에 특이적으로 돌연변이를 일으키는 방법이다. (D) CREATE법의 개요. CREATE는 CRISPR-Cas9 시스템을 사용함과 동시에 mutation에 대한 barcode-tracking법을 사용
하는 유전체 다중 엔지니어링 기술이다.
단일 염기쌍에 대한 돌연변이 제작을 통해, 더 한층 특이적인 in vivo 돌연변이법이 개발되었다. Liang 등은 CREATE법을 개발하였다(그림 3D). 이 방법은 CRISPR-enabled trackable genome engineering (CREATE)이라고 명명되었는데, 설계된 guide RNA와 homology arm을 활용하는 방법으로, 이들 DNA에는 PAM 서열에 mutation이 삽입되어 있어, 추후의 homologous recombination 후에 Cas9에 의한 DNA 잘림을 방지하는 것을 목표로 하였다. 이 방법을 통해 DHFR 단백질의 모든 잔기에 대해 saturation mutagenesis library를 구축하여, 그로부터 trimethoprim 저항성 DHFR를 선별하였다. 이러한 trimethoprim 저항성 효소는 기존에 보고된 세 가지의 돌연변이뿐만 아니라 기존에 보고되지 않았던 일곱 가지의 새로운 돌연변이를 찾아내었다. Liang 등은 이 방법을 대장균의 isopropanol 생산경로에도 적용하였다[46]. Isopropanol 생산경로 상에 있는 네 개의 유전자에 대하여 그들의 ribosome binding site에 대하여 돌연변이를 유발하였고, 이로부터 7.1 g/L의 isopropanol을 생산할 수 있는 대장균 돌연변이주를 제작할 수 있었다. CREATE는 cofactor regeneration에도 적용되었는데, Reynolds 등은 NAD(P)H 생성에 관여하는 11개의 유전자를 선정하고, 25,900개의 돌연변이를 그들 유전자에 유발시켰다. 이러한 돌연변이 균주 중 reactive oxygen에 저항성이 있는 균주를 선별하는 방법을 통해 그들이 원하는 균주를 선별하였다[47]. Reactive oxygen stress를 selection pressure로 사용한 이유는, NADPH/NADP+ 비율이 증가하게 되면 reactive oxygen stress에 대한 저항성이 증가하기 때문이었다. 그 결과, CREATE를 사용하여 NADPH/NADP+ 비율이 증가된 돌연변이 균주를 선별할 수 있었고, 이 균주는 야생주 대장균 보다 2배 이상 증가된 6.4 ± 0.5 g/L의 3-hydroxypropionic acid를 생산하는 돌연변이 균주를 제작할 수 있었다.
4. 결론
In vitro 돌연변이법이 개발되어 사용된 후, 이를 이용하여 고효율로 돌연변이주를 선별하기 위하여, 고효율 돌연변이 선별법이 고안되었고, 또한, 목표 특이적인 돌연변이 유발을 위하여 ‘smart library’ 설계가 제안되기도 하였다. In vivo 돌연변이법 또한 유사한 길을 걸어오고 있는 것으로 보인다. In vivo 돌연변이법은 돌연변이를 추적할 수 있는 방법이 어렵고, 돌연변이를 일으킨 부위 이외에 돌연변이가 유발될 수 있는 가능성(소위, off-target effect) 때문에, 사용이 제한되어 온 것이 사실이다. 그러나, CRISPR-Cas 시스템의 발달과 더불어, DNA 합성 가격 및 NGS 분석 가격이 하락함에 따라 이러한 최신 기술을 적용하는 in vivo 돌연변이법 개발이 가속화되고 있다. 또한, 이러한 최신 기술이 적용됨에 따라, 무작위 돌연변이 속에서 특성이 개량된 돌연변이를 선별하고 이들 돌연변이를 추적할 수 있는 기술 또한 적용 가시권에 들어오고 있다. In vivo 돌연변이법은 in vitro 돌연변이법에 비해 부가적인 유전자조작 개입이 필요하지 않으며 단백질의 특성을 세포의 성장과 동시에 빠르게 확인할 수 있다는 장점이 있다. 그러나 돌연변이 균주의 특성을 잘 나타낼 수 있는 고효율 선별 시스템의 개발은 in vivo 돌연변이법에서도 여전한 숙제이다. 아직 미성숙된 기술이기는 하나, in vivo 돌연변이법은 여러 가지 장점을 가진 실험법이다. 따라서 앞으로 더 많은 방법들이 고안되면 균주 및 단백질 진화에 있어 굉장히 활용도가 높은 연구방법이 될 것으로 생각된다.
참고문헌
1. Mullis, K. B. (1985). US Patent No. US4683202A., Cetus Corp.
2. Moore, J. C., and F. H. Arnold (1996) Directed evolution of a para-nitrobenzyl esterase for aqueous-organic solvents. Nature Biotechnology. 14: 458.
3. Gram, H., L.-A. Marconi, C. F. Barbas, T. A. Collet, R. A. Lerner, and A. S. Kang (1992) In vitro selection and affinity maturation of antibodies from a naive combinatorial immunoglobulin library. Proceedings of the National Academy of Sciences. 89: 3576.
4. Liao, H., T. McKenzie, and R. Hageman (1986) Isolation of a thermostable enzyme variant by cloning and selection in a thermophile. Proceedings of the National Academy of Sciences. 83: 576.
5. Chen, K., and F. H. Arnold (1993) Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. Proceedings of the National Academy of Sciences. 90: 5618.
6. Karplus, M., and G. A. Petsko (1990) Molecular dynamics simulations in biology. Nature. 347: 631.
7. Leach, A. R. (1994) Ligand docking to proteins with discrete side-chain flexibility. Journal of Molecular Biology. 235: 345.
8. Šali, A., L. Potterton, F. Yuan, H. van Vlijmen, and M. Karplus (1995) Evaluation of comparative protein modeling by MODELLER. Proteins: Structure, Function, and Bioinformatics. 23: 318.
9. Huang, P.-S., S. E. Boyken, and D. Baker (2016) The coming of age of de novo protein design. Nature. 537: 320.
10. Chica, R. A., N. Doucet, and J. N. Pelletier (2005) Semi-rational approaches to engineering enzyme activity: combining the benefits of directed evolution and rational design. Current Opinion in Biotechnology. 16: 378.
11. Morlock, L. K., D. Böttcher, and U. T. Bornscheuer (2018) Simultaneous detection of NADPH consumption and H2O2 production using the Ampliflu™ Red assay for screening of P450 activities and uncoupling. Applied Microbiology and Biotechnology. 102: 985.
12. Choi, Y. H., J. H. Kim, J. H. Park, N. Lee, D.-H. Kim, K.-S. Jang, I.-H. Park, and B.-G. Kim (2013) Protein engineering of α2, 3/2, 6-sialyltransferase to improve the yield and productivity of in vitro sialyllactose synthesis. Glycobiology. 24: 159.
13. Jung, E., B. G. Park, H.-W. Yoo, J. Kim, K.-Y. Choi, and B.-G. Kim (2018) Semi-rational engineering of CYP153A35 to enhance ω-hydroxylation activity toward palmitic acid. Applied Microbiology and Biotechnology. 102: 269.
14. Halperin, S. O., C. J. Tou, E. B. Wong, C. Modavi, D. V. Schaffer, and J. E. Dueber (2018) CRISPR-guided DNA polymerases enable diversification of all nucleotides in a tunable window. Nature. 560: 248.
15. Garst, A. D., M. C. Bassalo, G. Pines, S. A. Lynch, A. L. Halweg-Edwards, R. Liu, L. Liang, Z. Wang, R. Zeitoun, and W. G. Alexander (2017) Genome-wide mapping of mutations at single-nucleotide resolution for protein, metabolic and genome engineering. Nature Biotechnology. 35: 48.
16. Moore, C. L., L. J. Papa III, and M. D. Shoulders (2018) A Processive protein chimera introduces mutations across defined DNA regions in vivo. Journal of the American Chemical Society. 140: 11560.
17. Koch, D. J., M. M. Chen, J. B. van Beilen, and F. H. Arnold (2009) In vivo evolution of butane oxidation by terminal alkane hydroxylases AlkB and CYP153A6. Applied and Environmental Microbiology. 75: 337.
18. Yun, H., B.-Y. Hwang, J.-H. Lee, and B.-G. Kim (2005) Use of enrichment culture for directed evolution of the Vibrio fluvialis JS17 ω-transaminase, which is resistant to product inhibition by aliphatic ketones. Applied and Environmental Microbiology. 71: 4220.
19. Santoro, S. W., and P. G. Schultz (2002) Directed evolution of the site specificity of Cre recombinase. Proceedings of the National Academy of Sciences. 99: 4185.
20. Yi, L., M. C. Gebhard, Q. Li, J. M. Taft, G. Georgiou, and B. L. Iverson (2013) Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proceedings of the National Academy of Sciences. 110: 7229.
21. Aharoni, A., K. Thieme, C. P. Chiu, S. Buchini, L. L. Lairson, H. Chen, N. C. Strynadka, W. W. Wakarchuk, and S. G. Withers (2006) High-throughput screening methodology for the directed evolution of glycosyltransferases. Nature Methods. 3: 609.
22. Greener, A., M. Callahan, and B. Jerpseth (1997) An efficient random mutagenesis technique using an E. coli mutator strain. Molecular Biotechnology. 7: 189.
23. Zhou, Y., X. Zhang, and R. H. Ebright (1991) Random mutagenesis of gene-sized DNA molecules by use of PCR with Taq DNA polymerase. Nucleic Acids Research. 19: 6052.
24. Kano, H., S. Taguchi, and H. Momose (1997) Cold adaptation of a mesophilic serine protease, subtilisin, by in vitro random mutagenesis. Applied Microbiology and Biotechnology. 47: 46.
25. Cherry, J. R., M. H. Lamsa, P. Schneider, J. Vind, A. Svendsen, A. Jones, and A. H. Pedersen (1999) Directed evolution of a fungal peroxidase. Nature Biotechnology. 17: 379.
26. Stemmer, W. P. (1994) Rapid evolution of a protein in vitro by DNA shuffling. Nature. 370: 389.
27. Zhang, J.-H., G. Dawes, and W. P. Stemmer (1997) Directed evolution of a fucosidase from a galactosidase by DNA shuffling and screening. Proceedings of the National Academy of Sciences. 94: 4504.
28. Crameri, A., E. A. Whitehorn, E. Tate, and W. P. Stemmer (1996) Improved green fluorescent protein by molecular evolution using DNA shuffling. Nature Biotechnology. 14: 315.
29. Crameri, A., G. Dawes, E. Rodriguez Jr, S. Silver, and W. P. Stemmer (1997) Molecular evolution of an arsenate detoxification pathway by DNA shuffling. Nature Biotechnology. 15: 436.
30. Higuchi, R., B. Krummel, and R. Saiki (1988) A general method of in vitro preparation and specific mutagenesis of DNA fragments: study of protein and DNA interactions. Nucleic Acids Research. 16: 7351.
31. Caglio, R., F. Valetti, P. Caposio, G. Gribaudo, E. Pessione, and C. Giunta (2009) Fine-Tuning of Catalytic Properties of Catechol 1, 2-Dioxygenase by Active Site Tailoring. Chembiochem. 10: 1015.
32. Kim, H.-J., S. Y. Kang, J. J. Park, and P. Kim (2011) Novel activity of UDP-galactose-4-epimerase for free monosaccharide and activity improvement by active site-saturation mutagenesis. Applied Biochemistry and Biotechnology. 163: 444.
33. De Groeve, M. R., M. De Baere, L. Hoflack, T. Desmet, E. J. Vandamme, and W. Soetaert (2009) Creating lactose phosphorylase enzymes by directed evolution of cellobiose phosphorylase. Protein Engineering, Design & Selection. 22: 393.
34. Ehrig, T., D. J. O’Kane, and F. G. Prendergast (1995) Green-fluorescent protein mutants with altered fluorescence excitation spectra. FEBS letters. 367: 163.
35. Carr, R., M. Alexeeva, A. Enright, T. S. Eve, M. J. Dawson, and N. J. Turner (2003) Directed evolution of an amine oxidase possessing both broad substrate specificity and high enantioselectivity. Angewandte Chemie International Edition. 42: 4807.
36. Callanan, M. J., W. M. Russell, and T. R. Klaenhammer (2007) Modification of Lactobacillus β-glucuronidase activity by random mutagenesis. Gene. 389: 122.
37. Badran, A. H., and D. R. Liu (2015) Development of potent in vivo mutagenesis plasmids with broad mutational spectra. Nature Communications. 6: 8425.
38. Hu, J. H., S. M. Miller, M. H. Geurts, W. Tang, L. Chen, N. Sun, C. M. Zeina, X. Gao, H. A. Rees, and Z. Lin (2018) Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556: 57.
39. Esvelt, K. M., J. C. Carlson, and D. R. Liu (2011) A system for the continuous directed evolution of biomolecules. Nature. 472: 499.
40. Badran, A. H., V. M. Guzov, Q. Huai, M. M. Kemp, P. Vishwanath, W. Kain, A. M. Nance, A. Evdokimov, F. Moshiri, and K. H. Turner (2016) Continuous evolution of Bacillus thuringiensis toxins overcomes insect resistance. Nature. 533: 58.
41. Packer, M. S., H. A. Rees, and D. R. Liu (2017) Phage-assisted continuous evolution of proteases with altered substrate specificity. Nature Communications. 8: 956.
42. Wang, T., A. H. Badran, T. P. Huang, and D. R. Liu (2018) Continuous directed evolution of proteins with improved soluble expression. Nature Chemical Biology. 14: 972.
43. Wang, H. H., F. J. Isaacs, P. A. Carr, Z. Z. Sun, G. Xu, C. R. Forest, and G. M. Church (2009) Programming cells by multiplex genome engineering and accelerated evolution. Nature. 460: 894.
44. Wang, H. H., H. Kim, L. Cong, J. Jeong, D. Bang, and G. M. Church (2012) Genome-scale promoter engineering by coselection MAGE. Nature Methods. 9: 591.
45. Isaacs, F. J., P. A. Carr, H. H. Wang, M. J. Lajoie, B. Sterling, L. Kraal, A. C. Tolonen, T. A. Gianoulis, D. B. Goodman, and N. B. Reppas (2011) Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science. 333: 348.
46. Liang, L., R. Liu, A. D. Garst, T. Lee, G. T. Beckham, and R. T. Gill (2017) CRISPR enabled trackable genome engineering for isopropanol production in Escherichia coli. Metabolic engineering. 41: 1.
47. Reynolds, T. S., C. M. Courtney, K. E. Erickson, L. M. Wolfe, A. Chatterjee, P. Nagpal, and R. T. Gill (2017) ROS mediated selection for increased NADPH availability in Escherichia Coli. Biotechnology and Bioengineering. 114: 2685.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update 
