| 염기교정 유전자가위의 개발과 활용 | |
|---|---|
| Date 2019-10-05 18:23:22 |
|

박사
한국생명공학연구원 합성생물학전문연구단
1. 서론
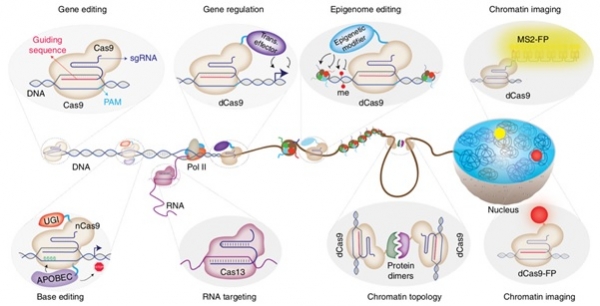
2012년 Jennifer Anne Doudna와 Emmanuelle Charpentier 연구진은 박테리아에서 유래된 유전자가위인 CRISPR-Cas9 시스템을 <Science>에 최초로 보고하였다. 이 크리스퍼 유전자가위는 sgRNA(single guide RNA)와 Cas9 단백질로 구성되어 있고 sgRNA의 20bp 서열만 설계하여 발현하면 sgRNA에 의해 표적 서열로 유도된 Cas9 단백질이 DNA를 절단할 수 있다. 이는 기존에 보고된 유전자가위인 ZFN(Zinc Finger Nuclease)과 TALEN(Transcription Activator-Like Effector Nucleases)에 비하여 쉬운 제작, 높은 효율, 높은 서열 특이성을 가지고 작동하기 때문에 생명과학 및 생명공학 분야에서 유전체 편집 도구로 큰 각광을 받아 왔다. 현재 크리스퍼 유전자가위는 질병의 진단 및 치료, 동식물 품종 개량 등에 폭넓게 활용되어 바이오 분야에 혁명을 불러일으키고 있다. CRISPR-Cas9 시스템은 유전체 편집 뿐만 아니라 유전자의 발현 조절, 후성유전체(epigenome) 편집, 염색질(chromatin) 이미징 등의 다양한 연구 분야에 적용할 수 있다(그림1)[1]. 이는 CRISPR-Cas9 시스템의 구성 성분인 Cas9 단백질과 sgRNA가 모듈러 디자인(modular design)이 가능하기 때문에 생기는 장점으로 특정 기능을 나타내는 모듈(전사활성조절 도메인, 후성유전적 변화인자, 형광 단백질 등)을 CRISPR-Cas9시스템에 결합시키면 새로운 기능을 나타내는 도구로 재탄생할 수 있다. 이 중 최근에 가장 주목할만한 기술로 탈아미노효소(deaminase)를 nCas9(nickase Cas9) 단백질에 결합하여 DNA 상에 존재하는 시토신(cytosine)을 티민(thymine)으로 편집하거나, 혹은 아데닌(adenine)을 구아닌(guanine)으로 편집할 수 있는 염기교정 유전자가위(CRISPR base editor)를 꼽을 수 있다. 기존의 유전자가위와 달리 염기교정 유전자가위는 DNA의 단일 나선만 절단하기 때문에 비상동말단연결(NHEJ, Non-Homologous End Joining)에 의한 삽입/결실 (indel) 변이가 최소화되고 고효율로 단일염기를 편집할 수 있는 획기적인 기술이다. 이에 본 기고에서는 염기교정 유전자가위의 개발 및 활용에 관한 최신 연구 동향을 소개하고자 한다.
그림 1. CRISPR-Cas9 시스템의 다양한 활용. CRISPR-Cas9 시스템은 유전자 정정gene editing) 뿐만 아니라 유전자 발현 조절(gene regulation), 후성유전체 편집(epigenome editing), 염색질 이미징(chromatin imaging), 유전자 염기교정(base editing), RNA 절단(RNA targeting), 염색질 위상 변화(chromatin topology)와 같은 다양한 용도로 활용되고 있다[1].
2. 본론
2.1 시토신 염기교정 유전자가위(Cytosine Base Editor, CBE)의 개발
염기교정 유전자가위는 하버드 대학의 David R. Liu 연구진에서 2016년도 <Nature>에 처음 보고하면서 알려지게 되었다[2]. 논문에서 보고한 시토신 염기교정 유전자가위는 DNA 염기인 시토신을 탈아미노화하여 우라실(uracil)로 변환할 수 있기 때문에 DNA가 복제되면서 우라실이 티민으로 편집되게 된다(그림2). 이때 사용한 시티딘 탈아미노효소는 쥐에서 유래한 APOBEC1 단백질로서 APOBEC1 단백질은 RNA에 결합할 수 있는 A1CF 단백질과 복합체를 형성하여 apolipoprotein B의 mRNA에 존재하는 시토신을 우라실로(uracil) 변환한다. David R. Liu 연구진은 APOBEC1단백질이 mRNA 뿐만 아니라 단일가닥 DNA의 시토신도 우라실로 변환할 수 있다는 점과 dCas9이 목표 서열을 인식할 때 sgRNA가 결합하지 않은 반대편에 시티딘 탈아미노효소가 작동할 수 있는 단일가닥 DNA가 생성되는 점을 착안하여 dCas9의 N 말단에 APOBEC1 단백질을 융합한 1세대 염기교정 유전자가위(BE1)를 제작하였다(그림 2). 그러나, 제작된 1세대 염기교정 유전자가위는 세포 외(in vitro)에서는 높은 염기교정 효율을 보인 반면 세포 내에서는 다소 낮은 활성을 나타내었다. 이에 연구진은 교정 효율을 올리고자 두 가지 아이디어를 고안하였다. 우선 1세대 염기교정 유전자가위의 C 말단에 Bacillus subtilis bacteriophage PBS1에서 유래된 우라실 DNA 글리코실레이즈 저해 단백질(uracil DNA glycosylase inhibitor, UGI)을 융합하여 세포의 염기절단복구(BER, base excision repair) 기능을 억제하였고, 이를 통해 교정효율이 향상된 2세대 염기교정 유전자가위(BE2)를 개발하였다. 다음으로 2세대 염기교정 유전자가위의 dCas9 부분을 nCas9으로 대체하여 sgRNA에 결합하는 단일가닥 DNA를 절단하였고, 이렇게 절단된 DNA 가닥이 복구되면서 티민으로 교정되는 효율이 증가된 3세대 염기교정 유전자가위(BE3)를 개발하였다. 3세대 염기교정 유전자가위는 인간세포에서 최대 75%까지 단일염기 교정 효율을 나타냈으며 삽입/결실 변이는 1% 미만으로 발생하는 우수한 효과를 보여주었고 추후 개발되는 다양한 시티딘 염기교정 유전자가위의 프로토타입으로 자리 잡았다.
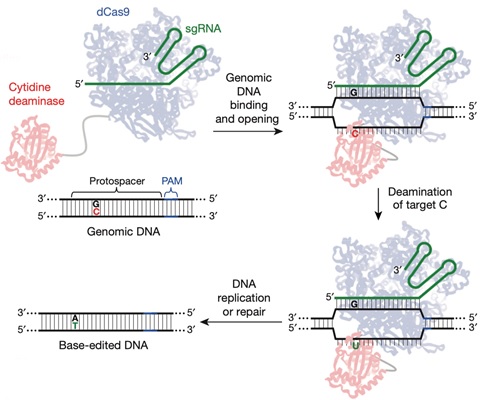
그림 2. 시토신 염기교정 유전자가위에 의한 단일염기 편집. 시토신 염기교정 유전자가위가 목표 서열에 결합하면 R-loop을 형성한다. 이때 생성된 단일가닥 DNA에 존재하는 시토신이 시티딘 탈인산화효소에 의해 우라실로 변환된다. 변환된 우라실은 DNA 복제과정 중에 아데닌과 염기쌍을 형성하면서 티민으로 염기서열이 편집된다[2].
2.2 아데닌 염기교정 유전자가위(Adenine Base Editor, ABE)의 개발
유전자 변이에 의한 질병들 중 약 58%는 단일염기 돌연변이에 의해 발생하고 단일염기 돌연변이 중 47% 정도는 C•G에서 T•A 로 바뀌는 염기전이(transition) 돌연변이에 의해 발생한다[3]. 이는 수많은 유전질환을 치료하기 위해서는 아데노신을 구아노신으로 편집할 수 있는 신규 염기교정 유전자가위의 필요성을 야기하였고, David R. Liu 연구진은 시토신 염기교정 유전자가위 논문을 발표한 이듬해인 2017년도에 아데닌 염기교정 유전자가위에 대한 연구결과를 <Nature>에 보고하였다(그림3)[4]. 이 논문의 핵심은 BE3에서 사용된 시티딘 탈아미노효소 대신에 단일가닥 DNA에 존재하는 아데닌을 탈아미노화 할 수 있는 효소를 찾는 일이었다. 그러나 자연계에서 단일가닥 DNA에 작동하는 아데노신 탈아미노효소(adenosine deaminase)가 존재하지 않기 때문에 연구진은 tRNA의 아데노신을 탈아미노화하여 이노신(inosine)으로 변환하는 대장균 유래의 TadA를 nCas9의 N 말단에 융합하였다. 그러나 불행히도 제작된 아데닌 염기교정 유전자가위는 동물세포에서 단일염기를 편집하지 못하였다. 이 문제를 해결하고자 연구진은 TadA 단백질에 다양한 변이를 도입한 후 고활성 염기교정 유전자가위를 선별하는 유도진화(directed evolution) 실험을 대장균에서 반복적으로 수행하였다. 또한 TadA 단백질이 동종이량체(homodimer)를 형성한다는 점을 착안하여 염기교정 유전자가위의 N 말단에 TadA 단백질을 추가로 융합하여 이량체 형성을 유도하는 전략을 도입하였다. 이렇듯 전략적인 개량을 통해 최종적으로 개발된 아데닌 염기교정 유전자가위인 ABE7.10은 고효율(약 50%)로 아데노신 주변 염기서열에 상관없이 단일염기를 편집할 수 있는 능력을 나타내었다.
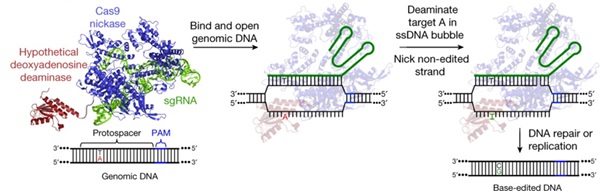
그림3. 아데닌 염기교정 유전자가위에 의한 단일염기 편집. 아데닌 염기교정 유전자가위는 디옥시아데노신 탈아미노효소(개량된 TadA 단백질)와 nCas9 융합단백질로 구성되어 있다. 염기교정 유전자가위가 목표 서열에 결합하면서 생성된 단일가닥 DNA에 존재하는 아데노신이 개량된 TadA 단백질에 의해 탈인산화하여 이노신으로 변환된다. 이후 DNA 복제 과정 중에 이노신은 구아노신으로 염기서열이 편집된다[4].
2.3 진보된 염기교정 유전자가위의 개발
현재 David R. Liu 연구진에서 보고한 CBE 혹은ABE를 기반으로 염기교정 유전자가위의 단점을 보완하면서 다양한 효용성을 갖는 신규 염기교정 유전자가위가 급속도로 개발되고 있다. 그 중 본 기고에서는 DNA 혹은 RNA의 비특이적 교정(off-target editing)을 줄이거나 Cas9 단백질을 개량하여 다양한 목표 서열에 결합할 수 있도록 개선한 연구 내용을 소개하도록 하겠다. 첫번째로, 염기교정 유전자가위는 3.2Gbp나 되는 방대한 인간 유전체에서 목표 서열만을 편집할 수 있어야 되기 때문에 서열 특이성(specificity)이 매우 높아야 한다. 이 특이성을 향상시키기 위하여 염기교정 유전자가위 구성 단백질을 개량하는 실험들이 여러 연구진들에 의해 수행되었다. 일례로, Cas9 단백질 자체에 변이를 도입하여 특이성이 향상된 HF-Cas9(high-fidelity Cas9) 혹은 Sniper-Cas9을 BE3에 사용한 전략이 시도되었고, 이때 진화된 염기교정 유전자가위는 비특이적 교정이 최소화되면서 목표 서열에 대한 교정 효율은 높은 결과를 나타내었다[5, 6]. 또한 염기교정 유전자가위의 경우 sgRNA가 결합하면서 생긴 단일가닥 DNA의 특정 범위에 존재하는 여러 개의 시토신을 동시에 탈아미노화 할 수 있는 편집 윈도우(editing window)가 존재하는데, 이는 목표 시토신이 아닌 주변의 시토신도 동시에 교정하는 결과를 초래한다. 이를 줄이고자, APOBEC1 단백질에 합리적 설계(rational design)를 통한 변이를 도입함으로써 편집 윈도우를 5bp에서 1-2bp로 축소시킨 염기교정 유전자가위도 개발되었다[7]. 두번째로, 염기교정 유전자가위를 다양한 유전자에 적용하기 위해서는 높은 특이성을 가지면서 다양한 목표 서열에 결합할 수 있도록 설계 범용성이 증가된 단백질 개량이 필요하다. 특히, 유전자가위에 사용되는 Streptococcus pyogenes 유래의 Cas9은 5′-NGG-3′의 PAM(protospacer adjacent motif) 서열을 인식하기 때문에 목표 서열을 설계하는 데 제약이 따른다. 이를 해결하고자, Cas9 단백질을 진화시켜 비 NGG(non-NGG) PAM 서열을 인식하는 염기교정 유전자 가위가 다양하게 개발되었다[7, 8]. 특히 David R. Liu 연구진이 개발한 xCas9 단백질의 경우 NG, GAA, GAT와 같이 넒은 범위의 PAM 서열을 인식하면서도 높은 특이성을 나타내기 때문에 다양한 염기서열을 편집하는 데 적절하다고 볼 수 있다[8]. 세번째로, 최근 연구 결과에 의하면 염기교정 유전자가위에 의하여 세포 내에서 발현되는 RNA의 염기 서열이 심각하게 변이되는 현상이 CBE와 ABE에서 공통적으로 발견되었다[9, 10, 11]. 이 현상은 염기교정 유전자가위에 존재하는 탈아미노효소의 활성에 의해 발생하기 때문에 연구진들은 탈아미노효소를 개량하여 RNA에 작동하는 비특이적 교정 현상을 제거하고자 하였다. 이를 위해 J. Keith Joung 연구진은APOBEC1에서 RNA의 시토신을 우라실로 변환하는데 관여하는 잔기에 변이를 도입하여 RNA 비특이적 교정을 최소화하면서 목표로 하는 DNA를 효율적으로 교정하는 진화된 시티딘 염기교정 유전자가위를 개발하였다[9]. 또한 동일 연구진에 의해 ABE 단백질도 개량되었는데 이때 사용한 방법은 TadA 단백질을 축소시켜 TadA에 의한 세포내 RNA 인식 능력을 줄이면서 동시에 TadA의 구조 기반 분석을 통하여 RNA 인식 및 활성에 관련이 있는 잔기들에 변이를 도입하는 것이었다[11]. 이런 통합적인 개량을 통해 RNA 비특이적 교정이 최소화된 ABE 개발에 성공하였다.
2.4 염기교정 유전자가위를 이용한 다양한 활용
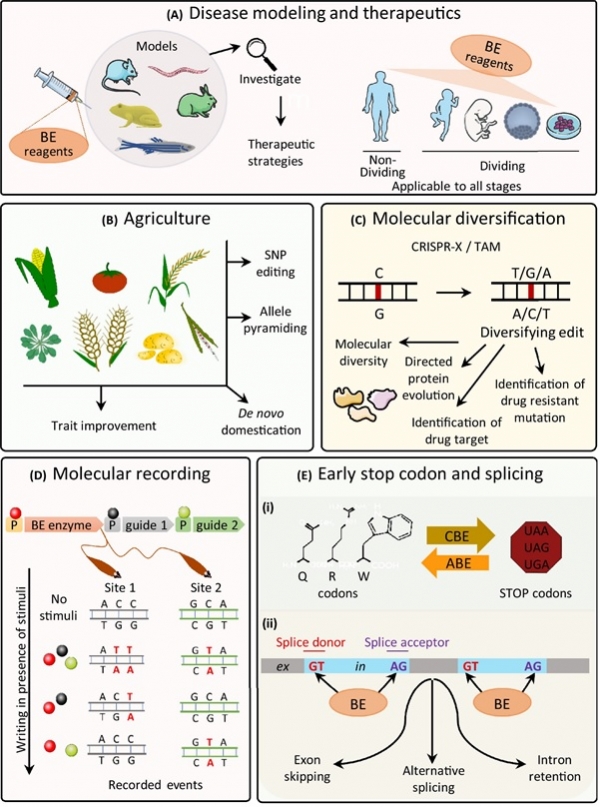
염기교정 유전자가위를 이용한 난치성 질환 치료의 가능성에 힘입어 2016년 CBE가 첫 개발된 후 짧은 시간 안에 염기교정 유전자가위 기술이 급속도로 발전하고 있다. 이를 반영하듯 2018년 크리스퍼 연구의 권위자인 Feng Zhang, David R. Liu, J. Keith Joung 박사들에 의해 설립된 염기교정 유전자가위를 기반으로 한 빔 테라퓨틱스(Beam Therapeutics) 회사가 작년에 이어 올해에도 유전자 교정을 이끄는 10대 회사들 중 하나로 손꼽히고 있다[12]. 또한 놀랍게도 David R. Liu 연구진이 개발한 염기교정 유전자가위 관련 플라스미드 6000개가 1000명 이상의 과학자들에게 Addgene을 통해서 전 세계적으로 분양되었다[13]. 이런 열풍적인 현상을 보여준 염기교정 유전자가위는 의료, 농업, 합성생물학 등 다양한 분야에 적용되어 활용되고 있다(그림4)[14]. 그 중 가장 각광을 받고 있는 분야인 동물 모델 구축 및 유전자 치료제 개발에 관한 연구 동향을 소개하도록 하겠다. 염기교정 유전자가위는 높은 특이성을 가지고 단일염기를 편집하기 때문에 질환 동물 모델 구축 및 단일염기변이(SNP, single nucleotide polymorphism)의 교정을 통한 선천적 유전질환 치료에 활용이 가능하다. 일례로 Hoxd13 유전자에 Q321R 변이를 유도할 수 있는 ABE mRNA와 sgRNA를 배아에 주입하여 생쥐에서 합지증(syndactyly)이 발병한 동물 모델구축에 성공하였다[15]. 유사한 방법으로 기초과학연구원 김진수 박사 연구진은 생쥐 배아에 색소 형성 유전자인 Tyr에 H420R 변이를 생성하는 염기교정 유전자가위를 주입하여 알비노 형질을 나타내는 생쥐를 제작하였고, 더 나아가 듀시엔형 근이영양증(Duchenne muscular dystrophy)을 앓고 있는 성체 생쥐의 근육세포에 AAV(adeno-associated virus)를 이용한 염기교정 유전자가위 전달을 통해 성공적으로 Dmd 유전자의 종결코돈을 글루타민 코돈으로 교정하였다[16]. 한편, Gerald Schwank 연구진은 성체 생쥐의 페닐케톤뇨증(phenylketonuria)을 치료하기 위하여 염기교정 유전자가위를 AAV를 사용하여 전달하였고 유전자 치료를 받은 생쥐는 phenylalanine hydroxylase 효소의 활성이 증가하여 혈액 내 phenylalanine 농도, 체중 증가 등과 같은 병증 완화 효과를 보여주었다[17]. 이렇듯 염기교정 유전자가위가 개발된 2016년 이후 짧은 기간 안에 전 세계 연구진들에 의해 질환 동물 모델의 구축 성공 사례와 더불어 유전자 치료제로서의 발전 가능성을 보여주는 보고들이 잇따르고 있다.
그림4. 염기교정 유전자가위의 응용. (A) 염기교정 유전자가위는 다양한 동물 모델 구축에 활용되어 치료제 개발 연구를 가속화 시킬 뿐만 아니라 모든 발생 단계의 인간 세포에 적용되는 유전자 교정 치료제로 사용될 수 있다. (B) 농업 분야에서는 염기교정 유전자가위를 이용하여 제초제, 질병, 환경 스트레스에 대한 내성 및 식물의 생산성을 증대시키는데 활용되어 농업생산성을 증대 시킬 수 있다. (C) 또한 염기교정 유전자 가위는 유전자에 다양한 돌연변이를 도입할 수 있기 때문에 단백질의 활성을 개량하거나 약의 표적 부위 혹은 내성에 관여하는 잔기를 추적하는 연구에 도움을 준다. (D) 만일, 염기교정 유전자가위의 sgRNA를 세포 외부 및 내부의 특정 신호(화합물, 빛)에 반응하는 프로모터와 연결하면 자극이 존재할 때 DNA에 관련 정보를 기록할 수 있는 장치로도 활용이 가능하다. (E) 마지막으로 염기교정 유전자가위에 의해 단백질에 종결코돈을 생성하여 단백질 번역을 중단시키거나 반대로 종결코돈을 제거하여 단백질 번역을 재개할 수 있고 RNA splicing에 관여하는 서열을 변화시켜 다양한 형태의 mRNA를 생성할 수 있다[14].
3. 결론
1958년 Francis Crick은 "DNA는 RNA를 만들고 RNA는 단백질을 만든다"라는 분자생물학의 중심원리(Central Dogma)를 제안하였다. 그 후, 인류는 생명과학의 혁명을 불러일으킨 크리스퍼 유전자가위를 통해 생명체의 표현형을 결정하는 유전정보인 핵산을 자유자재로 교정할 수 있게 되었다. 더불어 단일염기를 교정할 수 있는 염기교정 유전자가위의 탄생과 진화는 정교하게 유전자를 교정하여 난치성 유전질환을 안전하게 치료할 수 있는 새로운 방법으로 자리잡고 있다. 또한, 염기교정 유전자가위는 외래 DNA를 사용하지 않고 생명체의 유전체를 자연변이와 구분할 수 없도록 편집하기 때문에 유전자 변형 생물(LMO) 문제에 대한 해결책이 될 수 있고, 생명체의 특정 유전자에 다양한 돌연변이를 축적시켜 유용한 형질을 나타내도록 진화를 가속화할 수 있다. 이렇듯 짧은 기간 안에 염기교정 유전자가위는 빠르게 발전하였고 활용 범위도 다양하게 확장되고 있다. 앞으로 염기교정 유전자가위 기술이 어디까지 발전할지 기대가 된다.
참고 문헌
1. Adli M: The CRISPR tool kit for genome editing and beyond. Nature Communications 2018, 9:1911.
2. Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR: Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 2016, 533:420-424.
3. Rees HA, Liu DR: Base editing: precision chemistry on the genome and transcriptome of living cells. Nature Reviews Genetics 2018, 19: 770-788.
4. Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR: Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 2017, 551:464-471.
5. Rees HA, Komor AC, Yeh WH, Caetano-Lopes J, Warman M, Edge ASB, Liu DR: Improving the DNA specificity and applicability of base editing through protein engineering and protein delivery. Nature Communications 2017, 8:15790.
6. Lee JK, Jeong E, Lee J, Jung M, Shin E, Kim YH, Lee K, Jung I, Kim D, Kim S, Kim JS: Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications 2018, 9:3048.
7. Kim YB, Komor AC, Levy JM, Packer MS, Zhao KT, Liu DR: Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions. Nature Biotechnology 2017, 35:371-376.
8. Hu JH, Miller SM, Geurts MH, Tang W, Chen L, Sun N, Zeina CM, Gao X, Rees HA, Lin Z, Liu DR: Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature 2018, 556: 57-63.
9. Grünewald J, Zhou R, Garcia SP, Iyer S, Lareau CA, Aryee MJ, Joung JK: Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature 2019, 569: 433-437.
10. Zhou C, Sun Y, Yan R, Liu Y, Zuo E, Gu C, Han L, Wei Y, Hu X, Zeng R, Li Y, Zhou H, Guo F, Yang H: Off-target RNA mutation induced by DNA base editing and its elimination by mutagenesis. Nature 2019, 571: 275-278.
11. Grünewald J, Zhou R, Iyer S, Lareau CA, Garcia SP, Aryee MJ, Joung JK: CRISPR DNA base editors with reduced RNA off-target and self-editing activities. Nature Biotechnology 2019, 37:1041-1048.
12. Alex P: Top 10 companies leveraging gene editing in 2019. Genetic Engineering & Biotechnology News (2019.08.12).
13. Kevin D: All about that base editing. Genetic Engineering & Biotechnology News (2019.05.01).
14. Molla KA, Yang Y: CRISPR/Cas-Mediated Base Editing: Technical Considerations and Practical Applications. Trends in Biotechnology 2019, DOI: 10.1016/j.tibtech.2019.03.008.
15. Liu Z, Lu Z, Yang G, Huang S, Li G, Feng S, Liu Y, Li J, Yu W, Zhang Y, Chen J, Sun Q, Huang X: Efficient generation of mouse models of human diseases via ABE- and BE-mediated base editing. Nature Communications 2018, 9:2338.
16. Ryu SM, Koo T, Kim K, Lim K, Baek G, Kim ST, Kim HS, Kim DE, Lee H, Chung E, Kim JS: Adenine base editing in mouse embryos and an adult mouse model of Duchenne muscular dystrophy. Nature Biotechnology 2018, 36:536-539.
17. Villiger L, Grisch-Chan HM, Lindsay H, Ringnalda F, Pogliano CB, Allegri G, Fingerhut R, Häberle J, Matos J, Robinson MD, Thöny B, Schwank G: Treatment of a metabolic liver disease by in vivo genome base editing in adult mice. Nature Medicine 2018, 24: 1519-1525.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update