| 생명공학 인공지능에 대한 기대와 고려사항 | |
|---|---|
| Date 2020-07-29 17:22:01 |
|

교수
KAIST 생명화학공학과
ehukim@kaist.ac.kr
생명공학 속의 AI
바야흐로 인공지능(AI)의 시대이다. 모든 과학기술 분야에서는 앞다투어 AI를 외치고 있다. 온라인에는 AI와 관련된 뉴스와 교육자료가 넘쳐나고 있다. 우리 사회가 AI에 관심을 가지게 된 결정적인 사건으로 많은 사람들이 영국 DeepMind 사의 AlphaGo와 이세돌 전 프로바둑 기사와의 대결을 꼽을 것이다. 대학원생 시절부터 컴퓨터 관련 분야인 시스템 생물학 연구를 수행한 필자는 AlphaGo의 등장 이후로 시스템 생물학에 대한 관심도 덩달아 높아졌음을 느낄 수 있었다.
우연히도 필자 연구실의 책꽂이에 BT News 2009년도 가을호 (2009 Vol. 16 No. 2)가 꽂혀 있는 것을 발견하였다 (그림 1). 벌써 10년도 더 지난 이 호의 기획특집은 시스템 생명공학(systems biotechnology)을 주제로 다루고 있었다. 비록 AI나 빅데이터라는 표현은 사용하고 있지는 않지만, 여전히 AI와 빅데이터 기반 생명공학 연구와 일맥상통하는 단어들이 눈에 띈다.
오믹스, 시스템 수준, 생물 네트워크 등이 대표적인 예가 되겠다. 매우 거시적이며, 정략적인 개념들이다.

그림 1. BT News 2009년도 가을호(2009 Vol. 16 No. 2)의 표지와 목차
그렇다. 시간이 흐르면서, 새로운 기술들이 많이 쏟아지고 있지만, 생명공학과 시스템 생물학이 추구하는 기본 철학에는 변함이 없다. 시스템 생물학은 많은 데이터로부터 중요한 패턴을 찾아 내고, 이를 기반으로 생명체의 특성과 행동을 거시적인 관점에서 알아보는 학문이다. 시간이 흐르면서 달라진 점은 데이터의 양과 종류가 나날이 빠른 속도로 늘어나고 있다는 것과, 방대해진 데이터를 더욱 효율적으로 처리하기 위한 새로운 기술들이 꾸준히 개발되었다는 것이다. AI 중에서도 특히 딥러닝 (deep learning)으로 대표되는 머신러닝 (machine learning)은 그 어느 때보다 많은 관심을 받고 있다. AI는 앞으로 생명공학 연구를 어떻게 바꿀 것인가?
거대해지고 빨라지고 있는 생명공학 연구
분석 기술의 발달로 생명공학 연구의 규모가 커지는 것은 쉽게 생각해 볼 수 있다. 10년 전에도 ‘게놈 수준’(genome-scale)이라는 표현은 흔히 사용되어 왔다. 한 세포 안의 모든 유전자들을 고려하기에 게놈 수준이라는 표현이 사용되어 왔지만, 이제는 약간 진부한 표현이 되어 버린 것 같다. 근래에는 여러 세포의 유전체를 전체적으로 분석하는 연구를 자주 볼 수 있게 되었다 [1,2]. 일례로 가장 잘 알려져 있는 박테리아인 대장균 (Escherichia coli)의 경우, 50여균주에 대한 게놈 수준의 대사 (metabolism) 연구가 2013년에 첫 선을 보였으며 [3], 그 이후로 박테리아 범유전체(pan-genome) 연구가 여럿 나오기 시작하였다. 올해 초에 발표된 38가지 암 종류에 해당하는 총 2658개 암 조직 샘플에 대한 유전체 분석 연구도 대표적인 예가 될 수 있겠다 [4]. 이 연구는 37개국 1300여명의 과학자들이 참여한 Pan-Cancer Analysis of Whole Genomes (PCAWG) 국제 컨소시엄이 수행하였으며, 이 대규모의 국제공동연구를 통해서 38종 암들의 유전자 돌연변이 특징을 자세히 분석하였다.
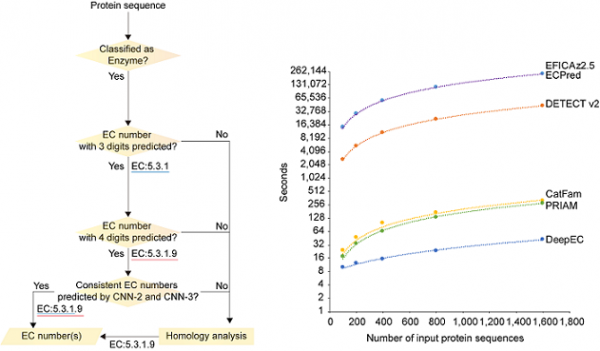
이렇게 데이터의 양이 빠른 속도로 커지는 것은 그만큼 빠른 분석을 가능하게 하는 새로운 방법론들이 개발되었다는 것을 의미한다. 필자가 속한 공동연구팀은 최근에 단백질서열의 EC 번호 (Enzyme Commission number)를 빠르고 정확하게 예측할 수 있는 딥러닝 기반 프로그램인 DeepEC를 개발하였다 [5] (그림 2). 효소의 기능을 나타내기 위한 방법 중 하나인 EC 번호는 효소가 매개하는 특정 생화학 반응을 4자리 숫자로 나타내는, 일련의 생화학 암호에 해당한다.게놈분석을 하게 되면, EC 번호도 예측을 하게 되는 데, NCBI RefSeq 등 게놈 데이터베이스에 등록된 GenBank 파일을 보면, 예측된 EC 번호의 개수가 매우 적고 정확성도 높은 편이 아니다. EC 번호의 중요성 때문에 지금껏 십여 개의 EC 번호 예측 프로그램들이 개발되었지만, DeepEC는 이전에 발표된 EC 번호 예측 프로그램들보다 성능과 속도, 용량 면에서 월등하다는 것이 검증되었다. DeepEC와 같은 프로그램들뿐만 아니라 샘플을 초고속으로 처리할 수 있는 로봇도 동원이 되면서, 오늘날의 생명공학 연구가 점점 더 거대해지고 빨라지고 있다는 것은 쉽게 생각해 볼 수 있다.
그림 2. DeepEC의 EC 번호 예측을 위한 단백질 서열정보 처리 과정 (왼쪽) 및 DeepEC와 다른 EC 번호 예측 프로그램들과의 정보처리 속도 비교 (오른쪽)
머신러닝은 양날의 검
생명공학 분야에서도 머신러닝에 관심을 가지는 연구원들을 많이 접하고 있다. 머신러닝은 잘 활용하면 득이 되지만, 제대로 활용하지 못할 경우 독이 될 수 있다. 이에 머신러닝 수행 시의 고려사항을 공유하고자 한다.
첫째, 머신러닝 기술 자체보다는 연구의 목적이 우선시되어야 한다. 생명공학 관련 연구목표를 달성하기 위해서 머신러닝 기술은 수단으로 보는 것이 대개의 경우에 해당한다. 연구의 목적에 따라서는 컴퓨터 프로그래밍은 필요하지만 머신러닝이 필요하지 않은 경우도 많다. 특히 생명공학 분야에서 사용되는 대부분의 머신러닝 기술은 전산학 분야에서 이미 치밀하게 검증된 기술이기에, 사용자 입장에서 연구목적 달성에 적합한 머신러닝 기술을 적시적소에 활용하는 것이 매우 중요하다. 단연 생명공학에 대한 충분한 지식 없이는 올바른 연구 목적을 가질 수 없으며, 실제로 잘못된 연구목표를 설정하고 머신러닝 모델을 만드는 경우를 목격한 적이 있다. 사실 연구 목적을 설정하는 것은 매우 어려운 단계에 해당한다.
둘째, 분석하고자 하는 데이터의 특성을 정확히 파악하고 있어야 한다. 이 두 번째 고려사항은 매우 중요해서 몇 가지 세부사항으로 더욱 나눌 수가 있다. 당연한 얘기처럼 들릴 수 있지만 사용하려는 데이터가 연구목적에 적합한 것이어야 한다. 그러나 주어진 데이터가 연구 목적에 적합한지 여부를 판단하는 것은 생각보다 쉽지 않다. 주어진 데이터를 가지고 실제 머신러닝 모델을 구축하고 나서야 깨닫게 되는 경우도 흔하다. 또 다른 어려운 점은 우리가 다루게 될 대부분의 데이터는 머신러닝 적용에 적합하도록 반드시 전처리 (data preprocessing)를 거쳐야 한다는 점이다. 데이터 전처리는 불완전한 형태의 데이터를 머신러닝 적용이 가능한 ‘깨끗한’ 형태의 데이터로 전환하는 것을 의미한다. 주어진 데이터에 일부 누락된 수치가 있을 수 있고, 표준화(standardization)가 필요할 경우 특정 생물 시스템의 특징에 맞게 수행해야 할 것이며, 분석 기기의 어떠한 특징 때문에 측정 오차가 생기는 지를 파악하고 있어야 한다. 이 문제는 생명공학 지식 없이 머신러닝 지식만으로는 해결하기 어려운 영역에 해당한다. 머신러닝 분야에서 흔히 접하는 ‘garbage in, garbage out’이라는 말은 데이터의 올바른 선택과 전처리의 중요성을 잘 나타내준다 (그림 3).
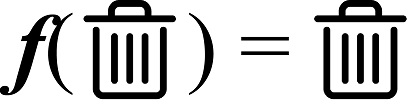
그림 3. 머신러닝 분야에서 흔히 사용되는 ‘garbage in, garbage out’ 개념
셋째, 머신러닝 예측결과가 처음 설정한 연구 목적에 대한 답변이 되는지 검증을 하여야 한다. 데이터 전처리만큼이나 어려우며, 많은 시간을 필요로 하는 단계이다. 역시나 전문적인 생명공학 지식을 필요로 하는 단계이며, 연구 목적과 관련된 세부 전문가들과의 많은 토론을 필요로 한다.
종합해 보면, 생명공학 연구에 AI를 적용한다는 것은, 로봇의 활용도 포함이 되지만 대부분의 경우는 오믹스 데이터 내지는 충분히 큰 분량의 데이터에 머신러닝을 활용하는 연구가 되겠다. 이 과정에서 활용하고자 하는 머신러닝에 대한 충분한 이해가 중요하지만, 그에 못지 않게 연구주제에 대한 깊은 생물학적 지식도 필요하다. 이러한 이유 때문에 시스템 생물학 연구를 하기 위해서는 생명공학과 컴퓨터 프로그래밍을 동시에 익혀야 하며, 생물 및 전산학 전문가와도 활발히 대화를 나누는 것이 연구의 진행에 매우 중요하다고 볼 수 있다.
배우기 쉬워진 시대
실험을 전문으로 하는 연구원이더라도, 조금이나마 컴퓨터와 더욱 친숙해졌으면 하는 바람을 가지고 있다. 다루게 되는 데이터가 시간이 지날수록 많아지다 보니, 컴퓨터를 제대로 활용할 수 있다는 것은 큰 장점으로 다가올 것이라는 생각 때문이다. 지금은 무엇이든 지 배우기 쉬워진 시대가 된 것 같다. 특히 컴퓨터와 관련된 것은 더더욱 그렇다. 머신러닝만 하더라도, YouTube, 블로그 등에 너무나도 유용한 무료 자료들이 많아서 마음만 먹으면 쉽게 접할 수 있다 (그림 4). 다만 실험을 전문으로 하는 연구원이 컴퓨터를 처음 접할 때, 어디서부터 시작해야 할 지 명확하지 않을 수 있겠다는 생각이 들었다. 즉 컴퓨터 환경 (computing environment)의 설정인데, 이를 조금이나마 수월하게 실행할 수 있도록 최근에 프로토콜 (protocol) 논문 형태로 관련 내용을 정리하였다 [6].
그림 4. YouTube에서 ‘machine learning’ 검색어를 통해 검색된 수 많은 교육자료들
생명공학 연구에서 컴퓨터의 중요성 때문에, 필자는 모든 수업에서 관련 소프트웨어 프로그램을 적극적으로 사용하고 있다. 일례로 2020년 봄학기에 대사공학 (metabolic engineering)을 가르치고 있는데, 미생물 대사를 연구할 때 필요한 대사 모델링 (metabolic modeling)을 주요 주제로 다루고 있다. 이를 위해서 프로그래밍 언어 중 하나인 Python으로 기본 연산부터 시작하여, 대사모델을 시뮬레이션할 수 있는 COBRApy를 가르치고 있다 [7]. 생물정보학 및 머신러닝 활용과 관련해서는, 작년에 ‘생명공학을 위한 빅데이터 분석 및 기계학습’ 수업을 개설하였다. 모든 분야가 그렇듯, 시스템 생물학 분야도 너무 빠르게 발전해서, 수업 시간에 새로운 소프트웨어들이나 컴퓨터 기술들을 적극적으로 소개하려고 한다. 이러한 교육 기회를 통해서, 생명공학 분야의 AI 전문가 양성에 최선을 다하고자 한다.
결론 및 전망
기술의 발전으로 생명공학 연구는 앞으로 더더욱 빨라지고 거대해 질 것이다. 이 과정에서 생명공학과 컴퓨터 프로그래밍을 동시에 익히는 것은 경쟁력 있는 연구를 수행함에 있어서 중요한 밑거름이 될 것이다. 다만 연구주제에 정말 빅데이터 또는 머신러닝이 필요한지, 주어진 데이터는 연구주제에 적합한지를 꼼꼼히 살펴보아야 한다. 컴퓨터 프로그래밍은 이제는 비교적 쉽게 접할 수 있는 영역이 되어 버렸다. 앞으로의 도전은 우리나라 고유의 바이오 데이터 확보와 관리 인프라 확충이 될 것이다. 우리나라 고유의 바이오 데이터에는 한국인 특이 유전체 정보일 수 있고, 우리나라 연구진이 개발한 산업용 미생물 균주일 수도 있다. 특히 일부 해외 연구소에서는 화합물 대량생산을 위해서 로봇을 구축하여 미생물 균주를 제작하는 데에 활용하고 있다. 이러한 도전은 한 연구실뿐만 아니라, 기관 내지는 국가 차원에서도 고민해야 할 문제가 되겠다. 현 시점에서 이러한 새로운 기술 장착은 향후 우리나라 생명공학 경쟁력에 지대한 영향을 끼칠 것으로 예상한다.
참고 문헌
[1] Kim Y, Gu C, Kim HU & Lee SY. Current status of pan-genome analysis for pathogenic bacteria. Current Opinion in Biotechnology 63, 54-62 (2020)
[2] Gu C, Kim GB, Kim WJ, Kim HU & Lee SY. Current status and applications of genome-scale metabolic models. Genome Biology 20, 121 (2019)
[3] Monk JM, Charusanti P, Aziz RK, Lerman JA, Premyodhin N, Orth JD, Feist AM & Palsson BØ. Genome-scale metabolic reconstructions of multiple Escherichia coli strains highlight strain-specific adaptations to nutritional environments. Proceedings of the National Academy of Sciences U S A (PNAS) 110:20338-43 (2013)
[4] The ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Nature 578:82-93 (2020)
[5] Ryu JY, Kim HU & Lee SY. Deep learning enables high-quality and high-throughput prediction of enzyme commission numbers. Proceedings of the National Academy of Sciences U S A (PNAS) 116, 13996-14001 (2019)
[6] Jeon J & Kim HU. Setup of a scientific computing environment for computational biology: Simulation of a genome-scale metabolic model of Escherichia coli as an example. Journal of Microbiology 58, 227-234 (2020)
[7] Ebrahim A, Lerman JA, Palsson BO & Hyduke DR. COBRApy: COnstraints-Based Reconstruction and Analysis for Python. BMC Systems Biology 7:74 (2013)
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update