| 예쁜꼬마선충 세균감염 모델 기반의 새로운 항감염제 스크리닝 플랫폼 | |
|---|---|
| Date 2020-07-30 16:19:04 |
|

석사과정 / 교수
이화여자대학교 약학대학 약학과
soominkim14@gmail.com / wooseong_kim@ewha.ac.kr
1. 서론
최근 여러 신문기사들에서 ‘슈퍼박테리아’라는 단어가 많이 언급되고 있다. 이는 현재 우리사회가 항생제 내성 위기에 직면하고 있다는 점과 무관하지 않다. 슈퍼박테리아는 여러 항생제에 대한 저항성을 지녀 현재의 항생제 화학요법으로는 효과적인 치료가 어려운 다제내성균 (multidrug-resistant bacteria)을 의미한다. 특히 항생제 내성문제는 오늘날 세계 보건을 위협하는 주요한 문제 중 하나로 대두되고 있다. 질병관리본부에서 실시한 연구에 따르면 국내의 경우 매년 9000여 명의 슈퍼박테리아에 의한 감염증 환자가 발생하며, 이 중 3900여 명의 사망 케이스가 보고되고 있다. 또한 영국의 한 연구에서는 항생제 내성균에 의한 사망자수가 2016년 약 70만 명에서 2050년 1000만 명까지 폭증할 것이며, 내성균 감염성 질환은 암을 넘어선 사망 원인 1위의 질병이 될 것이라고 예상하였다. 특히 Methicillin-resistant Staphylococcus aureus (MRSA) 는 1961년 영국에서 처음으로 발견된 후, 오늘날 전 세계적으로 가장 널리 퍼진 항생제 내성균이 되어, 병원 획득 감염 뿐만 아니라 지역사회 획득 감염도 일으키고 있다. 현재 S. aureus 감염증의 30 – 60%의 경우 MRSA로 인한 감염으로 보고되고 있으며, MRSA 감염증은 세균 감염질환 중 가장 높은 사망률을 나타내고 있다. 현재 MRSA감염에 최후의 항생제 (last resort antibiotics)로 vancomycin, linezolid, 그리고 daptomycin이 사용되고 있지만, 이들에 대한 내성을 보이는 S. aureus 변이균주도 발견되고 있는 추세다 [1]. 이에 과학자들은 현재 인류의 항생제 속도가 세균의 항생제 내성 발현 속도를 따라가지 못하고 있다는 사실을 인식하게 되었다 [2].
세균은 horizontal gene transfer나 de novo mutations과 같이 유전자 변이를 통해 항생제에 대한 내성을 획득한다. 하지만 유전적 변이 없이도 비성장 휴면상태 (nongrowing, dormant state)로 전환함으로써, 세균들은 현재 사용되고 있는 거의 모든 항생제에 대해 Tolerance (내성)을 보일 수 있다. 임상에서 흔히 사용되고 있는 항생제들은 대부분 병원균의 성장 및 분열 과정에서 일어나는 DNA의 전사 및 RNA, 단백질 또는 세포벽 합성의 생합성 과정들을 저해함으로써 항균작용을 나타내고 있다. 하지만 비성장 휴면상태인 박테리아의 경우 이러한 생합성기작들이 비활성 (inactive) 되어있기 때문에, 항생제가 항균작용을 할 수 없게 된다 [3]. 두 내성을 구분하기 위해, 유전적 변이에 의한 항생제 내성은 ‘Resistance’, 비성장 휴면상태에 의한 항생제 내성은 ‘Tolerance’로 정의하고 있다 [4]. 이와 같이 비성장 휴면상태로 인해 항생제 tolerance를 나타내는 세균을 ‘persister’라 정의한다. 특히, Persister는 기존의 항생제로는 효과적으로 제어할 수 없는 재발성 만성 감염질환의 원인으로 알려져 있다. 따라서 multidrug-resistant 및 -tolerant 세균에 대항하기 위한 새로운 항생제의 개발은 인류가 해결해야할 가장 시급한 문제 중으로 하나로 대두되고 있다.
2. 기존 항생제 스크리닝 방법의 한계 및 대안
현재 임상에서 자주 처방되는 항생제들은 1950-60년대에 발견되었으며, 주로 토양 미생물 추출물에서 항균작용을 나타내는 물질을 찾는 방법을 통해 발견되었다. streptomycin 개발 공로로 1952년에 노벨 생리의학상을 수상한 Selman A. Waksman의 이름을 따 이런 방식을 Waksman platform이라고 한다 [2]. 이후 여러 생명공학 기술의 발달로 항생제의 새로운 타겟들이 발굴되었으며, 이를 저해하는 물질들을 찾는 high-throughput screening (HTS)이 진행되었다. 하지만 이 스크리닝 방법들은 신항생제 개발에 성공적인 결과를 보여주지 못했다. ‘Hit’들의 대부분은 이미 발견된 항생물질이거나, 숙주 세포독성을 나타내거나, in vivo 활성을 나타내지 않았기 때문이다. 이러한 실패는 고전적 스크리닝 플랫폼이 더 이상 새로운 항생제를 찾는 데에는 유효하지 않다는 결론을 이끌어내었다 [2]. 따라서 고전적 스크리닝의 한계를 극복하기 위한 새로운 항생제 스크리닝 플랫폼 개발은 인류가 해결해야 할 시급한 과제가 되었다.
새로운 플랫폼 개발 시도 중에, Gerhard Domagk가 sulfanilamide계 항생제인 prontosil을 발견한 방법이 다시 주목을 받았다. Domagk는 배지에서 세균을 죽이는 방법이 아닌 감염된 생쥐를 치료하는 물질을 찾는 방식으로 실험을 진행하였다. 실제로 이러한 방법으로 in vitro 항균작용을 보이지 않지만 감염된 생쥐를 살리는 prontosil을 발견하게 되었다. Prontosil은 비슷한 시기에 발견된 페니실린과는 다르게 그 자체는 항균 활성이 없지만, 생체 내로 들어가 숙주의 효소에 의해 분해되면 대사물 중의 하나인 sulfanilamide가 항균작용을 하게 되는 것이다 [2]. Prontosil과 같이 그 자체로는 약리활성이 없지만, 분해된 대사물이 활성을 보이는 약제들을 Prodrug이라고 한다. Prodrug 타입의 항생물질은 in vitro 에서 세균의 생장을 저해하는 물질만을 hit으로 선별하는 고전적 스크리닝 방법으로는 발견할 수 없다. 이에 착안하여 병원균에 감염된 동물을 사용하여 숙주의 수명을 연장시키는 물질들을 선별해내는 스크리닝 방법이 제시되었다. 동물감염 모델을 기반으로 한 스크리닝 방법은 병원균에 직접적으로 작용하는 정균, 살균 작용을 나타내는 물질뿐만 아니라 병원균의 독성 인자에 작용하거나 숙주의 면역을 조절하는 등의 방법으로 감염을 치료하는 물질들과 같이 감염과정에서 병원균과 숙주 사이에서 일어나는 모든 과정 중 일부를 약물 작용기전으로 삼는 물질도 발견할 수 있다 [5]. 또한 숙주의 생존을 기준으로 hit을 판단하기 때문에, 스크리닝 과정에서 in vivo 효능이 확인되며, 동시에 물질의 세포독성을 확인할 수 있다는 장점이 있다. 생쥐는 윤리적 측면과 HTS에 적합하지 않기 때문에, 세균 감염 병인 연구의 모델 동물로 활발히 연구되어 온 초파리 (Drosophila melanogaster), 지브라 피쉬 (Danio rerio), 예쁜꼬마선충 (Caenorhabditis elegans)을 숙주감염 모델 기반의 새로운 스크리닝 방법에 적용하려는 시도들이 있었다 [6].
3. C. elegans 감염모델 기반의 항감염제 스크리닝 플랫폼
C. elegans는 길이 1 mm, 너비 50 μm 크기의 투명한 선충이다 (그림 1, [7]). 일반적으로 흙 속에 서식하며 박테리아를 주요 영양원으로 하고 있다. 실험실 환경에서는 agar 플레이트에서 비병원성 박테리아인 Escherichia coli OP50를 먹이로 공급하는 경우 평균 2-3주 동안의 수명을 보인다. 먹이가 E. coli 대신 병원균으로 대체되는 경우, C. elegans의 장내 병원균의 축적, 표피에 biofilm 형성, 그리고 현저한 수명 단축 (1.5-7일)과 같은 감염에 대한 증상이 명백하게 나타난다. 열 또는 항생제에 의해 사균 처리된 병원균이 먹이로 공급되는 경우에는 위와 같은 감염에 대한 증상이 나타나지 않는다. 이러한 사실은 C. elegans의 수명 단축 또는 감염에 대한 증상들이 단순히 먹이의 변화에 의한 것이 아니라 전형적인 감염증의 발병 과정에서 나타나는 것임을 유추할 수 있게 한다 [8].
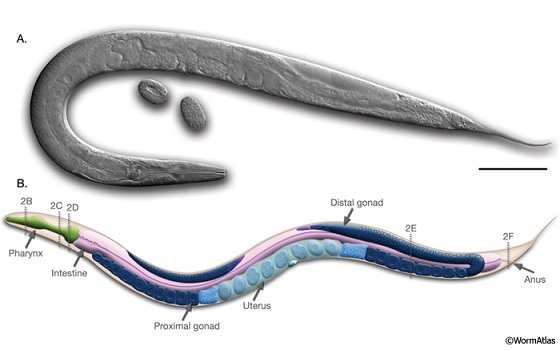
그림 1. C. elegans 성충과 그의 해부학적 모식도. 본 그림은 WormAtlas [7]에서 발췌함.
병원균의 독성 인자 (virulence factors)는 선충을 감염시키는 과정에서 중요한 요소로 작용한다.특히, 많은 수의 독성인자들은 C. elegans뿐만 아니라 사람과 같은 고등동물에서의 감염증을 유발하는 데에도 중요하게 작용한다고 알려져 있다 [9]. C. elegans는 후천 면역은 없지만 진화적으로 보존된 선천 면역 (evolutionarily highly conserved innate immunity)이 존재한다. 이러한 선천 면역 신호전달 경로는 사람에서도 매우 유사한 형태로 발견된다. C. elegans에는 사람의 p38 MAP kinase 경로, insulin/insulin-like 경로, transforming growth factor-β (TGF-β) 경로에 상응하는 PMK-1 경로 [10], DAF-2, DAF-16 경로 [11], 그리고 DBL-1 경로 [12]가 존재하며 이를 통하여 면역반응이 활성화된다. C. elegans의 면역반응은 병원균이 먹이의 형태로 침투하므로 장내 상피세포 (intestinal epithelial cells)에서 활발하게 일어난다. C. elegans와 사람이 동일한 병원균에 의하여 유사한 독성 인자의 작용기전으로 감염증이 발병하며 이에 대응하기 위하여 유사한 신호전달 경로로 면역반응을 활성화 시킨다는 사실에 기반하여 C. elegans의 감염증을 치료할 수 있는 물질은 사람의 감염증을 치료하는 데에도 효과적일 수 있다는 가정을 설립하였다. 실제로 현재 처방되고 있는 항생제들은 C. elegans를 감염으로부터 보호하며, 주요독성인자가 knock-out된 병원균은 생쥐와 C. elegans 모두에게서 감염을 유발시키지 않는다는 것이 보고 되었다.
이를 바탕으로, Moy et al.은 C. elegans 감염모델을 기반으로 한 항감염제 스크리닝 전략을 개발하였다 (그림 2, [13]). 기회감염성 병원균인 Enterococcus faecalis에 작용하는 항감염제를 발견하기 위하여, E. faecalis에 감염된 C. elegans를 사용하여 7136개의 합성 물질과 천연물 추출을 스크리닝 하였고, 이 중 16개의 합성 물질과 9개의 천연 추출물이 감염된 C. elegans를 치료하였다. 25개의 물질 중에서 12개의 물질은 항균작용 없이 감염된 C. elegans를 치료하였다. 이는 고전적 항생제 스크리닝 방법으로는 발견할 수 없는 Prodrug, anti-virulence agents, immunomodulatory agents에 속하는 항감염 물질들일 것이라고 유추되었다 [5]. 이후 Moy et al.은 선충 자동 분배기, 자동 현미경 및 이미지 분석을 이용하여 C. elegans 기반 HTS 방법을 자동화 시켰으며 이러한 기술의 발전으로 대단위의 항감염제 스크리닝이 가능해졌다 1[4].
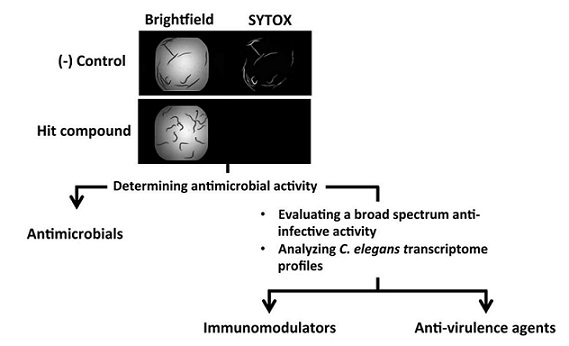
그림 2. C. elegans 감염모델 스크리닝 분석법. 본 그림은 참고문헌 [13]에서 발췌함. SYTOX는 죽은 C. elegans를 선택적으로 염색시킨다.
C. elegans 기반 HTS 기술은 중요한 병원균에 대한 항감염제를 발굴하기 위한 large-scale 스크리닝 프로젝트에 도입되었다. Moy et al.은 E. faecalis 감염에 대한 항감염 물질을 발견하기 위하여 ~37,000개의 물질을 스크리닝하였고 C. elegans를 보호하는 28개의 새로운 항감염 후보물질을 발굴하였다 [14]. Kim et al.은 Anti-MRSA agents를 발굴하기 위해 ~82,000개의 화합물을 스크리닝하여, 185개의 후보물질을 확보하였다 [15, 16]. Kirienko et al.은 그람음성 병원균인 Pseudomonas aeruginosa에 의한 감염증을 치료할 수 있는 항감염제를 찾기 위하여 86,441개의 물질을 스크리닝하였고 69개의 새로운 항감염 후보물질을 발견하였다 [17]. 이들뿐만 아니라 여러 과학자들에 의해 Acinetobacter baumannii, Klebsiella pneumoniae, Burkholderia pseudomallei, Burkolderia cenocepacia, Francisella tularensis, 그리고 Candida albicans과 같은 다양한 병원성 세균 및 곰팡이균에 의한 감염증을 치료하는 물질들을 찾기 위한 C. elegans 감염모델 기반의 스크리닝 방법이 개발되었다.
4. C. elegans 기반 HTS에서 발견된 항감염제
실제로 대단위 스크리닝 실험들에서 여러 항생물질들이 발견되었는데 이 물질들은 활성에 따라 크게 항균제 (antimicrobials), 항독성제 (anti-virulence agents), 그리고 면역 조절제 (immunomodulatory agents) 로 분류된다 (그림 3). C. elegans 감염모델 기반의 스크리닝 플랫폼으로 발굴된 주요한 신 항감염물질에 대해 소개해 보겠다.
기존에 임상에서 사용된 약물의 추가적인 약 활성을 찾고 이를 이용하여 다른 질환 치료제로 이용하는 것을 신약 재창출 (drug repurposing)이라고 한다. 신약 재창출의 경우, 약동학적 (pharmacokinetics/pharmacodynamics) 프로파일과 독성에 대한 연구가 실행되어 있으므로 de novo 신약 개발하는 것에 비해 시간과 비용 그리고 효율적인 측면에서 이점이 있다. Niclosamide와 oxyclozanide의 경우 (그림 3), 각각 사람과 동물에 사용하는 구충제로 개발되어 임상에서 사용되고 있는 약물들이다. 이 두 약물의 경우 C. elegans 기반 HTS에서 MRSA에 감염된 선충을 치료하는 것으로 확인되었다 [18]. Niclosamide와 oxyclozanide는 각각 MIC 0.0625-0.5 μg/mL, 0.125-0.5 μg/mL로 다제내성 S. aureus에 대한 강한 항균 활성을 가지고 있음이 확인되었다. 두 약물은 구조적으로 유사하지만 niclosamide는 정균제, oxyclozanide는 살균제로 서로 다른 항균 기작을 가지고 있다. Oxyclozanide는 병원균의 막 투과성을 증가시킴으로써 MRSA를 사멸시킨다 [18]. 반면 Niclosamide의 경우에는 병원균의 proton motive force를 감소시킴으로써 세균의 성장을 억제한다고 밝혀졌다 [19]. 두 약물의 경우, 구충제로 개발하기 위해 약동학 및 독성 프로파일의 연구가 이미 이루어져 있기 때문에, 다제내성균 감염증에 대한 항생제로서 빠르게 개발될 가능성 높다.
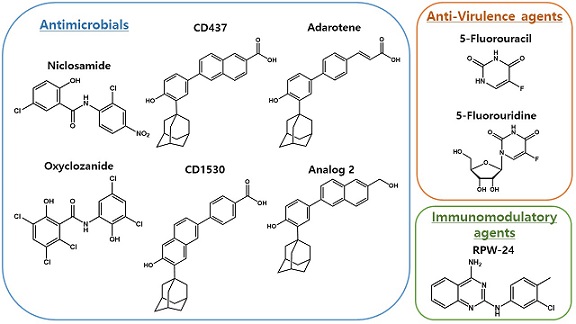
그림 3. C. elegans 기반 HTS에서 발견된 항감염 물질들
C. elegans 감염기반 스크리닝을 통해, 내성균인 MRSA 항균활성을 보이는 유사 비타민 A인 합성 레티노이드 (synthetic retinoids) 물질도 발굴되었다 [20]. 비타민 A 구조 유사체인 CD437, CD1530 그리고 adarotene은 각각 MIC 1, 1, 2 μg/mL로 last resort antibiotics인 vancomycin, linezolid, daptomycin과 동일한 항균 활성을 보이고 있다 (그림 3). 이들은 기존 항생제와 달리 생합성 기작 저해가 아닌, 병원균의 지질 이중층을 붕괴시킴으로써 항균작용을 나타내는 것으로 알려졌다. 하지만 이 합성 레티노이드들은 사람세포의 지질막에는 손상을 초래하지 않았다. 특히 현재 효과적인 항생제가 존재하지 않는 MRSA persisters에 대한 강한 항균작용을 보이는 것은 합성 레티노이드의 가장 큰 장점이다. 이 합성 레티노이드들은 aminoglycoside계 항생제 gentamicin과 상승 항균 작용 (synergistic antimicrobial activity)을 보이며, 함께 사용한 경우 생쥐를 이용한 만성 MRSA 감염모델에서 약효를 보였다. 또한 구조-활성 상관 (Structureactivity relationships)를 통해 CD437의 항균 활성과 세포독성 프로파일을 증진시킨 선도유사물질인 Analog 2도 새롭게 개발되었다 [20]. 이 결과는 새롭게 발굴된 레티노이드 계열 항생제가 MRSA persister가 일으키는 만성감염 질환의 치료제로서 개발될 높은 잠재성을 지니고 있음을 시사한다.
C. elegans를 이용한 HTS에 의해 발견된 항독성제에는 5-fluorouracil (5-FU)과 그것의 대사물질인 5-fluorouridine (FUR)이 있다 (그림 3) [21]. 현재 임상에서 대장암 치료제로 활발히 사용되는 대장암 치료제인 5-FU는 Kirienko et al.에 의해 P. aeurginosa에 감염된 C. elegans를 치료한다고 알려졌다 [21]. 또한 5-FU의 여러 유사체를 실험한 결과 5-FU의 대사물인 FUR도 P. aeruginosa에 대한 항감염효과가 있다고 밝혀졌다. 5-FU와 FUR의 경우 각각 0.375, 2.437 μm에서 P. aeruginosa에 감염된 C. elegans를 구하는 효과를 나타내었는데 두 물질 모두 이 농도에서 P. aeruginosa의 생장에 영향을 주지 않았다. P. aeruginosa는 숙주의 철을 chelate하는 pyoverdine이라는 독성인자를 분비한다. 5-FU와 FUR은 pyoverdine의 생합성을 억제함으로써 C. elegans를 보호한다는 것이 밝혀졌다 [21].
C. elegans를 이용하여 발견된 면역 조절제에는 RPW-24가 있다 (그림 3) [22]. 면역조절제는 병원균이 아닌 숙주의 면역 반응을 조절하기 때문에, 동물 감염 모델을 통해서만 용이하게 식별될 수 있다.R PW-24는 박테리아 성장을 억제하지 않으면서 그람양성균인 E. faecalis와 그람음성균인 P. aeruginosa 모두로부터 C. elegans를 보호한다고 밝혀졌다 [22]. RPW-24는 11 μM에서 선충의 P. aeruginosa 감염증을 치료하였지만 이 농도에서 P. aeruginosa의 생장에는 영향을 주지 않는다. Transcriptome analysis, epistasis pathway analysis, 그리고 RNAi screen을 수행함으로써 RPW-24는 pmk-1/p38 MAPK 선천면역신호 전달 경로와 전사인자 atf-7을 통하여 숙주의 면역반응을 자극한다는 것이 발견되었다. 이 연구는 면역조절제가 그람 양성균과 그람 음성균 모두에 효과적인 광범위 항감염제로 개발될 높은 잠재성을 가짐을 보여주었다 [22].
5. 결론
지난 10년간, C. elegans 감염모델 기반 스크리닝 플랫폼은 항생제뿐만 아니라, 항독성제, 면역 조절제도 발굴함으로써 항감염 후보물질 pool의 범위를 확장시켰다. 특히, 항독성제와 면역 조절제는 기존 항생제와 달리 병원균의 생장에 영향을 미치지 않으므로 내성 문제 해결을 위한 대안 약제로서 큰 가능성을 보이고 있다. 또한 in vivo 효능이 낮거나 독성이 높은 물질들의 스크리닝 단계에서의 배제를 통해 잠재성 높은 후보물질의 확보가 가능하게 되었다. 이는 고전적인 항생제 스크리닝 플랫폼의 한계를 넘어서는 장점들이며, 따라서 C. elegans 기반의 항감염제 스크리닝 방법은 패러다임 전환의 약물 발굴 플랫폼으로 현재의 항생제 내성 위기를 극복하기 위한 혁진적인 접근법이 될 것이다.
참고문헌
1. Stryjewski, M.E. and G.R. Corey, Methicillin-resistant Staphylococcus aureus: an evolving pathogen. Clinical Infectious Diseases, 2014. 58(suppl_1): p. S10-S19.
2. Lewis, K., Platforms for antibiotic discovery. Nature reviews Drug discovery, 2013. 12(5): p. 371-387.
3. Keren, I., et al., Persister cells and tolerance to antimicrobials. FEMS microbiology letters, 2004. 230(1): p. 13-18.
4. Balaban, N.Q., et al., Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology, 2019. 17(7): p. 441-448.
5. Moy, T.I., et al., Identification of novel antimicrobials using a live-animal infection model. Proceedings of the National Academy of Sciences, 2006. 103(27): p. 10414-10419.
6. Clatworthy, A.E., K.P. Romano, and D.T. Hung, Whole-organism phenotypic screening for anti-infectives promoting host health. Nat Chem Biol, 2018. 14(4): p. 331-341.
7. Altun, Z.F., Herndon, L.A., Wolkow, C.A., Crocker, C., Lints, R. and Hall, D.H. (ed.s), WormAtlas. 2002-2020. https://www.wormatlas.org/hermaphrodite/introduction/mainframe.htm
8. Garigan, D., et al., Genetic analysis of tissue aging in Caenorhabditis elegans: a role for heat-shock factor and bacterial proliferation. Genetics, 2002. 161(3): p. 1101-1112.
9. Sifri, C.D., J. Begun, and F.M. Ausubel, The worm has turned–microbial virulence modeled in Caenorhabditis elegans. Trends in microbiology, 2005. 13(3): p. 119-127.
10. Kim, D.H., et al., A conserved p38 MAP kinase pathway in Caenorhabditis elegans innate immunity. Science, 2002. 297(5581): p. 623-626.
11. Garsin, D.A., et al., Long-lived C. elegans daf-2 mutants are resistant to bacterial pathogens. Science, 2003. 300(5627): p. 1921-1921.
12. Mallo, G.V., et al., Inducible antibacterial defense system in C. elegans. Current Biology, 2002. 12(14): p. 1209-1214.
13. Kim, W., et al., An update on the use of C. elegans for preclinical drug discovery: screening and identifying anti-infective drugs. Expert Opin Drug Discov, 2017. 12(6): p. 625-633.
14. Moy, T.I., et al., High-throughput screen for novel antimicrobials using a whole animal infection model. ACS chemical biology, 2009. 4(7): p. 527-533.
15. Kim, W., et al., Identification of an antimicrobial agent effective against methicillin-resistant Staphylococcus aureus persisters using a fluorescence-based screening strategy. PLoS One, 2015. 10(6).
16. Rajamuthiah, R., et al., Whole animal automated platform for drug discovery against multi-drug resistant Staphylococcus aureus. PLoS One, 2014. 9(2): p. e89189.
17. Kirienko, Natalia V., et al., Pseudomonas aeruginosa Disrupts Caenorhabditis elegans Iron Homeostasis, Causing a Hypoxic Response and Death. Cell Host & Microbe, 2013. 13(4): p. 406-416.
18. Rajamuthiah, R., et al., Repurposing salicylanilide anthelmintic drugs to combat drug resistant Staphylococcus aureus. PLoS One, 2015. 10(4): p. e0124595.
19. Tharmalingam, N., et al., Repurposing the anthelmintic drug niclosamide to combat Helicobacter pylori. Sci Rep, 2018. 8(1): p. 3701.
20. Kim, W., et al., A new class of synthetic retinoid antibiotics effective against bacterial persisters. Nature, 2018. 556(7699): p. 103-107.
21. Kirienko, D.R., A.V. Revtovich, and N.V. Kirienko, A High-Content, Phenotypic Screen Identifies Fluorouridine as an Inhibitor of Pyoverdine Biosynthesis and Pseudomonas aeruginosa Virulence. mSphere, 2016. 1(4).
22. Pukkila-Worley, R., et al., Stimulation of host immune defenses by a small molecule protects C. elegans from bacterial infection. PLoS genetics, 2012. 8(6).
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update 
