| 신종 바이러스 감염병 팬데믹 대응을 위한 차세대 진단기술 | |
|---|---|
| Date 2021-04-21 15:55:51 |
|

선임연구원
대구경북첨단의료산업진흥재단 첨단의료기기개발지원센터
hwahui03@dgmif.re.kr
1. 서론
2019년 12월경에 중국 우한에서 원인 미상의 중증 폐렴이 다수 발생하는 일을 시작으로, 중국 지역뿐만 아니라 세계 전 지역으로 빠르게 신종 감염병인 코로나19 (COVID-19)가 확산하였다. 2020년 3월 세계보건기구 (World Health Organization, WHO)에서는 팬데믹 (Pandemic)을 선언하였으며 현재까지 약 1.21억 명의 확진자와 268만의 사망자가 발생하고 있는 유례없는 감염병을 전 세계적으로 겪고 있다 [1]. 또한, 우리나라는 2002년 사스 (SARS), 2009년 신종플루 (H1N1), 2015년 메르스 (MERS) 등을 겪으며 새로운 바이러스 발생에 따른 감염병 유행을 경험한 바 있으며 그에 따르는 사회적, 경제적 비용을 치렀다. 현재 전 세계는 1일 생활권으로 좁아진 환경을 비롯하여 증가된 인구와 높은 인구밀도, 사회적 빈곤, 사회구조의 변화, 공중보건 인프라의 결핍 등이 서로 영향을 주기 때문에 향후 신종 감염병의 위기의 빈도나 관리 비용이 증가할 것으로 예측되며 인류의 커다란 위험으로 급부상하고 있다 [2,3].
코로나19와 같이 새로운 신종 감염병이 발생한 초기에는 진단 및 치료제가 부재하기 때문에 기존 개발된 치료제 및 백신 등의 사용 가능 여부에 대한 임상이 요구되며, 새로운 치료제와 백신이 승인되기까지 적지 않은 시간이 필요하다. 때문에 감염병 발생 초기 단계에서 적극적인 진단과 추적을 통해 신종 감염병의 확산을 최대한 늦추는 것이 무엇보다 중요하다. 이에 이 글에서 감염병 대응을 위한, 특히 코로나19 대응과 관련된 진단기술에 대한 최신 기술 동향을 소개하고자 한다.
2. 본론
2.1. 팬데믹 바이러스 감염병 분자진단기술
코로나19와 같이 바이러스 원인으로 발생하는 감염병의 경우 바이러스의 핵산을 증폭하여 진단하는 분자진단 (Molecular diagnostics)과 면역반응을 이용하여 진단하는 면역진단 (Immunological diagnostics)으로 크게 나뉜다 (그림1).
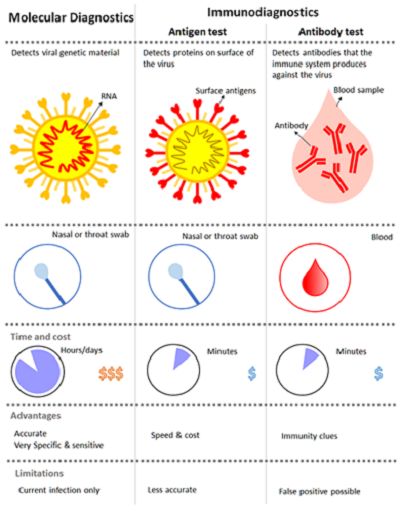
그림1. 분자 진단과 면역 진단의 비교.
분자진단은 병원체의 DNA, RNA 등의 핵산의 특이적 유전자 염기서열을 증폭하는 PCR 기반 진단법이 주를 이루고 있다. 특히, 코로나19 바이러스인 SARS-CoV-2와 같이 RNA 바이러스인 경우, real-time reverse-transcription PCR (RT-qPCR) 진단법을 많이 활용하고 있으며, 우리나라를 비롯한 선진국들은 real time-PCR(qPCR)을 주로 사용하나, 개발도상국의 경우 보담 저렴한 conventional PCR을 주로 사용한다 [4]. RT-qPCR은 정량이 가능하고 대량생산 및 비용적 측면에서 유리해 상업적 제품으로 많이 생산되며, 검사 소요시간은 2~6시간으로 비교적 짧고 정확도가 높다는 점이 특히, 무증상 감염자가 발생하는 코로나19 진단에서 Gold Standard로 사용되고 있다. 하지만, 편의성과 신속성이 면역진 단법에 비해 떨어지며 고가의 장비와 전문인력이 필요해 인프라가 부족한 나라에서는 적용이 제한된다.
2.2. 팬데믹 바이러스 감염병 면역진단기술
면역진단에는 바이러스의 항원을 직접 검출하는 항원 진단 (Antigen test)과 바이러스 감염으로 체내에 생성된 항체를 검출하는 항체 진단 (Antigody test)으로 나뉘며, 항원 진단은 비인두 혹은 구인두 채취, 항체 진단은 혈액을 채취하여 검사하며 샘플 채취 방식, 측정 방식, 정확도 및 민감도 등에 따라서 활용과 적용이 결정된다 (그림1).
면역진단법은 분자진단법과는 다르게 증폭 과정이 없어 신속하게 진단 가능하며 (~10분) 고가의 장비나 전문 인력이 필요 없어 현장검사 적용에 용이하다. 이에 POCT (Point-of-care test) 장비와 결합되거나 신속진단키트 (Rapid Diagnostics Kit) 등의 상용화된 제품들이 출시되어 있다. 하지만, 감염 초기이거나 항원, 항체의 양이 적을 때, 위양성 (False-positivie) 및 위음성 (False-negative)이 높아 부정확한 진단 가능성이 있으며 정확도는 보통 50~70%로 분자 진단에 비해 낮다 [7]. 이에 코로나19 바이러스 진단에서는 WHO에서 분자진단을 통한 확진을 권고하고 있으며 면역진단 키트는 요양병원 혹은 단체 생활을 하는 곳에서 많은 수의 대상자를 검사하여 1차 방어선의 역할로써 RT-qPCR의 보완재로 사용되는 편이다 [7].
2.3. 코로나19 대응을 위한 차세대 진단 기술
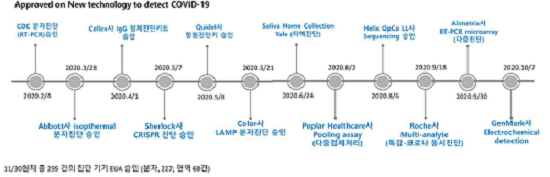
코로나19 팬데믹으로 많은 나라에서 감염자가 폭발적으로 증가하면서 진단제품에 대한 긴급승인이 시행되면서 분자 진단과 면역진단뿐만 아니라, 등온핵산증폭 (Isothermal amplification), 검체 채취에서부터 결과 판독까지 집에서 진단 가능한 home testing kit, CRISPR 유전자 가위 진단 등을 포함하는 다양한 진단 기술들의 시장 도입이 이루어지고 있다 (그림2) [5].
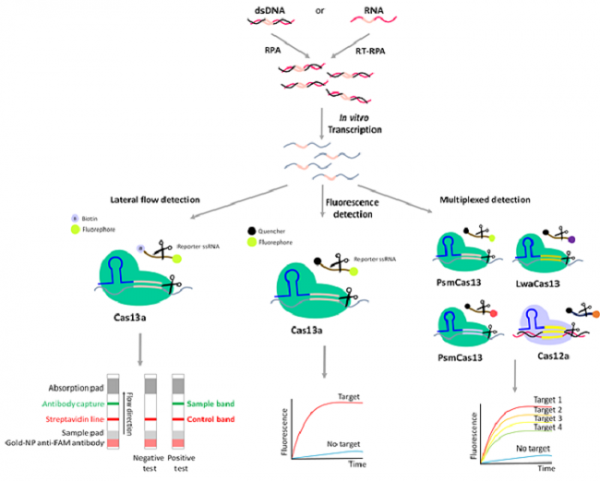
이 중에서도, CRISPR 유전자 가위를 이용한 진단기술은 2017년 MIT의 Feng Zhang 그룹에서 SHERLCOK (Specific High-Sensitivity Enzymatic Reporter UnLOCKing)이란 이름으로 CRISPR 유전자 가위를 이용한 진단법 개념을 Science지에서 처음 소개하며 [8] 시작되어 차세대 진단 유망 기술로 손꼽히고 있다. CRISPR 유전자 진단법의 기본적 원리는 CRISPR-Cas 시스템이 특정 유전자 서열을 인식하고 자르는 기본 작동에 부가적으로 주변에 존재하는 single strand 핵산을 비특이적으로 같이 절단하는 특성 (collateral cleavage activity)에 기반한다 (그림3) [9,10]. Cas-RNA complex가 traget DNA 혹은 RNA의 PAM (Protosppacer adjacent motif) 부근의 서열을 인식하고 특이적으로 절단하면서 주변에 존재하는 reporter DNA/RNA를 비특이적으로 절단하게 되면 reporter DNA의 qunecher와 fluorescence dye가 분리되어 형광 신호가 발생한다. 이 때, Cas enzyme의 종류에 따라서 인식 및 절단 가능한 핵산의 종류가 달라지는데, Cas12는 double strand DNA (dsDNA)를 target gene, ssDNA를 reporter DNA로, Cas14는 ssDNA를 target 및 reporter DNA로 적용 가능하며, Cas13은 RNA를 대상으로 가능하다 (그림3) [8-10]. Cas9의 경우 collateral cleavage activity는 없지만, nuclease-deactivation (dCas9) 하여 target gene에 특이적으로 binding 하게 하여 isothermal amplification 혹은 다른 labeling strategy를 같이 사용하여 진단에 적용하는 경우도 있다 [11-13]. 검출하고자 하는 대상의 Target 유전자의 종류에 따라 적절한 CRISPR/Cas 시스템을 선택하여 진단법을 개발할 수 있다.
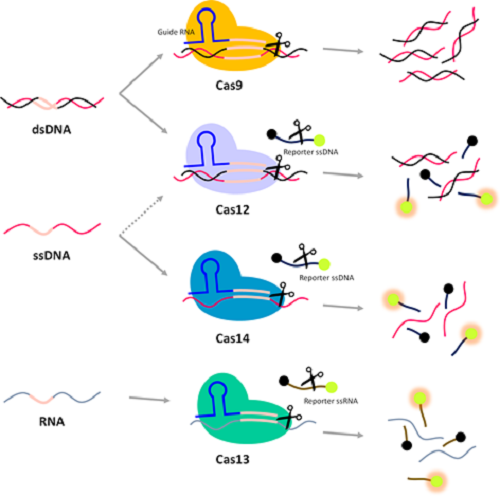
그림 3. CRISPR/Cas 진단 기본원리.
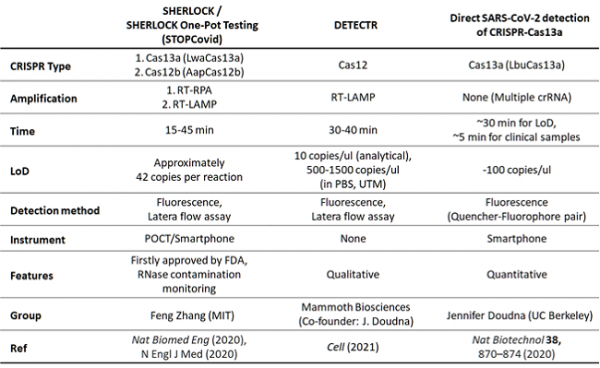
CRISPR 유전자 가위의 대표적 진단법 중 하나인 MIT Feng Zhang 그룹의 SHERLOCK은 DNA 혹은 RNA target을 RPA (reverse transcription Recombinase Polymerase Amplification) 혹은 RT-RPA로 각각 증폭시키고 T7 RNA polymerase로 증폭하는 단계와 CRISPR/Cas13a로 target RNA가 존재하는 경우, 주변 reporter RNA의 절단으로 생성되는 형광 신호로 검출하는 단계로 구성된다 (그림4) [8,13-17]. 이 진단법을 검증하기 위해 지카와 뎅기 바이러스 검출에 적용하여 특이적 검출이 가능하며 aM 수준의 민감도를 보임을 보고하였다. 또한, SHERLOCK은 간편한 신호 검출이 가능하도록 Lateral flow readout과 결합한 형태로 발전시켰다 [14-17]. 뿐만 아니라 2020년에는 SARS-Cov-2 검출에 대한 임상학적 검증도 하였는데, 형광 검출의 경우 100% 특이도, 100% 민감도를, Lateral flow readout의 경우 100% 특이도, 97% 민감도를 보임을 보고하였다 [18].
그림4. SHERLOCK CRISPR Diagnostics A. Complete SHERLOCK experimental workflow [17].
SHERLOCK은 증폭, 검출 2단계에 걸쳐서 진단이 이루어지는데, RNA 추출 과정, 샘플 핸들링 과정 등에서 교차 오염이 일어날 가능성이 있으며 또한, 현장 진단에서 사용 시에는 더 간편한 방법이 필요하다. 이를 위해 one-pot SHERLOCK (STOPCovid)를 새로 제안하였는데, 이는 target 유전자를 RT-LAMP (Loop-mediated isothermal amplification)로 증폭하고 CRISPR/Cas12b complex를 이용하여 형광 신호를 생성한다 [19]. 이 진단법은 15-45분 소요되며, SARS-CoV-2 patient 샘플을 이용한 테스트에서 98.5%의 특이도, 93.1%의 민감도를 보여주었다 [19].
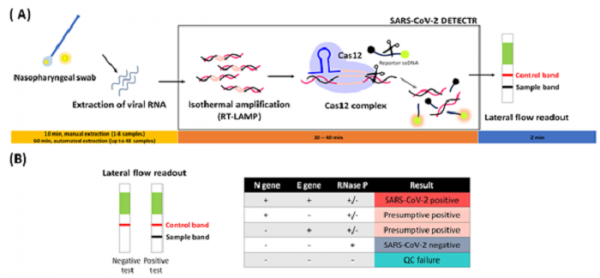
CRISPR/Cas system을 세계 최초로 보고한 UC Berkeley의 Jennifer Doudna 그룹도 2018년에 Cas12a가 collateral activity가 있음을 보고하였으며, 이를 이용한 진단법인 DETECTR를 제안하였다 [20]. Mammoth Bioscience사에서 이 기술의 상용화와 기술 개발을 이어가고 있는데, 2020년에 DETECTR를 이용한 SARS-CoV-2 진단에 대하여 Nature Biotechnology에 보고하였다 (그림5) [21]. 환자 샘플로부터 RNA를 추출한 뒤, RT-LAMP로 증폭을 거쳐 CRISPR/Cas12a가 target gene을 인식하면서 reporter DNA의 비특이적 절단이 일어나면서 형광 신호를 읽는다. DETECTR 또한 간편한 신호 검출을 위해 lateral flow readout을 함께 사용한다. 이 방법은 40분 이내로 가능해 빠른 진단이 가능하다는 장점이 있지만, LoD는 10 copies/ul로 CDC SARS-CoV-2 RT-qPCR이 1 copy/ul임에 비하면 민감도가 떨어지는 편이다 [21].
그림5. DETECTR CRISPR Diagnostics. (A) Schematic of SARS-CoV-2 DETECTR workflow (B) Lateral flow strip assay readout [21].
SHERLOCK이나 DETECTR 모두 isothermal amplification을 통해 target 유전자를 증폭했음에 비해, 2020년에 Jennifer Doudna 그룹에서 새로 제시한 Direct SARS-CoV-2 detection은 증폭 과정 없이 CRISPR/Cas13a를 이용하여 진단한다. 이는 기존 방법들과는 다르게 하나의 target 유전자 상에 여러 종류의 guide RNA가 인식하는 서열 종류수를 늘려 극복하였다 (그림6) [22]. 이 진단법 역시 SARS-CoV-2를 특이적으로 검출하며 LoD는 ~100 copies/ul 수준이다. 임상 샘플에 대해서는 5분 이내에 검출이 가능도록 진단 시간을 대폭 감소시켰으며, 휴대폰을 이용한 정량이 가능해 현장 진단에 적합하도록 개발하였다 [22].
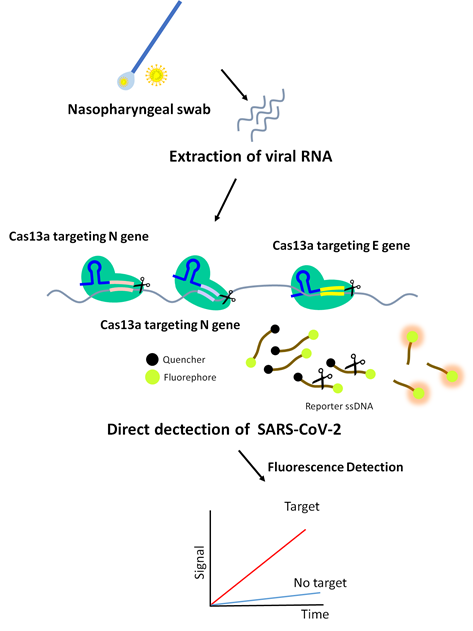
그림6. Direct SARS-CoV-2 detection of CRISPR-Cas13a [22].
CRISPR 유전자 가위 진단법은 qPCR에 비하면 비교적 간단한 장비로 빠른 시간 내에 검출할 수 있다는 장점이 있으며, lateral flow readout이나 POCT 장비 결합 등으로 정량할 수 있다는 가능성이 높아 현장 진단법으로 발전 가능성이 높다. 다만, 민감도를 위해 isothermal amplification 등의 증폭 과정 결합이 필요할 수 있다는 점과 PAM 서열이 존재해야 CRISPR 유전자 가위 진단법이 사용가능하다는 점이 진단법 개발에 제한이 될 수 있다.
표1. CRISPR 유전자 가위 진단법
3. 결론
앞서 서론에서 말했듯이 COVID-19 팬데믹을 전 세계적으로 겪으면서 신규 바이러스로 인한 세계적 유행에 대한 사회적, 경제적 피해뿐만 아니라 그로 인해 야기되는 공포와 불안을 우리는 겪고 있다. 바이러스는 끊임없이 돌연변이를 생성하기 때문에 오늘과 같은 팬데믹이 다시없으리라는 보장을 누구도 할 수 없다.
COVID-19 팬데믹을 겪으면서 진단법, 항체, 치료약 개발이 무엇보다 중요함을 알고 있다. 특히, 감염 초기에는 정확한 진단을 통해 감염 확산을 방지하는 것이 중요하므로 새로운 진단기술 혹은 기존 진단기술을 새로운 감염병에 맞게 발 빠르게 개발, 제품을 생산하는 것이 중요하다. 임상의들은 바이러스성 감염병일지라도 바이러스와 발병 특성에 따라 요구되는 진단 특성이 다르다고 말한다. MERS의 경우, 전파력은 낮은 대신 치사율이 높아, 높은 정확도 (100%)를 요구하며, 신종플루의 경우, 전파력은 높지만, 치사율은 낮아 빠른 검사법이 필요했으며, COVID-19의 경우 높은 전파력, 비교적 높은 치사율, 무증상 등 복합적이라 높은 특이도와 빠른 검사법을 요구한다고 한다. 신규 감염병 발생 시, 감염병 특성을 빠르게 파악하여 그에 요구되는 진단법으로 맞춤 개발하는 것 또한 필요할 것이다.
우리나라는 COVID-19 팬데믹에서 국내뿐만 아니라 국외 수출 확대 등을 통해 역량을 확인한 바 있지만, 아직 시약, 핵산추출키트, 장비 등에는 수입의존도가 높다. 또한, 차세대 진단기술 등에 대한 국산화 및 국내 독자 기술 개발 등에 힘써 신종 바이러스 감염병 출현 시에 신속하게 대응할 수 있도록 해야 한다.
참고문헌
1. Novel Coronavirus (COVID-19) Cases, Johns Hopkins University Center for Systems Science and Engineering (JHU CSSE),
https://github.com/CSSEGISandData/COVID-19.
2. Prepared in collaboration with the Boston Consulting Group(2017). Managing the Risk and Impact of Future Epidemics, Privacy
Policy & Terms of Service. Geneva: World Economic Forum [16-1-2016].
3. 현정희, 신종감염병 유행과 감염병 위기대응체계 구축, 한국건강증진개발원 Weekly Issue (2017), 21, 1-8.
4. 김주원, 여창민, 신종감염병 위기대응 기술(진단, 치료, 백신), KISTEP 기술동향브리프, 2020-05호, 1-33.
5. 이규선, 코로나19 대응을 위한 진단분야 연구동향, BioINpro, 2020, 82, 1-15.
6. 조선일보, https://biz.chosun.com/site/data/html_dir/2020/03/19/2020031900433.html
7. 데일리메디 , 코로나19 진단 , 유전자 증폭검사 v s 신속 면역항체검사, h t t p s: / /www.d a i l y m e d i.com/d e t a i l.
php?number=854752&thread=22r03
8. Gootenberg, Jonathan S., et al. "Nucleic acid detection with CRISPR-Cas13a/C2c2." Science 356.6336 (2017): 438-442.
9. Akkapol Suea-Ngam, Léonard Bezinge, Bogdan Mateescu, Philip D. Howes, Andrew J. deMello, and Daniel A. Richards, ACS
Sensors 2020 5 (9), 2701-2723.
10. Yi Li, Shiyuan Li, Jin Wang, Guozhen Liu, CRISPR/Cas Systems towards Next-Generation Biosensing, Trends in Biotechnology,
2019, 37(7), 730-743.
11. Mengqi Huang, Xiaoming Zhou, Huiying Wang, and Da Xing, Clustered Regularly Interspaced Short Palindromic Repeats/Cas9
Triggered Isothermal Amplification for Site-Specific Nucleic Acid Detection, Anal Chem 2018, 90, 3, 2193–2200.
12. Keith Pardee, Alexander A. Green, et. al, Rapid, Low-Cost Detection of Zika Virus Using Programmable Biomolecular
Components, Cell, 2016, 165(5), 1255-1266.
13. Jianyu Hu, Min Jiang, Rui Liu, and Yi Lv, Label-free CRISPR/Cas9 Assay for Site-Specific Nucleic Acid Detection, Anal Chem,
2019, 91, 10870-10878.
14. Gootenberg Jonathan, Abudayyeh Omar, et al., Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and
Csm6, Science, 2018, 360(6387), 439-444.
15. Freije et al., Programmable Inhibition and Detection of RNA Viruses Using Cas13, Molecular Cell, 2019, 76, 826–837
16. Myhrvold et al., Field-deployable viral diagnostics using CRISPR-Cas13, Science, 2018 360, 444–448.
17. Kellner, M. J., et al. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nature protocols, 2019, 14.10: 2986-3012.
18. Patchsung M., ea al, Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA, Nature Biomedical
Engineering, 2020, 4.12: 1140-1149.
19. Joung, Julia, et al. Detection of SARS-CoV-2 with SHERLOCK one-pot testing. New England Journal of Medicine, 2020, 383.15:
1492-1494.
20. CHEN, Janice S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science, 2018,
360.6387: 436-439.
21. BROUGHTON, James P., et al. CRISPR–Cas12-based detection of SARS-CoV-2. Nature biotechnology, 2020, 38.7: 870-874.
22. FOZOUNI, Parinaz, et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy.
Cell, 2021, 184.2: 323-333. e9.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update