| 시험관진화기술의 재발견: pH 활성화 DNA 나노기계의 제작 | |
|---|---|
| Date 2017-04-01 17:02:06 |
|

교수
포항공과대학교(POSTECH) 신소재공학과
seungsoo@postech.ac.kr
1. 새로운 약물전달기술로의 DNA 나노구조
세포들은 흔히 분자의 이동과 처리를 위해 국부적 pH 변화를 활용한다. 예를 들어, 세포 내부로 진입한 리간드(ligand)는 엔도솜(endosome)에 의해서 세포의 특정 부위로 운송되거나, 분해를 위해서 리소좀(lysosome)으로 운반되기도 한다[1]. 엔도솜과 리소좀의 pH는 대략 5.2, 4.5이고 이는 세포 외부의 pH가 7.4라는 것을 고려했을 때 강한 산성적 변화라고 할 수 있다. 따라서 특정 세포로 약물을 전달하는 방법으로써 이러한 pH 차이를 응용하는 연구들이 현재 활발히 진행되고 있는 가운데[2], 기능성 DNA 나노구조를 이용하려는 시도들이 최근 두드러지고 있다. DNA 나노구조의 경우, 기본적으로 안정한 구조를 갖을 뿐만 아니라 다양한 핵산 배열의 프로그래밍이 가능하고, 역배열(antisense)의 DNA나 siRNA와 같은 다른 기능성 핵산들을 쉽게 통합시킬 수 있는 독특한 장점들을 지니고 있다. 더 나아가, 이러한 DNA 나노구조는 pH변화에 민감하게 반응할 수 있는 잠재력을 보유하고 있기에, pH에 반응하여 프로그래밍된 기능들을 수행하는 DNA 나노구조들을 새로운 약물전달기술로 활용하고자 하는 연구들이 현재 점점 더 큰 관심을 받고 있다[3].
2. pH에 반응하는 DNA 나노구조의 연구동향
최근까지 대부분의 pH에 반응하는 DNA 나노구조는 pH에 즉각 반응한다고 알려진 모티프들을 중심으로 제작되어 왔다. 현재 pH 변화에 의해 응답하는 DNA의 범주로는 i-motif[4]와 삼중나선구조(triplex) [5]가 가장 활발하게 연구되고 있고, 이 밖에도, A-motif[6]를 활용하려는 연구들이 보고되고 있다.
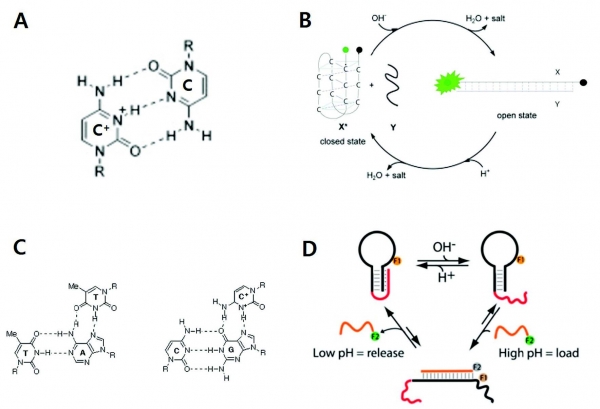
그림 1. 모티프 기반의 pH에 반응하는 DNA 나노구조. A. i-motif의 기본을 이루는 C-CH+ base pairing. B. i-motif를 기반으로 하여 pH에 반응해 목표 핵산 배열과 결합하는 DNA 나노구조(참고문헌 [8]에서 발췌 및 편집). C. 삼중나선구조를 이루는 TA-T와 CG-CH+ base pairing. D. 삼중나선구조를 이용하여 목표 핵산 배열을 선적/방출 가능한 DNA 나노구조(참고문헌 [14]에서 발췌).
특히, 많은 연구자들이 i-motif를 응용하여 pH에 따라 변하는 DNA 나노구조를 제작해오고 있다. 1993년 프랑스 연구진에 의해 처음 발견된 i-motif는 사이토신 사중나선-quadruplex)을 뜻하며, pH 6.5 이하의 C-rich DNA 서열에서 발견할 수 있다[1]. 그 구조를 좀 더 자세히 설명하면, 중성이나 염기성 환경에서의 C-C mispairing(그림 1A)은 pH 6.5 이하의 산성 환경에서 한 쪽 사이토신 염기의 질소 원자가 protonation되면서 평행한 C-CH+ 이중나선구조(duplex)를 형성하게 되고, 형성된 두 개의 C-CH+ 이중구조는 염기쌍 삽입을 통한 독특한 방식으로 안정한 사중나선 구조를 완성하게 된다[7]. i-motif는 pH 6.5에서 매우 급격한 구조 변화를 나타내고 반응이 가역적이며 매우 빠른 형성 시간(~100 ms)을 보이기에 현재 다양한 DNA 나노기계에 삽입되고 있다[8] (그림 1B). 대표적으로, 살아있는 세포의 엔도솜 내부의 pH 변화를 측정하기 위한 형광 센서[9], 특정 세포를 pH를 이용해 선택적으로 방출할 수 있는 전달체[10] 등으로 활용된 바 있다.
최근에, pH에 따라 이중구조와 삼중구조 모드로 가역적 변화하는 삼중나선구조 스위치(triplex switch)에 관한 흥미로운 연구가 발표된 바 있다[5]. Ricci와 동료연구자들은 전달하고자 하는 DNA 수하물의 선적과 방출을 pH에 따라 입체적으로 조절할 수 있는 삼중나선구조 모티프를 설계하였다. 우선, pH에 따라 구조를 변화시킬 수 있는 삼중나선구조의 경우 두 가지 종류를 설계할 수 있다. 먼저 T-A Watson-Crick pairing의 아데닌에 추가적으로 티민이 수소결합을 형성하여 삼중나선이 되는 것이 있고, 다음으로 C-G Watson-Crick pairing의 구아닌 염기에 protonation된 CH+가 수소결합을 형성하여 삼중나선을 형성하는 것이 있으며, 이러한 염기 간의 상호 작용은 Hoogsteen intera ction을 따른다(그림 1C). CG-CH+ 삼중나선의 경우 사이토신 염기의 protonation에 의해 pH 6.5 이하의 환경에서만 형성되고, TA-T 삼중나선의 경우 pH 10 이상의 염기성 환경이 되면 티민 염기가 deprotonation되면서 결합이 끊어짐에 따라pH에 의한 이중/삼중구조의 변화를 얻게 된다[11] (그림 1D). 이러한 원리를 이용해, pH의 변화에 따라 DNA nanotube를 조립할 수 있는 pH 회로[12], pH 변화에 따라 sol-gel transition을 보여주는 하이드로젤[13], 코카인과 같은 특정한 분자를 원하는 pH에서 방출할 수 있는 수용체[14] 등이 이제껏 개발되어 왔다.
그러나 지금까지 서술한 i-motif, 삼중나선구조 등은 모두, pH에 반응하는 DNA 나노구조를 설계하는데 있어서 핵산서열의 치명적인 제한이 있다. i-motif의 경우는 C-quadruplex 형성을 위해 C-rich한 배열이 반드시 필요하고, 삼중나선구조의 경우에도 CG, AT Watson-Crick pairing과 Hoogsteen interaction을 위한 C, T의 염기서열 중의 배치를 상당히 정교하게 설계해야 한다. 이 밖에, A-motif의 경우에도 역시 A-rich한 염기 배열의 삽입이 필수적이다. 따라서, 이러한 기존의 모티프 기반의 pH에 반응하는 DNA 나노구조의 설계적 제약을 해결할 일반적인 방법의 등장이 이제는 새롭게 요구되고 있다.
3. 시험관진화를 이용한 pH 활성화 DNA 나노기계의 개발
pH에 반응하는 DNA 나노구조를 제작하는 새로운 방법으로 mispairing(non-Watson-Crick pairing)이 현재 새롭게 주목받고 있다. DNA는 인산기, Deoxyribose 뼈대, 핵염기(A, T, G, C)로 이루어져 있는데, 그 중에서 산성 조건일때 핵염기의 질소 원자가 에너지적으로 안정화되기 위하여 protonation되고는 한다. 흥미롭게도, mispairing의 경우 중성의 조건에서는 불안정하여 결합하지 않는 것이 일반적이지만 산성 조건에서는 한 쪽 염기가 protonation되면서 이중나선구조를 형성하여 안정화되는 경우가 몇 가지 있다. 가장 대표적으로 C-AH+, C-CH+, G-AH+, U-CH+,A-AH+가 이론적으로 가능하다고 알려져 있고[15], 이러한 mispairing들을 사용하여 pH에 반응하는 DNA 나노구조의 설계가 가능하고 여겨진다. 그러나 이러한 mispairing을 구체적으로 DNA 구조에 어떻게 삽입 및 설계를 해야 pH에 의해 활성화되는 DNA 나노구조를 얻을 수 있는지는 또 하나의 큰 난제이다.
아주 최근에, 본 연구진과 Stanford 의대의 Tom Soh 교수 연구실에 의해 pH에 따라 구조를 변화시킬 수 있는 DNA 나노기계를 시험관진화기술을 통해 최초로 선별한 연구가 세계적인 학술지 Angewandte Chemie International Edition에 발표되었다[16]. 선별된 DNA나노구조는 놀랍게도 기존에 잘 알려진 pH에 반응하는 모티프가 아닌 C-A mispairing들을 이용하여 pH에 따라 구조를 바꾸는 것으로 밝혀졌다. 이제껏 시험관진화는 압타머(aptamer)라 불리우는 목표 물질 인식 기능이나, ribozyme 혹은 DNAzyme과 같은 촉매 기능을 갖는 핵산 배열을 찾기 위해 줄곧 이용되어 왔다. 하지만 본 연구진에 의해 시험관진화기술은 pH와 같은 환경변화를 인식할 수 있는 핵산 배열을 자유자재로 찾아낼 수 있는 방법으로 재탄생할 수 있음이 새롭게 발견되었다. 시험관진화는 보통 1014~15개에 달하는 무작위 핵산 배열 속에서 원하는 기능의 배열을 찾아내는 기술이기에, 복잡한 계산과 설계의 필요 없이 pH에 의해 활성화되는 DNA 나노구조를 손쉽게 찾아낼 수 있는 새로운 방법으로 사용될 수 있음이 입증되었다는데 큰 의미가 있다.
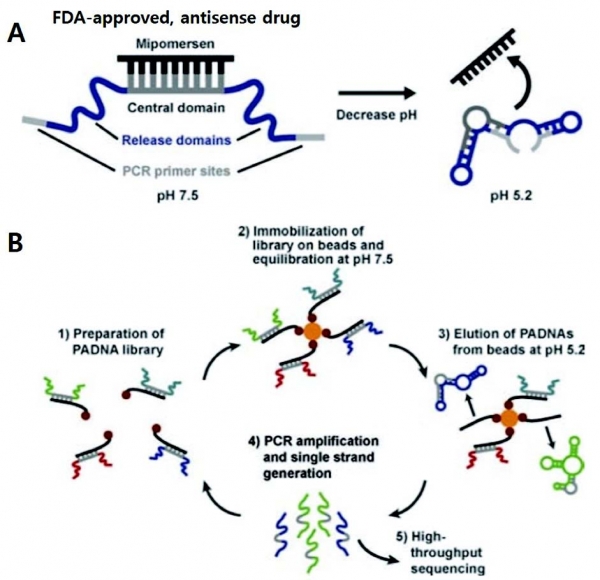
이번 연구는 pH에 반응하는 최적화된 특성을 지니고, pH에 의해 활성화되는 DNA 나노구조를 시험관진화에 기반해제작하는 새로운 방법을 제시하였다[16] (그림 2).
그림 2. pH에 반응하여 Mipomersen, antisense 약물을 선택적 방출할 수 있는 DNA 나노구조의 기본 원리 및 설계(A)와 이를 무작위 핵산 배열로부터 선택해낼 수 있는 시험관진화의 과정(B)(참고문헌 [16]에서 발췌 및 편집)
우리가 제시하는 이 새로운 전략은 시험관진화의 조건을 통해 pH 민감도와 목표 물질의 방출의 효율/속도를 조절할 수 있다. 이러한 전략을 새로운 약물전달기술로 응용하기 위해 이 연구는 Mipomersen을 선택적으로 pH에 의해 방출하는 DNA 나노기계를 제시하였다. 참고로 Mipomersen은 과콜레스테롤증(Hypercholesterolemia)을 치료하기 위해 FDA 승인을 받은 안티센스 DNA 의약품이다. 이 연구를 통해 제작된 pH 활성화 DNA 나노기계(PADNA)는 pH 민감도가 매우 탁월하며, pH가 7.5일 때와 비교했을 때 pH가 5.2일 때는 약 714배에 달하는 놀라운 Mipomersen의 압축적 방출 능력을 보이고 있다.
시험관진화를 위해 설계된 무작위 DNA 배열 라이브러리는 3개의 도메인(Domain)들로 구성되어 있다(그림 2A). 첫번째는 17개의 뉴클레오티드로 구성되어 있는 중앙 도메인이며 이 도메인에 Mipomersen을 선택적으로 결합시킬 수 있고, 이를 통해 pH 7.5에서 Mipomersen을 비활성화된 상태로 묶어 둔다. 이 중앙 도메인 옆에는 21, 22개의 뉴클레오티드로 구성된 임의의 방출 도메인(release Domain)들을 위치시키고, 이들은 시험관진화의 과정을 거쳐 pH에 의해 Mipomersen을 방출할 수 있는 최적의 DNA 배열로 선택될 것이다. 그리고 마지막으로, 라이브러리의 양 끝에 각각 중합효소연쇄반응(PCR, Polymerase Chain Reaction)을 할 때 사용하는 프라이머의 결합 부분들이 각각 위치되어 있다.
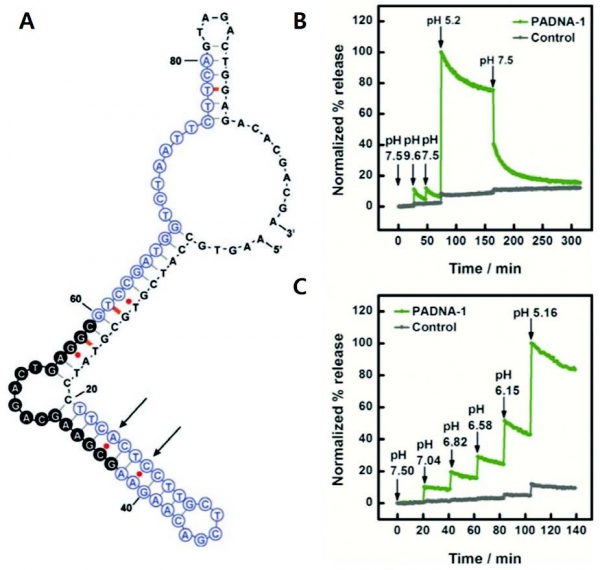
pH 활성화 DNA 나노기계를 선별하기 위한 시험관진화의 방법은 다음과 같이 적용되었다(그림 2B). 처음으로 100 pmole의 DNA 라이브러리와 150 pmole로 구성된 biotin을 결합시킨 Mipomersen을 혼성화하고 streptavidin이 코팅된 자기 구슬(Magnetic bead) 위에 이 하이브리드를 고정시킨다. 고정 후, pH 7.5 버퍼에서 배양하게 되고 고정되지 않은 무작위 PADNA들이 제거된다. 그 다음에 이 조립체를 pH 5.2 버퍼에 넣고 자기구슬들로부터 선택적으로 방출된 PADNA들만 모아지게 된다. 이렇게 pH에 의해 방출되는 PADNA들을 모아 PCR을 통해 증폭하고, 다음 회차의 시험관진화과정을 진행하기 위해서 단일 가닥의 PADNA들로 제작한다. 이러한 과정을 총 12번을 거쳐, pH 7.5에서는 Mipomersen과 효과적으로 결합하지만 엔도솜의 pH에서는 이를 빠르게 방출하는 PADNA를 염기서열분석을 통해 찾아내게 되었고, 찾아낸 PADNA-1는 지금까지 보고된 모티프들과 기계적으로 매우 구별되는 새로운 방식의 pH 활성화 구조를 갖고 있다. 그림 3A에서 볼 수 있는 것처럼 표시된 두 가지의 C-A쌍을 위시한 염기 서열들이 어긋난 것을 안정화시켜 주려는 경향을 갖고 있음이 확인된다.
찾아낸 PADNA-1이 Mipomersen을 pH에 따라 선택적으로 방출하는지 확인되었다. 흥미롭게도 PADNA-1은 원치않는 염기성 pH에 반응해서 Mipomersen을 전혀 방출하지 않지만, pH를 5.2까지 낮췄을 경우, Mipomersen이 PADNA-1 DNA 나노기계로부터 빠르게 방출되는 현상이 관찰되었다(그림 3B). 이는 PADNA-1이 목표로 하는 세포내의 엔도솜으로 이동 시에 빠르게 약물을 방출할 수 있는 매우 중요한 능력이다. 이에 더해서, pH 5.2와 7.5 사이에서 계단형의 연속적인 pH 적정 양상을 확인 시, PADNA-1으로부터 Mipomersen의 방출이 pH의 점차적인 감소와 비례적으로 증가한다는 것을 확인했다(그림 3C). 이 특성을 응용하면 엔도솜 말기의 pH 5.2에서만 제한적으로 Mipomersen을 전달하는 것이 아니라, 엔도솜이 성숙되는 과정 전반에 걸쳐서 Mipomersen을 연속적으로 분포해서 전달할 수 있다는 큰 장점을 보유하게 된다.
그림 3. Mipomersen을 선택적 방출하는 pH 활성화 DNA 나노기계. A. 시험관진화를 통해 찾아낸 PADNA-1의 예상되는 이차원 구조. 산성에서 구조를 안정화시키는 C-AH+ mispairing이 화살표로 표시되어 있다. B.PADNA-1은 염기성에는 반응하지 않고 산성 pH에 의해서만 Mipomersen을 빠르게 방출할 수 있다. C. 실시간 pH 적정 결과는 PADNA-1이 pH가 감소함에 따라 비례적으로 Mipomersen을 방출하는 것을 확인할 수 있다(참고문헌 [16]에서 발췌 및 편집).
4. 시험관진화기술의 재발견
우리 연구진들은 중성에서 산성으로 pH가 낮아질 때 특이적으로 반응하여, DNA 나노구조의 형태 변형을 통해 FDA승인을 받은 antisense 약물인 Mipomersen을 선택적으로 방출하는, pH 활성화 DNA 나노기계를 시험관진화의 기술을 이용하여 완성하였다. 우리가 제작한 pH 변화에 의존하는 구조 전환 배열 중 어느 것도 이미 알려진 모티프들과 관련이 없었으며, 이는 우리가 새로운 구조 전환 기제를 발견해냈음을 말해준다. 결국 시험관진화의 과정으로부터 자연스럽게 삽입, 조절된 C·AH+ mispairing을 통해 PADNA-1가 pH에 효과적으로 반응하는 기능을 갖추게 되었고, 이는 아주 정교하게 통제된 약물 전달 기제로 사용될 수 있는 잠재력을 갖고 있다.
기존의 i-motif나 삼중나선구조 스위치와 같은 모티프들을 사용하는 방법은 매우 매력적이긴 하지만, 안타깝게도 나노기계의 설계는 이미 결정된 기하학적 구조와 특정 배열 방향을 만족시켜야 한다는 점들로 인해 그 사용이 크게 제한된다. 더욱이 pH 민감도의 범위 조율과 같은 특정한 기능을 부여하기 위해서는 매우 복잡한 설계가 뒤따르기 마련이다. 이에 반하여, 시험관진화를 이용한 우리 연구진들의 창의적 방법은 새로운 기능적인 DNA 모티프들을 발견할 수 있는 기회를 줄 뿐만 아니라, 복잡한 설계의 과정 없이 pH에 확연히 반응하는 특성에 더하여 원하는 부가적 기능들을 장착한 DNA 나노기계의 제작으로 이어질 것으로 예상한다. 이를 통해, 완성된 DNA 나노기계들은 약물 전달, 바이오센서, pH에 의해 활성화되는 표면 등 다양한 분야로 응용할 수 있고, 시험관진화의 기술은 이번 연구에 사용된 pH 변화를 포함한 다양한 자극에 의해 더욱더 다양한 기능을 수행할 수 있는 혁신적 DNA 나노기계들[17-19]을 제작하는 중요한 방법론을 제공할 것으로 믿는다.
참고문헌
1. R. Duncan, S. C. W. Richardson, “Endocytosis and intracellular trafficking as gateways for nanomedicine delivery: opportunities and challenges”, Mol. Pharm., 9, 2380 (2012)
2. D. Schmaljohann, “Thermo- and pH-responsive polymers in drug delivery”, Adv. Drug Delivery Rev., 58, 1655 (2006)
3. P. Charoenphol, H. Bermudez, “Design and application of multifunctional DNA nanocarriers for therapeutic delivery”, Acta Biomater., 10, 1683 (2014)
4. K. Gehring, J. –L. Leroy, M. Guéron, “A tetrameric DNA structure with protonated cytosine-cytosine base pairs, Nature, 363, 561(1993)
5. A. Idili, A. Vallée-Bélisle, F. Ricci, “Programmable pH-triggered DNA nanoswitches”, J. Am. Chem. Soc., 136, 5836 (2014)
6. S. Chakraborty, S. Sharma, P. K. Maiti, Y. Krishnan, “The poly dA helix: a new structural motif for high performance DNA-based molecular switches”, Nucleic Acids Res., 37, 2810 (2009)
7. M. Guéron, J. –L. Leroy, “The i-motif in nucleic acids”, Curr. Opin. Struct. Biol., 10, 326 (2000)
8. Y. Dong, Z. Yang, D. Liu, “DNA nanotechnology based on i-motif structures”, Acc. Chem. Res., 47, 1853 (2014)
9. S. Modi, M. G. Swetha, D. Goswami, G. D. Gupta, S. Mayor, Y. A. Krishnan, “A DNA nanomachine that maps spatial and temporal pH changes inside living cells”, Nat. Nanotechnol., 4, 325 – 330 (2009)
10. W. Li, J. Wang, J. Ren, X. Qu, “Near-infrared- and pH-responsive system for reversible cell adhesion using graphene/gold nanorods functionalized with i-motif DNA”, Angew. Chem. Int. Ed., 52, 6726 (2013)
11. D. Leitner, W. Schröder, K. Weisz, “Influence of sequence-dependent cytosine protonation and methylation on DNA triplex stability”, Biochemistry, 39, 5886 (2000)
12. A. Amodio, A. F. Adedeji, M. Castronovo, E. Franco, F. Ricci, “pH-controlled assembly of DNA tiles”, J. Am. Chem. Soc., 138,12735 (2016)
13. J. Ren, Y. Hu, C.-H. Lu, W. Guo, M. A. Aleman-Garcia, F. Ricci, I. Willner, “pH-responsive and switchable triplex-based DNA hydrogels”, Chem. Sci., 6, 4190 (2015)
14. A. Porchetta, A. Idili, A. Vallée-Bélisle, F. Ricci, “General strategy to introduce pH-induced allostery in DNA-based receptors to achieve controlled release of ligands, Nano Lett., 15, 4467 (2015)
15. A. K. Jissy, A. Datta, “Designing molecular switches based on DNA-base mispairing”, J. Phys. Chem. B, 114, 15311 (2010)
16. F. Fong, S. S. Oh, C. J. Hawker, H. T. Soh, “In vitro selection of pH-activated DNA nanostructures”, Angew. Chem. Int. Ed., 55,15258 (2016)
17. S. S. Oh, K. Plakos, X. Liu, Y. Xiao, H. T. Soh, “In vitro selection of structure-switching, self-reporting aptamers”, Proc. Natl. Acad. Sci. U.S.A., 107, 14053 (2010)
18. S. S. Oh, K. Plakos, Y. Xiao, M. Eisenstein, H. T. Soh, “In vitro selection of shape-changing DNA nanostructures capable of binding-induced cargo release”, ACS Nano, 7, 9675 (2013)
19. H. Qu, A. Csordas, J. Wang, S. S. Oh, M. Eisenstein, H. T. Soh, “A rapid, label-free strategy to isolate high-quality aptamers for metal ions”, ACS Nano, 10, 7558 (2016)
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update