| 딥러닝 기반 생물공학의 진화 | |
|---|---|
| Date 2020-07-29 17:52:19 |
|

교수
울산과학기술원 에너지및화학공학부
dkim@unist.ac.kr
1. 딥러닝이란?
지난 몇 년간 인공지능 (artificial intelligence, AI)에 대한 보도가 미디어에서 경쟁적으로 있었으나, 머신러닝 (machine learning, ML), 딥러닝 (deep learning, DL)과 인공지능을 혼용해서 사용하면서, 생물공학 전공자들을 포함한 인공지능 비전문가들과 일반인이 관련 기술을 이해하는 데 어려움을 겪고 있다. 현재 그리고 미래의 인공지능 기술자들은 잡음 속에서 의미 있는 신호와 데이터를 잡아내듯이, 홍수처럼 쏟아지는 정보들 속에서 세상을 바꿀 기술 발전을 알아차리기 위해 인공지능/머신러닝/딥러닝의 정의부터 간단하게 정리를 하고자 한다.
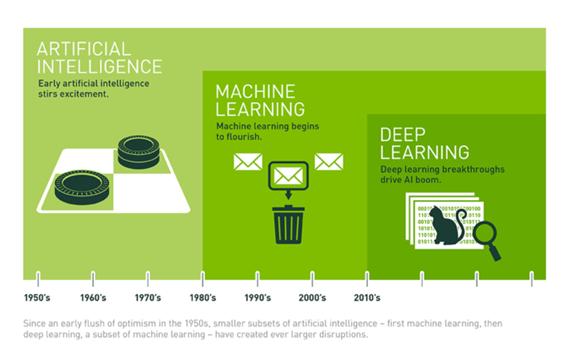
그림 1. 인공지능/머신러닝/딥러닝의 차이 (출처: Michael Copeland, Nvidia)
인공지능은 1950년대에 초기 컴퓨터과학 분야의 선지자들이 “컴퓨터가 ‘생각’할 수 있는가?”라는 질문에서 시작하였다. 이 철학적인 질문의 답변은 지금도 연구 주제로 남아있지만, 그동안의 연구를 통해 도달한 간결한 정의는 보통의 사람이 수행하는 지능적인 작업을 자동화하기 위한 연구 활동 또는 과학 분야로 정리할 수 있다. 이러한 인공지능은 머신러닝과 딥러닝을 포괄하는 종합적인 분야이며, 학습 과정이 전혀 필요 없는 다양한 방법을 포함하고 있다. 자동화된 기계가 인간과 얼마나 비슷하게 대화할 수 있는지를 기준삼아 기계에 지능이 있는지를 판별하고자 하는 시험으로 1950년 앨런 튜링에 의해 튜링 테스트라는 개념이 제안되었으며, 이미테이션 게임이라고도 부르며 얼마 전 동명의 영화가 개봉하였다.
인공지능의 한 분야인 머신러닝은 “우리가 특정 목표를 달성하기 위해 ‘명시적으로 지시하는 것’ 이상을 컴퓨터가 처리하는 것이 가능한가? 그리고 특정 작업을 수행하는 법을 스스로 학습할 수 있는가? 개발자가 직접 만든 명시적인 데이터 처리 규칙 대신 컴퓨터가 데이터에서 자동으로 이런 규칙을 학습할 수 있을까?”라는 질문에서 시작하였다. 머신러닝에 속하지 않는 전통적인 인공지능 분야에서는 명시적인 규칙과 데이터를 넣으면 해답이 나오는 패러다임이 기반이 되었으나, 머신러닝에서는 데이터와 해답을 넣으면 규칙이 나오는 새로운 패러다임을 바탕으로 하고 있다. 따라서, 머신러닝은 시스템을 명시적인 규칙으로 프로그램하는 것이 아니라 학습 혹은 훈련을 통해 시스템에서 사용할 규칙을 명시적/암시적으로 구체화하는 과정을 거치게 되며, 이때 통계적인 방법론을 사용한다는 점에서 기존의 인공지능과 다르다고 할 수 있다. 머신러닝에서 많이 연구된 방법론 중에는 생물학적인 원리를 적용한 신경망 (artificial neural network), 유전 알고리즘 (genetic algorithm), 수학적/통계적 모델을 기반으로 하는 선형 모델, 베이지안 모델, 커널 기법을 활용한 SVM (support vector machine) 등 다양한 방법론이 있다.
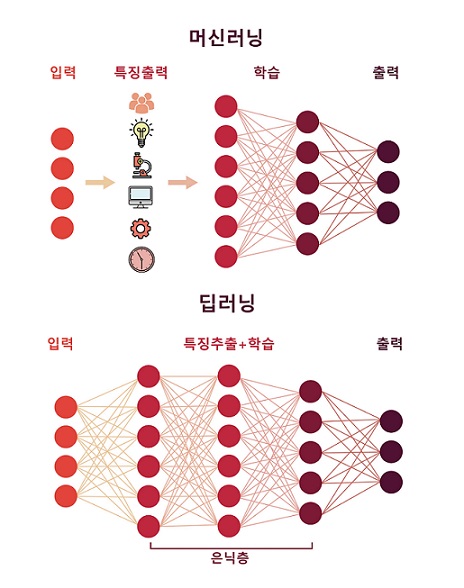
그림 2. 머신러닝의 신경망과 딥러닝의 차이
머신러닝의 한 분야인 딥러닝은 신경망을 근간으로 하지만, 다수의 은닉층(hidden layer)를 가지고 있는 깊은 신경망(deep neural network)를 사용하며, 2010년 이후에 폭발적인 각광을 받기 시작한다. 필자가 컴퓨터공학과에서 공부하던 2000년 초반에는 성능상의 문제를 포함한 몇 가지 이슈로 인해 신경망 연구가 많은 주목을 받지 못했다. 연구자들 사이에는 논문 제목에 신경망이라는 단어가 들어가면, 학술잡지 편집자가 바로 출판 거부를 한다는 소문이 있을 정도였다. 신경망의 성능을 상대적으로 낮게 보았던 주요 원인으로 1. 학습에 사용할 많은 양의 데이터가 필요하며, 2. 대용량 데이터 학습에 들어갈 계산을 수행할 하드웨어의 한계를 꼽을 수 있다. 하지만, 10-15년 동안 무어의 법칙을 따라 CPU/메모리를 포함한 하드웨어 성능이 비약적으로 향상되었으며, 무어의 법칙을 넘어서는 속도로 데이터가 생산되고 있어, 이 두 가지 문제가 해결되었다. 특히, 신경망 계산의 근간이 되는 순전파 (forward propagation), 역전파 (back propagation)에 들어가는 행렬 계산을 CPU뿐만 아니라, 그래픽 카드에 들어있는 GPU를 통해 계산할 수 있도록 하드웨어가 개선되고, 심지어 신경망 계산에만 최적화된 전용 TPU (tensor processing unit)가 개발되어 신경망 학습과 사용에 필요한 하드웨어의 성능이 비약적으로 향상되었다.
하드웨어의 향상과 대용량 데이터 생산이 가능해진 지금, 딥러닝은 인공지능/머신러닝 분야의 핵심 기술이 되었다. 획기적인 발전을 이룬 딥러닝을 이용해 사람과 비슷한 수준의 이미지/음성/필기 인식, 향상된 기계번역/자연어처리, 향상된 TTS (text-to-speech) 변환, 사람과 비슷한 수준의 자율주행 능력, 구글/빙/바이두에서 사용하는 광고 타게팅, 향상된 웹 검색 엔진, 사람을 능가하는 바둑 실력과 게임 실력이 가능해졌다.
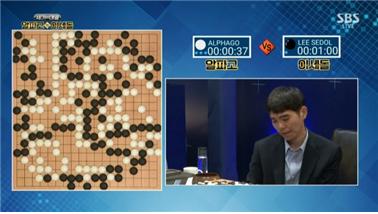
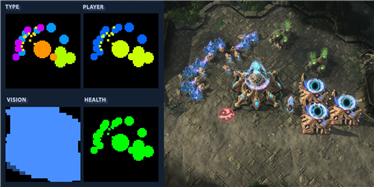
그림 3. 이세돌 알파고와 5국(위)와 알파스타 화면(아래) (출처: SBS와 알파스타 홈페이지)
2. 딥러닝의 장점과 한계
딥러닝은 머신러닝의 부분집합이지만 딥러닝 이전의 머신러닝에 비해 딥러닝이 가지는 차이점과 장점이 몇 가지 있다. 그중 가장 중요한 것은 특징추출 (feature extraction) 단계가 딥러닝에서 사라졌다는 점이다. 기존의 머신러닝에서는 사람이 입력 데이터에서 특징 중 중요한 것들을 추출해서 학습에 사용하는 특징 엔지니어링 (feature engineering) 단계가 매우 중요했다. 특징 엔지니어링은 학습 모델의 정확도를 향상시키기 때문에 머신러닝의 기본 작업이며, 이때 데이터가 만들어진 분야의 도메인 지식 (domain knowledge)을 요구한다. 생물공학 데이터를 활용하기 위해서는 생물학 데이터의 도메인 지식이 필요하며, 이미지 데이터를 활용하기 위해서는 이미지 데이터에 대한 도메인 지식이 필요하다. 즉, 딥러닝 이전의 머신러닝에서 는 해당 분야의 전문가들만 머신러닝을 통해 학습기능을 활용할 수 있었다. 딥러닝에서는 특징추출 과정은 딥러닝 모델의 은닉층에서 자체적으로 실행할 수 있어서 더 빠른 학습과 비전문가의 진입장벽을 낮추는 효과를 가져왔다.
딥러닝의 두 번째 장점은 비정형 데이터에서 좋은 성능을 보인다는 점이다. 회사와 연구소에서 사용하는 데이터 중 가장 많은 비중을 차지하는 데이터는 텍스트, 이미지, 문서와 같은 구조화되지 않은 데이터이다. 즉, 학습을 염두에 두고 만들어진 데이터가 아닌 데이터들을 주로 사용해야 한다. 생물학의 기본이 되는 핵산과 단백질의 염기서열도 텍스트와 비슷하게 학습을 염두에 두고 만들어진 데이터가 아닌 비정형 데이터의 예가 된다. 기존의 머신러닝과 비교하여 딥러닝 모델은 비정형 데이터에서 좋은 성능을 내는 것으로 알려져 있다.
딥러닝 모델 중 일부, 특히 합성곱신경망은 과적합(overfitting)이 상대적으로 덜한 것으로 알려져 있다. 과적합이란 머신러닝에서 학습 데이터를 과하게 학습하여, 학습 데이터에 대해서는 오차가 적어 데이터를 잘 표현하지만, 실제 데이터에서는 오차가 오히려 증가하는 현상을 뜻한다. 합성곱신경망을 포함한 딥러닝 모델에서는 층별 정규화 (normalization), 은닉층에 드랍아웃 (drop-out) 적용, 시그모이드 활성 함수 (sigmoid activation function)의 대체 등의 기술적인 발전을 적용해 과적합을 피하는 방법론이 개발되었다.
이러한 소프트웨어적인 기술 발전과 더불에 GPU와 TPU를 이용한 병렬 계산과 클라우드 컴퓨팅을 이용한 분산 시스템의 발전으로 빅데이터를 처리하는 기반 기술로 발전하였으며, 얕은 신경망 (shallow neural network)에 비해 더 적은 수의 처리 유닛으로 더 높은 성능을 내고 있으며, 전문가의 개입이 필요했던 특징추출도 자동으로 수행되어 생물공학을 포함한 다양한 분야에서 성능을 발휘하고 있다.
대부분의 머신러닝 모델들은 데이터의 양이 증가할수록 성능이 향상되다가 어느 시점에 성능 향상이 멈추는 경향을 보이지만, 딥러닝 모델들은 데이터의 양이 증가할수록 꾸준히 성능이 향상되는 장점을 보인다. 이러한 딥러닝 모델도 한계가 있는데, 머신러닝 모델들의 약점 중 일부를 가지고 있다. 머신러닝과 딥러닝 모델들은 환경변화에 따라 지속해서 재학습이 필요하다는 점이며, 아직 딥러닝이 가장 못 하는 것 중의 하나가 실시간 학습이라는 점도 약점으로 꼽힌다. 예를 들어, 보행자, 자동차, 오토바이를 구분하는 딥러닝 모델을 디자인하고 학습시킨 후, 트럭을 추가로 구분하도록 하려면 학습을 처음부터 다시 해야 한다.
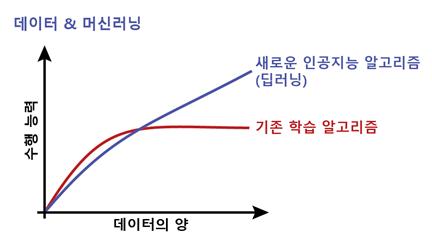
그림 4. 데이터의 양과 학습 모델의 성능 사이의 상관관계
딥러닝 모델을 이용한 학습의 또 다른 한계는 모델이 동작하는 방식에 대한 설명이 되지 않는다는 점이다. 즉, 주어진 문제에 대해 동작이 우수한 모델을 디자인하고 학습한 뒤에도 왜 우수한 성능을 내는지를 설명할 수 없다. 이런 특징 때문에 딥러닝 모델은 보통 블랙박스 (black box)로 비유되곤 한다. 이러한 딥러닝 모델의 한계는 학습의 재현성이라는 심각한 문제를 유발하고 있으며, 냉소적인 어조로 딥러닝을 사용하는 사람들이 외계인의 기술을 운영하는 것처럼 느낀다거나, 21세기의 연금술이라고 비유하는 경우도 있다. 하지만, 이런 기술적인 한계들은 다양한 기법과 기술의 개발로 점차 해소될 것으로 기대하고 있다.
3. 생물공학에 적용된 딥러닝
딥러닝 모델들은 다른 머신러닝 모델보다 성능이 상대적으로 좋은 비정형 데이터들에 먼저 적용이 되었다. 텍스트와 음성 데이터를 이용한 자연어 처리 (natural language processing), 이미지와 비디오 데이터 처리 등의 문제에 가장 활발히 적용됐다. 어느 정도 기술적인 성숙이 된 이후에 기존의 비정형 데이터와 유사한 생물공학 및 생물학 데이터에도 적용이 가속화되었으며, 그 대표적인 예로 단백질의 3차원 구조 예측 모델인 알파폴드 (AlphaFold)를 들 수 있다.
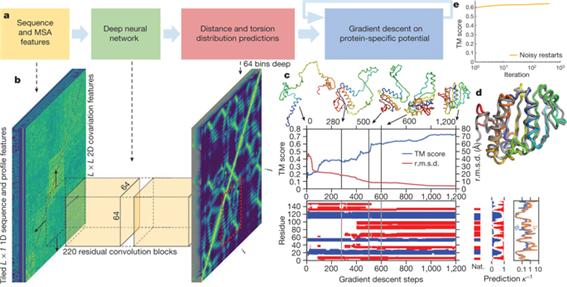
그림 5. <알파폴드 동작 개요도> (출처: Senior AWe t al., Nature, 2020)
알파폴드가 풀고자 했던 문제는 단백질의 1차원적인 염기서열 정보로부터 3차원 구조를 예측하는 문제이다. 단백질의 구조는 단백질의 기능과 밀접히 연관되어 있어 매우 중요하나, 그동안 Cryo-EM, 핵자기 공명 (nuclear magnetic resonance), 엑스선결정학 (X-ray crystallography)과 같은 실험적인 방법으로만 구조를 결정할 수 있었다. 하지만 실험적인 구조 결정 방법은 성공확률이 낮고 연구 기간이 길어, 실험 없이 계산으로 구조를 예측하고자 하는 시도가 계속해서 있었으며, CASP (critical assessment of structure prediction)13 대회도 이런 시도의 연장선에 있다. 알파폴드는 수십년간 단백질 구조 계산을 연구하던 모든 대학 연구팀을 물리치고, 압도적인 성능 차이로 CASP13 대회를 우승한 뒤, 학술지에 연구 결과와 딥러닝 모델 코드의 일부분을 공개하였다.
단백질 구조를 계산만으로 예측할 수만 있다면, 단백질 구조에 다른 화학 분자가 물리 화학적으로 어떻게 결합할 것인지를 효율적으로 예측할 수 있게 되며, 신약 개발/효소와 연관된 질병 이해 등에 획기적인 이바지를 할 것으로 예상한다. 알파폴드는 분자들의 상호 거리 예측→후보구조 예측→적합한 후보들의 우선순위를 결정하는 방법을 적용했다. 핵심적인 딥러닝 알고리즘으로는 딥러닝 모델 중의 하나인 GAN (generative adversarial network) 생성 모델을 사용하였다. 알파폴드를 개발한 딥마인드 (DeepMind)의 기술 향상은 구글의 거대한 데이터 센터의 컴퓨터 자원을 최대한 지원받음으로써 가능했다. 즉, 알파폴드의 성공은 소프트웨어의 발전도 중요하지만 풍부한 하드웨어/컴퓨터 자원도 중요하다는 시사점이 있다.
2012년에 있었던 머크 분자 물성 예측 대회 (Merck molecular activity challenge)에서도 분자들의 구조와 물성 관계를 예측하는데 딥러닝 모델을 적용한 팀이 우승하면서 신약 개발에도 딥러닝 모델이 유효하다는 것이 입증되고 있다. 머크 (Merck)와 노바티스 (Novartis)같은 신약 개발에 투자하는 회사들은 단백질 구조예측 문제를 딥마인드처럼 잘 해결하지는 못하고 있다. 하지만, 후보물질 및 바이오 마커 발굴과 같은 신약 개발 과정의 여러 다른 과정에서 딥러닝을 이미 적용해 왔다. 노바티스의 경우는 이러한 딥러닝 기술이 신약 개발 분야에 적용되는 것은 시간문제라고 여기고 있으며, 미래에는 신약 개발 전체에 딥러닝 기술이 확장 및 적용될 것이라고 예상한다.
단백질과 화학물질의 구조뿐만 아니라 염기서열 자체를 연구하는 데도 딥러닝 모델들이 적용되어 성과를 내고 있다. 여러 딥러닝 모델을 활용해 특정 유전적 변이로 인해 세포 내 단백질의 활성이 어떻게 변화하는지 연구하였으며, 딥러닝 기술을 이용해 조직별로 다양한 엑손-인트론 스플라이싱 패턴을 예측하는 SPIDEX가 2014년에 개발되었다. 단백질이 결합하는 유전체 상의 위치를 예측하는 딥바인드 (DeepBind)라는 프로그램이 2015년에 개발되었으며, 2017년 구글에서 개발한 딥베리언트 (DeepVariant)는 차세대 유전체 해독 (next generation sequencing, NGS) 데이터에서 정확한 염기서열의 판독을 위해 적용되기 시작하였다.
합성생물학 분야의 다양한 구성요소에도 딥러닝이 활발히 적용되고 있다. 유전자 발현 조절에 중요한 프로모터의 염기서열 디자인에 딥러닝을 적용한 연구 결과가 2020년에 발표되었으며, 딥러닝 모델을 이용해 효소의 기능 예측에 중요한 EC 번호를 신속하게 예측하는 DeepEC 알고리즘도 2019년에 개발되었다. 또한, 2019년에 새로 발굴된 유전체에서 전사인자를 찾아내는 알고리즘도 개발되었으며, ChIP-exo처럼 전사인자의 유전체 결합 위치를 확인하는 실험 데이터로부터 실제 결합 위치를 예측하는 알고리즘도 활발히 연구되고 있다.
4. 맺음말
컴퓨터과학과 컴퓨터공학의 토양에서 자라온 머신러닝과 딥러닝은 그 기술이 지속해서 성숙하고 있을 뿐만 아니라 사용의 용이성도 좋아지고 있다. 딥러닝 기술 사용에 필요한 기술적인 진입장벽이 낮아지면서, 이제는 컴퓨터 전공자들의 전유물이 아닌 모든 연구자가 비교적 손쉽게 사용할 수 있는 엑셀과 같은 도구가 되어가고 있다.
물론 딥러닝이 여전히 가지고 있는 여러 단점과 한계가 분명히 존재하지만, 다양한 기법과 새로운 모델의 개발로 한계점들은 하나씩 해결될 것으로 기대한다. 그리고 한동안은 전통적인 컴퓨터공학에서 다루던 텍스트, 음성, 이미지와 같은 비정형 데이터에서 개발된 새로운 기법들이 생물학과 생물공학의 데이터에 적용되는 기술 유입의 방향으로 연구와 개발이 진행될 것으로 예상한다.
딥러닝 모델에서 활발히 연구되고 있는 합성곱신경망, 생성 모델 뿐만 아니라 스타일 전달 (style transfer), 강화 학습(reinforcement training)을 포함한 다양한 기법과 모델이 어떻게 생물공학 기술에 접목될지 상상하니 그 미래가 매우 흥미롭다고 할 수 있다.
참고문헌
Has artificial intelligence become alchemy?. Hutson M. Science. 2018 May 04. doi:10.1126/science.360.6388.478.
Improved protein structure prediction using potentials from deep learning. Senior AW et al. Nature. 2020 Jan 15. doi:10.1038/s41586-019-1923-7.
Deep learning in biomedicine. Wainberg M et al. Nature Biotechnology. 2018 Oct 01. doi:10.1038/nbt.4233.
Synthetic promoter design in Escherichia coli based on a deep generative network. Wang Y et al. Nucleic Acids Research, 2020 May 19. doi:10.1093/nar/gkaa325.
Deep Learning Enables High-Quality and High-Throughput Prediction of Enzyme Commission Numbers. Ryu JY et la. Proceedings of the National Academy of Sciences of the United States of America. 2019 Jul 9. doi:10.1073/pnas.1821905116.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update 
