| 딥러닝 (CNN) 기반 DNA 서열 분석 가이드 | |
|---|---|
| Date 2020-07-30 10:15:24 |
|

선임연구원
한국생명공학연구원 합성생물학전문연구단
haseong@kribb.re.kr
코로나19가 몰고 온 유래 없는 사회적, 경제적 충격으로 한참을 뜨겁게 했던 AI 이슈가 조금은 잠잠해진 듯 하다. 이제는 코로나19의 최초 백신을 누가 그리고 언제 만들지가 전세계인들의 관심사가 되었다. 그 최초에 가장 근접한 것으로 알려진 기업 Moderna는 코로나19 서열 해독 후 42일만에 임상 1상에 들어갔고 임상 1상에서 참가자 전원에 항체를 만들어내는 긍정적인 결과를 보였다. 물론 백신개발이 실패로 돌아갈 가능성은 여전하지만 적어도 이제는 생물학과 관계없는 전세계 모든 사람들이 그들의 연구 결과에 주목하고 있다. Moderna는 스타트업으로 시작할 때부터 철저히 AI와 자동화 기술을 기반으로 운영되어온 합성생물학 기업이다. Moderna의 10-year vision에서는 그들의 모든 연구활동에 자동화와 디지털 기술을 적용하는 것을 핵심 목표로 세웠고 기존 한 달에 40개 mRNA를 설계하고 만들어내던 효율을 지금은 1000개 이상까지 발전시켰다. Moderna가 운영하는 연구 시스템의 최상위에는 AI가 위치해 있으며 백신의 성공 여부를 떠나 기존 틀을 깨는 새로운 도전에 AI가 핵심 역할을 하고 있음은 틀림없는 듯 하다.
최근 딥러닝 기반의 AI 기술은 급속도로 발전하여 이제는 중고등학교 학생들도 python이나 R 등의 프로그래밍 언어를 공부하면 쉽게 AI를 구현할 수 있게 되었다. 이제 관련 도메인 지식과 해당 분야의 빅데이터를 확보하는 것이 성공적인 AI 활용을 위해서 더욱 중요해지는 추세이다. 생물공학을 포함한 생물학 분야에서는 서열 데이터가 대표적인 빅데이터이며 본 기고에서는 이러한 서열데이터를 이용해서 딥러닝을 수행하기 위한 기본적인 기술을 소개하고자 한다. 프로그래밍 언어는 Python을 이용하며 해당 기초 문법은 알고 있음을 가정한다. Python은 전 세계적으로 가장 많이 사용되는 언어로서 만약 사용 경험이 없다 해도 다양한 문헌이나 인터넷 사이트를 통해 어렵지 않게 익힐 수 있다. 특히 자료구조인 List, Tuple, Dictionary 그리고 Numpy의 ndarray 개념을 명확히 이해하는 것은 서열 데이터를 다루는 데 필수이다.
DNA 서열데이터 분석을 위한 딥러닝 기술은 크게 두 종류로 나눌 수 있다. 하나는 Convolutional neural network (CNN)이며 [1] 다른 하나는 Recurrent neural network (RNN)이다 [2]. 이들은 이미지 분석과 텍스트 분석에 각각 널리 사용되는 알고리즘들로서 이미지와 텍스트는 빅데이터와 Goldstandard 데이터를 가장 많이 확보한 분야들이며 딥러닝이 가장 활발히 발전하는 분야라고 볼 수 있다. 생물학적 서열데이터를 분석하는 데 CNN과 RNN 알고리즘을 그대로 활용할 수 있다는 것은 데이터 생산이 느리고 Goldstandard 데이터가 없는 생물학 분야의 연구자들에게는 무척 다행스러운 일이 아닐 수 없다. 본 기고에서는 CNN 기반의 DNA 서열분석을 수행하기 위한 기술을 소개하며 딥러닝 프레임워크 중 하나인 Keras를 이용한 실제 python 분석 코드를 제공함으로써 딥러닝 활용에 관심 있는 생명공학 연구자들이 실제로 활용할 수 있는 정보를 공유하고자 한다. 단, CNN 작동 원리는 이미 많은 참고 자료들이 존재하므로 본 글에서 는 작동 원리보다는 Python 코드를 중심으로 설명을 진행하겠다.
기계학습을 위한 컴퓨터 환경 설정
대용량의 데이터를 다루기 위해서는 python과 같은 프로그래밍 언어의 사용이 필수적으로 요구된다. 특히 많은 계산을 필요로 하는 딥러닝의 경우 CPU나 GPU 같은 하드웨어 의존성이 높은 관계로 오픈소스 기반의 Linux와 라이브러리 패키지들을 선호한다. 생물정보 분석 관련 패키지만 하더라도 약 7000개가 넘는 것으로 알려져 있으며 이들을 설치하거나 관리하기 위해서 다양한 패키지 관리 프로그램이 있으나 본 글에서는 Anaconda 라는 패키지 관리 프로그램을 사용한다 (https://www.anaconda.com/). 아나콘다는 특히 운영체제나 프로그래밍 언어의 종류 그리고 다른 패키지관리 프로그램들에 제약을 받지 않고 함께 사용될 수 있어서 데이터 과학 연구자들 사이에서 널리 사용되고 있다. 본 기고를 작성하는 2020년 5월을 기준으로 다음과 같은 순서로 Python (3.7.7) 과 Anaconda를 (Anaconda3-2020.02) 설치한다 (Anaconda 설치는 컴퓨터 환경에 따라서 시간이 오래 걸릴 수 있음).
1. Python 3.7 다운로드 및 기본 설정으로 Python 설치 [3]
2. Anaconda 다운로드 및 기본 설정으로 Anaconda 설치 [4]
위 순서로 아나콘다를 설치하면 윈도우 시작 메뉴에 Anaconda3가 생성되고 Anaconda Navigator 아이콘을 클릭해서 실행할 수 있으며 Navigator 화면 왼쪽의Environments 탭을 통해 필요한 패키지들을 설치/삭제 할 수 있다. 본 글에서 사용한 biopython 패키지 설치를 위해 “Search package” 버튼에 biopython을 입력하고 리스트 아이템 체크박스에 체크한 후 우측 하단의 apply 버튼을 누르면 설치가 시작된다. 만약 원하는 패키지 검색이 안 될 경우 “Update index” 버튼을 누르면 보일 수 있다. 참고로 윈도우에서 Navigator를 이용한 패키지 관리 시 컴퓨터 리소스를 과도하게 사용해서 느려지는 현상이 가끔 나타나는 이유로 가능하면 명령창 환경을 이용하여 패키지를 관리하는 것이 좋다. 본 글에서 소개하는 실습을 위해 biopython 외에 tensorflow와 keras를 설치할 필요가 있으며 명령창에서 conda 또는 pip를 이용해서 이들 패키지를 설치하는 법은 인터넷 자료 등을 참고하기 바란다. 아래 에디터 사용법을 소개한 글에서도 명령창 사용법을 참고할 수 있다.
Jupyter Lab 에디터 사용
코드를 입력하고 컴파일 할 수 있는 에디터로서 Jupyter Lab을 사용할 것이고 이 소프트웨어는 아나콘다를 설치하면 기본으로 설치된다 (Jupyter Lab은 Jupyter Notebook의 차세대 버전으로서 아나콘다 설치 시에 Notebook과 Lab 둘 다 설치됨). Jupyter Lab은 웹브라우저에서 실행되는 대화형 에디터로서 데이터 분석과 딥러닝 연구에 적합한 편의성과 효율성으로 최근 사용자가 크게 증가하고 있다. 웹브라우저는 Chrome이나 Firefox를 사용하길 권장하며 브라우저를 사용하는 특성상 일반적인 아이콘을 클릭해서 프로그램을 실행하는 방법과는 다르다. 본 글에서는 필자가 주로 이용하는 방법을 소개하며 인터넷 자료를 참고해서 다른 방법으로 실행할 수도 있다. 방법은 윈도우를 기준으로 “제어판” > “시스템 및 보안” > “시스템” > “고급 시스템 설정”으로 이동하여 그림 1과 같이 윈도우 PATH에 anaconda3 경로를 추가한다 (추가할 경로는 anaconda3 설치 디렉토리의 Scripts 디렉토리이며 필자의 경우 로그인 사용자가 user 라고 가정할 경우 다음과 같은 경로임 C:\Users\user\anaconda3\Scripts\).
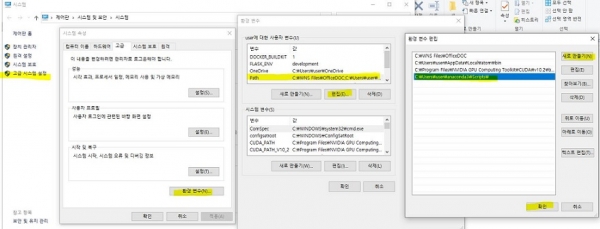
그림 1. 윈도우 환경 설정 (Anaconda3 경로 PATH 추가). 왼쪽에서 오른쪽 방향으로 Highligh 된t 항목부터 차례대로 실행
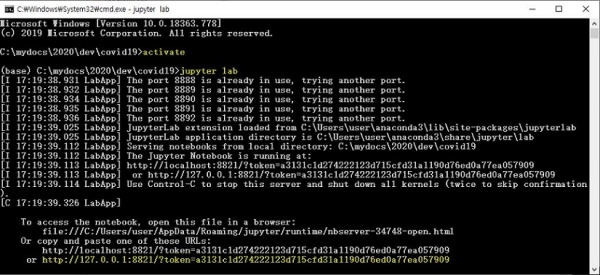
이 후 파일 탐색기를 열고 분석을 원하는 데이터가 있는 디렉토리로 (예를 들어 C:\mydocs\2020\dev\covid19) 이동한 후 탐색기 주소창에 “cmd” 를 입력하고 엔터를 누르면 해당 디렉토리를 홈으로 하는 command 명령창이 나온다 (그림 2). 이 후 “activate” 명령어와 “jupyter lab” 명령어를 실행한다 (참고로 “activate” 명령을 실행하면 (base) 라는 anaconda 환경에 진입했다는 표시가 프롬프트 앞에 붙음, 그림 2 참고).
그림 2. 명령창을 이용한 anaconda 환경 진입과 Jupyter lab 실행
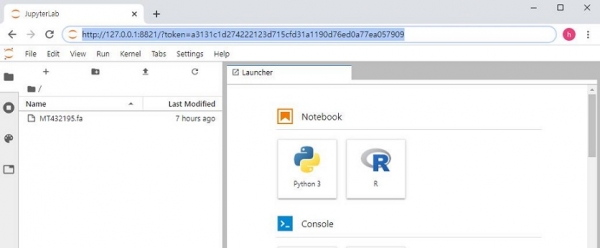
“jupyter lab” 명령어를 실행하면 그림 2와 같이 http://127.0.0.1:8892/?token=99... 로 시작하는 메시지가 출력되며 이 부분을 복사하여 크롬 브라우저의 주소창에 붙여 넣으면 그림 3과 같이 Jupyter lab 에디터가 실행된다 (컴퓨터 설정에 따라 jupyter lab을 실행하면 바로 브라우저가 실행되는 경우도 있음). 에디터 화면에서 왼쪽 판넬은 파일 탐색창 등이 보여지고 오른쪽 화면 Launcher의 Notebook 중 Python3를 클릭하면 파이썬 코드를 작성할 수 있는 에디터가 실행된다. Jupyter lab을 사용하는 방법에 대한 자료는 인터넷에서 쉽게 찾을 수 있으며 자주 사용하는 몇 가지 단축키 사용법도 같이 익혀두도록 한다.
그림 3. 인터넷 브라우저에서 주소창에 복사한 내용을 붙여넣으면 Jupyter lab이 실행됨
딥러닝을 위한 서열 데이터
딥러닝을 위해서는 라벨링 데이터가 필수이다. 예를 들어 동물을 인식하기 위한 딥러닝 모형을 학습한다고 가정할 때 고양이 사진은 “고양이”라는, 강아지 사진은 “강아지”라는 라벨이 붙은 훈련데이터를 모형에 제공해야 한다. 서열분석의 경우에는 DNA 서열과 함께 해당 서열의 표현형이 라벨이 될 수 있다 (Genotype-phenotype 짝 데이터). 예를 들어 특정 전사인자가 결합하는 DNA 서열을 예측하는 딥러닝 모형을 학습하고자 할 경우 전사 시작 위치로부터 약 500bp길이의 DNA 서열 데이터와 해당 전사인자가 위 DNA에 실제로 붙는지를 나타내는 True 또는 False 라벨이 붙은 데이터가 필요하다. 일반적으로 통계적 분석을 위한 데이터는 샘플의 개수와 (행) 변수의 개수로 (열) 구분되어 2차원 배열 형태로 표현된다 (그림 4). 딥러닝에서도 같은 방식으로 데이터를 표현하며 필요한 샘플의 수는 학습할 모형의 복잡도에 따라서 달라질 수 있지만 최소 수천 개 이상이 필요하며 수 만개 이상의 가능한 많은 데이터를 사용하기를 권장하고 있다. 예를 들어 ImageNet 데이터베이스의 경우 약 140만개 이상의 라벨링된 이미지를 보유하고 있으며 계속해서 증가하고 있다 [5]. 딥러닝을 위해서 수집된 데이터 세트는 모형 학습을 위한 Training 데이터와 Test 데이터로 나누어 사용되며 Training 데이터는 또다시 Training 데이터와 Validation 데이터로 나누어 구분할 수 있다. 딥러닝 모형 훈련 과정에서 Training 데이터와 Validation 데이터가 무작위로 분할되어 모수를 학습하고 이 과정을 반복 수행하면서 얻어진 최종 훈련된 모형을 Test 데이터를 이용해서 평가한다.
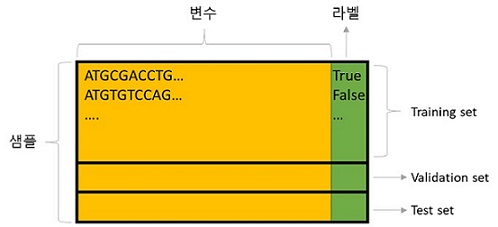
그림 4. 딥러닝 모형 학습 및 평가에 사용되는 데이터 설명
One-hot DNA 서열 인코딩 기술
python은 전세계적으로 가장 많이 사용되는 프로그래밍 언어로서 기계학습이나딥러닝 관련된 다양한 소프트웨어들이 python 라이브러리 형태로 제공되고 있다. DNA 서열 분석을 위해서는 biopython 패키지를 사용하나 딥러닝에서는 서열을 문자열로 다루기 때문에 필수 요구 조건은 아니라고 할 수 있다. 서열을 이용한 기계학습을 수행하기 위해서는 먼저 염기 서열 (A, T, G, C) 문자들을 컴퓨터가 계산하기 쉽도록 변환하는 과정이 필요하다. 크게 두 가지 접근법이 있으며 하나는 One-hot encode 방법과 다른 하나는 Text 데이터와 같이 취급하여 Bag of words를 만들어서 분석하는 방법이다. 이들은 앞에서 언급한 CNN과 RNN 알고리즘이 각각 연계되어 이미지와 자연어처리 분석에 사용된다. 본글에서는 One-hot encoding을 적용하는 CNN 방법을 소개한다. One-hot encoding은 딥러닝에서 가장 널리 사용되는 방법이며 4 종류의 염기를 갖는 DNA의 경우 “A”는 [1,0,0,0], “T”는 [0,0,0,1], “G”는 [0,0,1,0], 그리고 “C”는 [0,1,0,0] 으로 인코딩 할 수 있다. 따라서 “ATGCTA”는 [[0,0,0,1], [0,0,0,1], [0,0,1,0], [0,1,0,0], [0,0,0,1], [1,0,0,0]]의 6x4 행렬로 표현 된다. scikit-learn 패키지는 이러한 인코딩을 쉽게 수행할 수 있는 함수 OneHotEncoder를 제공하고 있으나 아래와 같이 직접 서열에 대한 인코딩을 수행 할 수도 있다.
import numpy as np
my_string=“ATACAA”
my_array=np.array(list(my_string))
onehot_encode = np.zeros((len(my_array),4), dtype=int)
base_dict = {‘A’:0, ‘C’:1, ‘G’:2, ‘T’:3}
for i in range(len(my_array)):
onehot_encode[i, base_dict[my_array[i]]]=1
print(onehot_encode)
[[1 0 0 0]
[0 0 0 1]
[1 0 0 0]
[0 1 0 0]
[1 0 0 0]
[1 0 0 0]]
결과에서 보는 바와 같이 2차원의 6x4 배열이 생성되었음을 확인할 수 있으며 이러한 인코딩을 적용한 서열 데이터는 CNN 모형의 첫 레이어에 입력되는 데이터로 사용된다.
CNN 모형 이해를 위한 PWM 계산과 모티프 탐색
CNN 모형을 이해하기 위해서는 먼저 PFM (Position Frequency Matrix)와 PWM (Position Weight Matrix) 개념의 이해가 필요하다. PWM 방식의 계산을 통해 특정 전사인자의 결합 서열을 찾는 연구는 생물정보학 분야에서 오랫동안 연구되어온 분야 중 하나이다 [6]. 설명을 위해 alignment가 수행된 몇 개의 서열들을 가정하면 PFM은 이 서열들의 특정 위치에 A, T, G, C 각 염기들의 빈도수를 나타내며 PWM은 각 염기의 비율을 나타낸다. 다음은 세 개의 서열에서 PFM과 PWM을 구하는 코드로서 PWM 값이 계산되는 과정을 익혀두자.
from Bio import motifs
from Bio.Seq import Seq
instances = [Seq(“TACAA”), Seq(“TACGA”), Seq(“TACAA”)]
m = motifs.create(instances)
pfm = m.counts
pwm = m.counts.normalize(pseudocounts=0.5)
print (pwm)
0 1 2 3 4
A: 0.10 0.70 0.10 0.50 0.70
C: 0.10 0.10 0.70 0.10 0.10
G: 0.10 0.10 0.10 0.30 0.10
T: 0.70 0.10 0.10 0.10 0.10
pseudocounts는 계산시 NULL이나 0으로 나누어지는 경우를 방지하기 위한 조치로 각 PFM의 각 원소에 0.5를 더하여 PWM을 구한다. 이렇게 얻어진 특정 서열 모티프의 PWM은 새로운 서열이 One-hot encoding 방식으로 주어져 있을 경우 서열 처음 위치부터 마지막까지 Sliding window 방식으로 해당 모티프가 있는 위치를 탐색할 수 있다. 앞서 One-hot encoding을 설명하기 위한 “ATACAA” 서열에서 위 PWM 모티프가 존재하는지 탐색하는 과정을 살펴보자. “ATACAA”는 길이 5인 슬라이딩 윈도우를 사용하면 “ATACA”와 “TACAA” 두 개의 서열로 나눌 수 있다. 이 두 서열을 One-hot encoding으로 전환 후 위 PWM과 원소들끼리 곱하면 One-hot encoding에서 0이 아닌 위치와 동일 위치의 PWM 값들만 남게 된다. 여기서 0이 아닌 값들을 모두 곱한 후 log를 취해 주면 해당 서열이 모티프와 얼마나 비슷한지를 나타내는 스칼라 값이 구해진다. 이론적으로 이 값이 0이면 동일한 서열이다. 아래 코드의 s2 값이 0에 더 가까우므로 두 번째 서열 TACAA이 첫 번째 서열보다 주어진 모티프와 유사하다는 올바른 결론을 보여주고 있다.
pwm_arr = np.array(list(pwm.values())).transpose()
s1 = np.multiply(onehot_encode[0:5,], pwm_arr)
s2 = np.multiply(onehot_encode[1:6,], pwm_arr)
print(np.log(np.prod(np.sum(s1, axis=1)))) #s1 score
print(np.log(np.prod(np.sum(s2, axis=1)))) #s2 score
-9.567015315914915
-2.119846956314875
DNA 서열 분석용 모의 데이터 생성
앞서 설명한 PWM을 이용한 모티프 탐색 과정을 실제 딥러닝 과정에서 구현할 필요는 없다. One-hot encoding 방법으로 서열을 변환하여 입력 데이터로 사용할 경우 앞에서 언급한 바와 같이 이미지 분석에서 사용한 딥러닝 알고리즘과 동일한 알고리즘을 그대로 가져다 사용할 수 있다. 본 글에서는 분석에 필요한 데이터를 외부에서 읽어오는 대신 모의 서열 데이터를 생성하여 분석을 수행하도록 한다. 전사인자가 결합하는 특정 DNA 모티프를 찾는 딥러닝 모형을 개발하는 것을 목표로 하며 바인딩 모티프, 즉, PWM 생성에 사용된 모티프는 “CCGGAA”로 미리 설정해 둔다. 전체 서열의 길이는 20bp로 모티프 양쪽 7bp를 랜덤 서열로 할당했으며 위 모티프를 갖는 positive 서열과 20bp 전부가 랜덤 서열인 negative 서열 각각 1000개씩을 생성했다.
import numpy as np
seq_length = 20
num_sample = 1000
#motif CCGGAA
motif_pwm = np.array([[10.41, 22.86, 1.92, 1.55, 98.60, 86.66],
[68.20, 65.25, 0.50, 0.35, 0.25, 2.57],
[17.27, 8.30, 94.77, 97.32, 0.87, 0.00],
[4.13, 3.59, 2.81, 0.78, 0.28, 10.77]])
pwm = np.hstack([np.ones((4, 7)), motif_pwm, np.ones((4, 7))])
pos = np.array([np.random.choice( ['A', 'C', 'G', 'T'], num_sample,
p=pwm[:,i]/sum(pwm[:,i])) for i in range(seq_length)]).transpose()
neg = np.array([np.random.choice( ['A', 'C', 'G', 'T'], num_sample,
p=np.array([1,1,1,1])/4) for i in range(seq_length)]).transpose()
print(pos.shape)
[''.join(x) for x in pos[1:5,]]
['AGTTCCAGCCGAAGCCCGAG',
'TAGGAACCCGGAATGGGCAA',
'TTCTGGACAGGAAGTCCAAA',
'GTTGGTTCCTGAAGAGGTTC']
위 출력에서 보듯 positive (pos) 서열의 8번째 위치부터 CCGGAA 패턴의 서열이 많이 발생하고 있는 것을 확인할 수 있으며 negative (neg) 서열을 출력해 본다면 전체 서열이 랜덤하게 분포되어 있는 것도 확인할 수 있을 것이다. 전사인자가 결합하는 유무를 나타내는 각 서열의 라벨링 데이터는 다음 전처리 과정에서 One-hot encoding 형식으로 바로 생성하도록 한다.
DNA 서열 데이터 전처리
다음으로 생성된 모의 서열 데이터를 CNN 모형에 적용하기 위한 One-hot encoding 변환과 training 및 test 데이터세트를 분할하는 과정을 수행한다. One-hot encoding 변환은 전반부에 설명한 방법과 동일하며 다만 positive와 negative 서열들 각각에 대한 코딩과 이들을 vstack 함수를 이용해서 하나의 매트리스로 병합하는 과정이 필요하다. 서열들의 라벨링 데이터는 결합 모티프가 존재하는 positive 서열의 경우 [0,1], negative 서열의 경우는 [1,0]으로 Onehot encoding 형식으로 생성한다.
base_dict = {‘A’:0, ‘C’:1, ‘G’:2, ‘T’:3}
onehot_encode_pos = np.zeros((num_sample, seq_length, 4))
onehot_encode_pos_label = np.zeros((num_sample, 2), dtype=int)
onehot_encode_pos_label[:,0] = 1
onehot_encode_neg = np.zeros((num_sample, seq_length, 4))
onehot_encode_neg_label = np.zeros((num_sample, 2), dtype=int)
onehot_encode_neg_label[:,1] = 1
for i in range(num_sample):
for j in range(seq_length):
onehot_encode_pos[i,j,base_dict[pos[i,j]]] = 1
onehot_encode_neg[i,j,base_dict[neg[i,j]]] = 1
X = np.vstack((onehot_encode_pos, onehot_encode_neg))
y = np.vstack((onehot_encode_pos_label, onehot_encode_neg_label))
print(X.shape, y.shape)
(2000, 20, 4) (2000, 2)
위 출력에서 확인한 것처럼 인코딩된 데이터 X는 positive와 negative가 병합된 2000개의 샘플이고 각 샘플은 20bp 길이의 서열이며 4개 배열로 one-hot encoding 형식으로 저장되어 있다. 또한 라벨링 데이터 y도 2000개 서열 샘플에 대해서 모두 one-hot encoding 형식의 배열 데이터가 저장되어 있다. 이들 데이터를 training 과 test 세트로 분할하는 과정은 scikit-learn 패키지의 train_test_split 함수를 사용하면 간편하게 수행할 수 있다.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=125)
print(X_train.shape, y_train.shape)
(1600, 20, 4) (1600, 2)
여기서는 test 데이터 세트의 크기를 20%로 설정하여 training 세트는 1600개의 샘플 크기를 가지게 된다. 실제 코드를 실행해보고 각 데이터들의 실제 값들과 차원 등이 올바로 만들어졌는지 확인해 보도록 하자.
Keras 기반 CNN 모형 생성 및 훈련
이제 실제로 CNN 모형을 만들어 보겠다. Keras는 딥러닝을 쉽게 수행할 수 있는 프레임워크로 CNN 모형을 위해 Conv1D, Conv2D 등의 함수를 제공한다. 아래 코드를 참고하자. 우선 Conv1D로 하나의 레이어를 추가하고 레이어의 Unit은 10개로서 10개의 서로 다른 모티프 (Filter)를 이용한다. 하나의 모티프를 기준으로 모티프의 길이로 볼 수 있는 Filter 크기를 5로 설정하였다. 즉, 5x4의 sliding window가 (PWM) 입력 데이터인 20x4의 one-hot encoding 서열에 대해서 1bp씩 움직여가며 score를 계산하는 것과 같은 개념이다. padding을 “same”으로 하고 Maxpooling 크기를 4로 설정하여 20개의 score가 계산되도록 했으며 이를 4개씩 나누어 Max 값을 저장하므로 최종 5개의 score 배열이 출력되며 이런 모티프가 (Unit) 10개 이므로 Max pooling을 수행한 후 출력 shape는 (5, 10) 이다. Flatten 함수는 이를 50 개 원소를 갖는 1차원 배열로 변환하고 다음 레이어에서 sigmod activation 함수를 이용하여 두 개의 클래스에 (결합 또는 비결합) 대한 결과를 얻는다.
import keras
from keras.models import Sequential
from keras.layers import Dense, Flatten
from keras.layers import Conv1D, MaxPooling1D
# options
num_classes = 2
conv1_hidden_units = 10
conv1_filter_size = 5
maxpool1_width = 4
# CNN model
model = Sequential()
model.add (Conv1D(conv1_hidden_units, kernel_size=(conv1_filter_size),
activation=’relu’, input_shape=(20, 4), padding=’same’))
model.add(MaxPooling1D(pool_size=maxpool1_width))
model.add(Flatten())
model.add(Dense(num_classes, activation=’sigmoid’))
# compile
model. compile(optimizer=’adam’,
loss=’categorical_crossentropy’,
metrics=[‘accuracy’])
model.summary()
Model: “sequential_30”
Layer (type) |
Output Shape |
Param # |
conv1d_30 (Conv1D) |
(None, 20, 10) |
210 |
max_pooling1d_28 (MaxPooling) |
(None, 5, 10) |
0 |
flatten_28 (Flatten) |
(None, 50) |
0 |
dense_28 (Dense) |
(None, 2) |
102 |
Total params: 312 |
|
|
위 모형의 summary 함수를 통한 각 레이어들에서 출력되는 shape를 앞서 소개한 코드의 설명과 비교할 수 있다. 이렇게 만들어진 CNN 모형을 실제 데이터를 이용해 학습하는 과정이 필요하다. 이를 데이터를 모형에 적합 (fit) 한다고 말하며 학습은 모형의 파라메터를 추정하는 과정이다. batch_size는 100으로 총 1600개 데이터를 100개씩 읽어 들여서 반복적으로 모형 학습에 사용하겠다는 것이고 epochs=20은 이런 과정을 20번 반복하겠다는 것이다.
# Training
batch_size = 100
epochs = 20
# Train
history = model.fit(X_train, y_train,
batch_size=batch_size,
epochs=epochs,
verbose=1,
validation_split=0.1)
위 코드를 실행하고 모형에 대한 정확도와 (accuracy) 손실 (loss) 정도를 그래프로 그려보면 다음과 같다. 여기서 정확도는 맞게 예측한 개수를 전체 예측 수로 나눈 비율이고 손실은 예측값과 실제값의 차이를 cross-entropy 형식으로 계산한 값이다.
import matplotlib.pyplot as plt
plt.figure()
plt.plot(history.history[‘val_loss’])
plt.plot(history.history[‘val_accuracy’])
plt.title(‘valid accuracy and valid loss’)
plt.ylabel(‘val accuracy and val loss’)
plt.xlabel(‘epoch’)
plt.legend([‘valid loss’, ‘valid accuracy’])
plt.show()
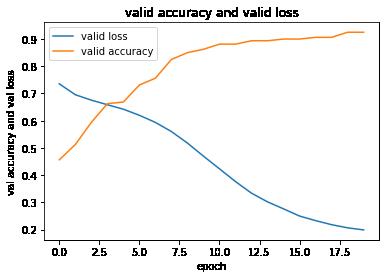
반복적으로 학습이 진행됨에 따라서 (Epoch이 증가함에 따라서) 모형이 서열의 결합 모티프를 찾아내는 정확도가 점점 증가하고 손실은 감소하는 것을 확인할 수 있다.
훈련된 CNN 모형을 이용한 예측
이렇게 훈련된 CNN 모형을 이용해서 앞서 분류해 둔 test 데이터 세트를 이용하여 모형의 예측 성능을 평가해 본다. model.evaluate 함수는 test 데이터 세트의 loss 값과 accuracy 값을 계산해 준다. 그러나 우리는 일반적으로 False positive (효과가 좋지 않지만 좋다고 판단) 비율이나 의료 분야의 False negative (암환자를 정상이라고 판단) 비율 등에 대한 관심이 더 높다. 이들은 각각 제1종 오류와 제2종 오류로 알려져 있으며 이들에 대한 정량 수치를 비교하여 조건에 맞는 딥러닝 모형을 평가하고 선별하여 사용할 수 있다.
from sklearn.metrics import confusion_matrix
score = model.evaluate(X_test, y_test, verbose=0)
print(‘Test loss:’, score[0])
print(‘Test accuracy:’, score[1])
Test loss: 0.1831807804107666
Test accuracy: 0.9399999976158142
predictions = model.predict_classes(X_test)
cm = confusion_matrix(y_test[:,1], predictions)
print(cm)
[[188 6]
[11 195]]
위와 같이 predict_classes 함수를 이용해서 분류를 수행할 수 있으며 confusion_matrix 함수로 분류표를 만들 수 있다. 위 결과에서는 전체 테스트 결과 중 모티프가 없는 서열을 없다고 예측한 개수가 188개, 모티프가 있는 서열에 대해서 있다고 예측한 경우가 195개 이다. 이러한 분류표를 이용해서 모형의 예측력을 평가하는 지표를 계산할 수 있다. 잘 알려진 평가 지표로 Accuracy, Recall, Precision, F1 등이 있으며 각 항목에 대한 의미와 계산법은 인터넷을 통해 어렵지 않게 찾아서 참고할 수 있다.
맺음말
본 기고에서는 생물학 서열을 데이터로 활용하여 딥러닝 특히 CNN 알고리즘을 수행하는 방법을 알아 보았다. python 기반의 keras를 사용하였으나 tensorflow나 pytorch와 같은 다양한 딥러닝 프레임워크를 사용할 수 있고 python 외에도 R이나 C++ 등의 언어를 사용하여 AI를 구현할 수 있다. 이렇게 쉽게 접할 수 있는 딥러닝 기술을 활용하기 위해서 가장 중요한 점은 라벨링된 서열 데이터를 대량으로 수집하고 자유자제로 다룰 수 있는 능력을 기르는 것이다. 대량의 서열 데이터를 다루는 관점에서는 python에서는 biopython, numpy, 그리고 pandas에 익숙해져야 하고 R의 경우는 tidyverse, ggplot2 등의 패키지와 함께 bioconductor 저장고를 활용하는 데 익숙해져야 하겠다. 데이터가 커질수록 계산량은 많아지게 되고 자연스럽게 GPU를 활용하여 계산을 수행할 수 있다. 딥러닝 개발 환경이 점점 복잡해지게 된다면 다른 전문가들이 기존에 구축해 놓은 딥러닝 개발 환경을 그대로 가져와 사용하기 위한 Docker (https://www.docker.com/)도 활용할 수 있을 것이다.
딥러닝을 위한 코딩뿐만 아니라 일반적인 프로그래밍을 수행할 때 가장 쉽게 실력을 쌓는 방법 중 하나는 다른 사람이 만들어 놓은 코드를 보고 따라 하는 것이다. 최근에는 참고할 수 있는 무료 동영상 강좌를 어렵지 않게 찾을 수 있게 되어서 이들을 참고하는 것도 하나의 방법이지만 필자가 추천하는 방법 중 하나는 github나 (https://github.com/) 딥러닝의 경우 kaggle (https://www.kaggle.com/) 사이트에서 필요한 코드를 직접 검색해서 활용하는 방법도 고려해 볼 수 있다. 물론 사용하는 패키지나 함수의 reference를 꼼꼼히 읽어 보는 것도 필요하다.
참고자료
[1] B. Alipanahi, A. Delong, M. T. Weirauch, and B. J. Frey, “Predicting the sequence specificities of DNA- and RNA-binding proteins by deep learning,” Nat. Biotechnol., vol. 33, no. 8, pp. 831–838, 2015.
[2] Z. Shen, W. Bao, and D. S. Huang, “Recurrent Neural Network for Predicting Transcription Factor Binding Sites,” Sci. Rep., vol. 8, no. 1, pp. 1–10, 2018.
[3] “https://www.python.org/downloads/release/python-377/.” [Online]. Available: https://www.python.org/downloads/release/python-377/.
[4] “https://www.anaconda.com/products/individual.” .
[5] “http://image-net.org/index.” [Online]. Available: http://image-net.org/index.
[6] C. E. Lawrence, S. F. Altschul, M. S. Boguski, J. S. Liu, A. F. Neuwald, and J. C. Wootton, “Detecting subtle sequence signals: A gibbs sampling strategy for multiple alignment,” Science (80-. )., vol. 262, no. 5131, pp. 208–214, 1993.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update