| 단백질공학을 이용한 인공금속효소 (Artificial Metalloenzyme) 연구 동향 | |
|---|---|
| Date 2020-07-30 18:25:38 |
|

교수
덕성여자대학교 과학기술학부 바이오공학 전공
junehpark@duksung.ac.kr
1. 인공금속효소
인공금속효소 (Artificial Metalloenzyme, ArM)는 전이금속촉매와 효소의 매력적인 특징을 동시에 가지고 있다. 효율적이고 선택성 있는 비(非)자연계 반응을 가능하게 한다. 효소를 이용한 비자연계 반응에 대한 요구가 증가함에 따라 ArM의 수요 역시 증가하고 있다. ArM을 만들기 위해 단백질 스캐폴드 (Scaffold)에 전이금속촉매를 결합하는 것은 1970년대 후반 처음 연구되었다 [1,2]. 꽤 오랜 시간 동안 유기금속합성과 단백질공학의 기술적 한계로 극히 제한적이고 부정적이었지만, 두 분야의 기술이 비약적으로 발달함에 따라 다시금 흥미를 불러일으켰다. 연구자들을 가장 흥미롭게 하는 것은 단백질 스캐폴드의 개량을 바탕으로 ArM의 성능을 향상시킬 수 있다는 것이다. 이는 기존 자연계 효소에 대해 응용해왔던 것과 유사한 방법으로 ArM 또한 개선이 가능하며, 실제 응용 가능한 시스템에 적용할 수도 있다.
새로운 ArM을 제조할 때 전이금속촉매를 단백질 내부에 잘 위치시키는 것은 전이금속촉매의 제 2 배위 구체 (second coordination sphere) 상호 작용을 올바르게 유도하여 목표한 반응을 갖도록 한다. 전이금속촉매를 단백질 내부에 잘 위치시키고 결합하는 것을 앵커링 (Anchoring) 이라고 하고 흔히 4가지 전략으로 분류 할 수 있다 (그림 1). 공유 (covalent), 초분자 (supramolecular), 배위 (dative), 그리고 금속 치환 앵커링 전략이다. 공유 앵커링은 비가역적인 반응으로 이루어지며 결합수율이 높다 [3]. 예를 들어 시스테인 잔기나 활성화된 잔기와의 친핵성 치환, 이황화결합, 그리고 비천연 아미노산으로 치환된 잔기와 azide/alkyne의 첨가환화 결합을 포함한다 [4]. 초분자 앵커링은 비공유 inhibitor, cofactor 또는 기질 등의 높은 친화성을 이용한다. 배위 앵커링은 친핵성 아미노산 잔기 (His, Cys, Glu, Asp, Ser 등)와 배위 불포화 금속간의 배위 결합에 의존한다. 이 전략은 공유나 초분자 앵커링 전략을 보완 하기도 한다. 금속 치환은 천연금속효소의 이미 존재하는 금속 결합 부위와 전이금속의 반응성을 이용한다. 주로 cytochrome이나 myoglobin 계열의 효소를 단백질 스캐폴드로 사용할 때 이용하는 전략이다 [5]. 이러한 전략들을 바탕으로 전이금속촉매를 localization하여 새로운 ArM을 만들지만 초기 선택성이나 효율은 기대에 못 미치는 경우가 많다. 물론 불필요한 cofactor의 제거나 정제 같은 처리 과정으로 성능을 향상 시킬 수는 있지만, 단백질공학적 접근을 통한 근본적 진화가 필요하다. 필자는 University of Chicago에서 postdoctoral scholar로 근무할 때 Jared C. Lewis 교수 (現, Indiana University)와 함께 인공금속효소의 대한 연구를 시작하였다.
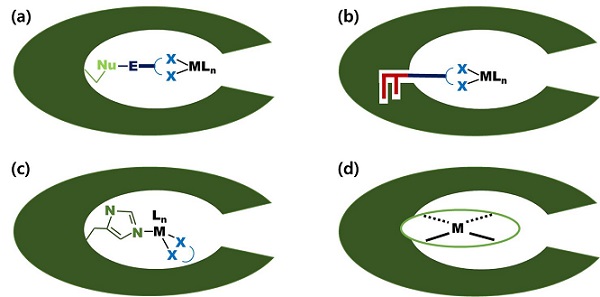
그림 1. 단 백질 스캐폴드에 금속촉매를 고정시키는 4가지 앵커링 전략 (a) 공유, (b) 초분자, (c) 배위, 그리고 (d) 금속치환.
2. 단백질 공학을 이용한 ArM의 개량
2.1. Random mutation의 적용
단백질공학의 발전은 효소 개량에 큰 디딤돌이 되었고 이 기술들을 ArM 효율 향상에 적용시킬 수 있다. 그러나 일반효소 개량과는 달리 ArM 시스템으로 적용은 좀 더 신중해야 한다. 단백질 스캐폴드의 구조를 알고 있다고 하더라도 전이금속촉매와 결합된 형태의 구조를 완벽히 예측 하기란 쉽지 않다. 금속치환으로 만들어진 ArM의 경우는 비교적 구조 정보가 정확해서 유용하게 이용된다. 스캐폴드 단백질이 Streptavidin, cyt cb562 및 미오글로빈인 경우에 구조-유도표적 돌연변이를 통해 선택성이 고도로 향상된 ArM의 개량이 보고된 바 있다. 특히, California Institute of Technology 의 Francis Arnold 교수의 연구실에서 cytochrome 계열의 heme-binding 단백질을 스캐폴드로 하여 heme의 modification 혹은 binding site의 변이를 통한 촉매 능력 향상에 대한 연구가 매우 활발히 진행 되고 있다 [6-8].
공유 앵커링 전략을 사용한 ArM의 경우, 유연성으로 인한 전이금속촉매의 활발한 움직임이 정확한 구조 모델 형성을 방해하고 결정구조 자체를 만들기 어렵다. 현재까지 결정 구조만 이용해서 보고된 ArM은 없다 (각각의 구조를 만들어 computational 모델링으로 구현한 구조는 보고되었다). 구조 정보가 부족할 경우 자연계 효소에 적용할 때처럼 Random mutation을 적용할 수 있다. 필자가 연구하는 ArM이 좋은 예시이다. Dirhoduim cofactor는 styrene 과 diazo 기질의 cyclopropanation 반응을 진행시킨다. 이 전이금속촉매를 공유 앵커링 방법을 이용 선택성을 가진 ArM으로 만들기 위해 Pyrococcus furiosus prolyl oligopeptidase (pfu POP)를 단백질 스캐폴드로 선정하였다. 스캐폴드의 활성 잔기세린을 비천연 아미노산인 azido 페닐알라닌 잔기로 대체하였고 SPAAC (Strain-promoted azide-alkyne cycloaddition) 결합을 통해 연결시켜 ArM으로 탄생시켰다 (그림 2a) [9]. 앞에서 언급한 스캐폴드들과는 달리 Pfu POP의 X-ray 결정구조는 연구 초기에 보고된 바가 없었다. 초기 반응성을 확인 후 활성부위라고 예측되는 스캐폴드의 β-프로펠러 도메인 부분을 error-prone PCR을 사용하여 변이 라이브러리를 만들었다. 라이브러리당 4~5개의 변이율을 유지하며 스크리닝을 진행하였고 3세대를 거쳐 높은 수준의 ArM을 얻었다 (그림 2b). 최종 변이주인 3-VRVH는 92% e.e. (초기 0-ZA4는 38% e.e)에 도달하였다 [10]. 뿐만 아니라, 최종 수율과 시간당 전환율도 선택성과 함께 증가하였다. 3-VRVH는 styrene moiety 와 diazo moiety 의 cyclopropanation반응도 고선택성 (86% e.e. 이상)을 보이며 반응 범위가 넓다는 것을 증명했다. 또한, 세대가 지날수록 단계별 선택성의 증가는 N-H, S-H, Si-H insertion을 포함한 다른 dirhodium 반응에도 적용되었다. 이 반응들 역시 세대별로 선택성이 증가하였고, 이는 한 가지 반응으로 개량된 ArM이 다른 반응 혹은 다른 기질의 반응에도 사용 가능한 범용적 촉매로서의 가능성을 보여준 예시라 본다.
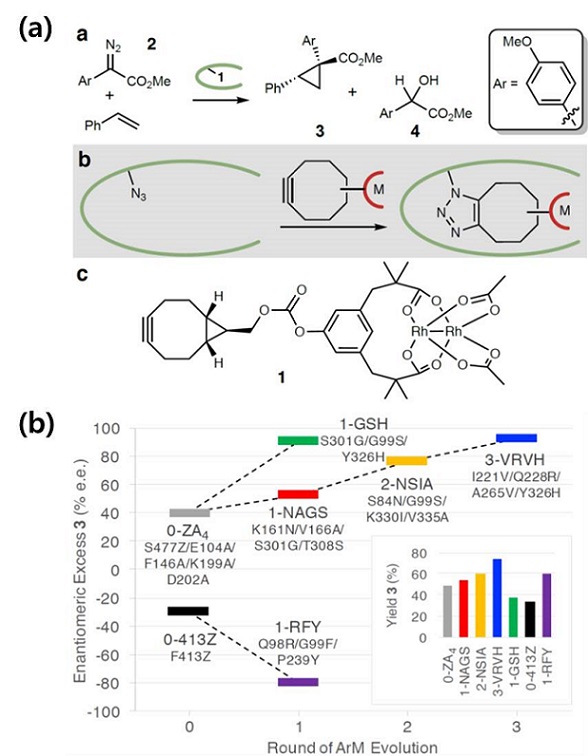
그림 2. (a) Dirhoduim ArM의 반응 및 앵커링, 금속촉매구조, (b) 개량된 ArM의 선택성 및 수율.
변이의 분석을 위해 세대별 reverse mutation을 통하여 key 변이를 찾아내었다. 각각의 세대에서 선택성에 major한 영향을 주는 변이를 selection하여 1-GSH를 제작 하였다. 선택성은 3-VRVH 정도로 높았지만 수율은 오히려 개량전인 0-ZA4 보다 낮은 수준을 보였다. 총 12개의 변이 중 3개의 선택성 dependent 한 변이는 수율에는 전혀 영향을 미치지 않는 변이로 확인 되었다. 또한 스캐폴드 단백질의 구조로 추후에 확인을 해보니 두 개의 변이는 금속촉매와 닿을 수 있는 부위에, 나머지 한 개의 변이는 외부 표면에 위치하는 것을 확인하였다. 이는 non-active site의 변이들 즉, 제 2배위 구체에 존재하지 않는 잔기들이 선택성과 수율에 영향을 준다는 것을 의미한다. 자연계의 효소들도 활성부위와 먼 거리의 변이가 구조 변형을 통해 활성에 영향을 주는 것처럼, ArM의 경우에도 동일한 현상이 적용된다는 것을 확인하였다.
2.2. 맞춤형 스크리닝 시스템의 필요성
랜덤 뮤테이션의 불확실성을 감안하고 볼 때, 비교적 적은 세대의 진화와 라이브러리 확인 수 (1세대 96개, 2세대 48개, 3세대 576개)의 판별로, 놀랍도록 향상된 개량형 dirhoduim ArM을 얻었다. 하지만 이를 가능하게 하기 위해서는 까다로운 준비 과정이 필요하다. 가장 중요한 준비 과정은 ArM을 선택적으로 선정할 수 있는 맞춤형 스크리닝 시스템의 구축이다. 전이금속촉매는 보통 bulky한 구조를 지니고 있다. 클릭 케미스트리를 이용해서 원하는 위치에 앵커링을 하여도 제조 과정에서 남은 전이금속촉매는 효소 표면에 흡착하게 되고 ArM에 비해 훨씬 빠른 속도로 반응을 하게 된다. 이는 선별과정에서 제조된 ArM이 얼마나 선택성이 변했는지 혹은 효율이 증가하였는지에 대한 기준점을 무용지물화 한다. 따라서 스캐폴드와 결합하지 않고 표면에 남아있는 전이금속촉매만을 선택적으로 washing out하는 과정이 필요하다. Dirhoduim ArM의 경우, 초기에는 azide 기능이 활성화 된 agarose resin을 첨가하여 남은 전이금속촉매를 binding하고 ArM이 있는 상등액만을 이용하여 스크리닝을 진행하였다. 이 시스템은 효과가 좋지만 비효율적인 부분이 존재하고 많은 시간을 최적화하는 데 투입해야 한다. 첨가하는 Agarose resin의 양, 금속촉매와 binding 시간, binding 하는 mechanical 방법 등의 최적화가 필요하고 무엇보다 azide기능기를 갖는 resin을 만드는 과정이 비싸고 힘든 작업이다. 따라서 보다 효율적이고 빠른 스크리닝 과정이 필요하다. 구하기 쉽고 비교적 저렴한 Ni-NTA resin과 필터가 달린 96 well 플레이트의 도입은 보다 빠르고 정확한 스크리닝을 가능하게 한다. 필터 플레이트에 Ni-NTA resin을 투여하고 His-tag 도입 발현이 된 단백질 스캐폴드를 결합시킨다. 그 후, 전이금속촉매를 충분히 binding한 다음 buffer로 씻어내고 resin에 붙어있는 ArM 상태 그대로 스크리닝 반응을 진행한다. 변화된 방법은 훨씬 정확하고 빠르게 선별이 가능하며, ArM이 고정화된 상태이기 때문에 같은 라이브러리를 가지고 다른 반응에 대한 재스크리닝이 가능하다는 장점도 가진다. 이 효율적인 프로토콜 플랫폼은 dirhoduim ArM뿐 아니라 어떤 ArM을 스크리닝에도 적용할 수 있다.
2.3. 스캐폴드 단백질의 구조 정보를 이용한 semi-rational design
Dirhoduim ArM의 경우, 앞에서 Random mutation으로 개량한 3-VRVH를 얻을 때쯤 단백질 스캐폴드의 구조를 얻을 수 있었다. 이 구조를 바탕으로 cyclopropanation 반응의 반대되는 선택성을 가질 수 있는 위치를 탐색하였고, 그 결과 개량 가능성을 보이는 F413Z (-30% e.e.)를 얻었다. Pfu POP의 결정 구조는 F413Z의 선택성을 향상시킬 수 있는 특정 잔기를 선정해주지는 않지만, 단백질 내부 방향으로 향해 있는 잔기가 무엇인지 명확하게 해주었다. 전이금속촉매 방향으로 돌출되어있는 아미노산 잔기들은 선택성을 향상시킬 가능성이 있는 부위 즉, 활성 부위라고 예측했고, 이들을 변이시켜 신속하게 개량된 ArM을 얻고자 했다. 25개의 활성 부위 잔기 중 평균 2~3개의 NDT 코돈을 함유하는 combinatorial codon mutagenesis (CCM) 라이브러리를 제작하였다 [11,12]. NDT 코돈의 사용은 정지 코돈 돌연변이의 가능성을 제거하고, CCM 방법은 25개의 위치에 선택적으로 변이를 가능하게 한다. 단 한번의 라이브러리 스크리닝을 통해 -80% e.e.의 선택성을 가지는 1-RFY 를 얻었다. 이러한 예처럼, 구조 정보를 바탕으로 ArM의 개량부위를 선정하고 전략적 라이브러리 제작 및 스크리닝을 통해 향상된 개량주를 얻는 방식이 좀 더 효율적이라고 생각된다.
3. 결론 및 전망
ArM에 대한 연구분야를 정리하면 크게 다음과 같이 나눌 수 있다. 목표하는 반응의 전이금속복합체 탐색, scaffold 단백질 탐색 및 구조분석, 금속과 단백질 조합방법 모색, 반응 스크리닝법 구축, 분자동역학을 이용한 구조 분석, 움직임 모식화, 반응 매커니즘 이해이다 (그림 3). 하나하나의 영역들이 큰 연구 주제이지만 기존의 연구들과 잘 조화롭게 어울려 빠르게 성장 중이다. 최근에는 효소 분류 체계 (E.C number 분류)처럼 현재까지 개발된 ArM들에 대해서 반응별 분류도 시도되었다[13]. Reduction, C−C Bond Formation, Oxygen Insertion, 그리고 hydration의 반응으로 분류를 하였다 (표 1). 기존 효소반응과는 다른 기준으로 분류되고 있으나 언젠가는 모든 촉매반응들이 더 큰 틀 안에서 같이 분류되는 날이 오지 않을까 생각해 본다.
그림 3. 인공금속효소 연구 분야.
표 1. 인공금속효소의 Reaction Scope 분류 표
Reduction Chemistry |
C-C Bond Formation |
Oxygen Insertion Chemistry |
Hydrolytic Cleavage |
Hydrogenation |
Allylic Alkylation |
Peroxidation or Oxygenation |
Ligation Reaction |
Hydrogen Generation: |
Suzuki Cross-Coupling |
Sulfoxidation |
Hydration of C=C and |
Transfer Hydrogenation |
Heck Reaction |
Alcohol Oxidation |
Lyases |
Hydroformylation |
C-H Activation |
Epoxidation |
|
Small Molecule Reductases |
Olefin Metathesis |
Dihydroxylation |
|
|
Cyclopropanation |
Cco Oxidase |
|
Polymerization (Non - ROMP) |
C-H Oxidation |
||
Diels-Alder Reaction |
Protein and Nucleic Acid Cleavage |
||
Huisgen [3 + 2] Cycloaddition |
|
||
Friedel-Crafts Reaction |
이 글에서 필자는 단백질공학을 이용한 ArM의 개량에 대한 연구동향을 소개하였다. Random mutation의 활용은 상세한 구조 정보가 없더라도 ArM의 개량에 유용하게 적용될 수 있음을 확인하였다. 이 방법은 구조 분석 접근으로는 탐색 불가한 유용한 변이를 도출하기도 하지만 때때로 시간이 많이 걸린다. 구조분석, 분자모델링, 분자동역학등과 같은 rational design approach로 기틀을 잡고 타겟형 라이브러리의 제조와 최적화된 스크리닝 프로토콜을 활용한다면 효율적이고 안정정인 ArM을 보다 쉽게 개발하고 지금까지 한계를 보여왔던 선택성 있는 비자연계 반응의 다양화를 가능하게 하리라 본다.
참고문헌
[1] Yamamura, K., Kaiser, E. T., Studies on the Oxidase Activity of Copper(Ii) Carboxypeptidase A. J. Chem. Soc., Chem. Commun. 1976, 830-831.
[2] Wilson, M. E., Whitesides, G. M., Conversion of a Protein to a Homogeneous Asymmetric Hydrogenation Catalyst by Site-Specific Modification with a Diphosphinerhodium(I) Moiety. J. Am. Chem. Soc. 1978, 100, 306-307.
[3] Hermanson, G. T. Bioconjugate Techniques, 3rd ed., Elsevier: Oxford, 2013.
[4] Yang, H., Srivastava, P., Zhang, C., Lewis, J. C., A General Method for Artificial Metalloenzyme Formation through Strain-Promoted Azide-Alkyne Cycloaddition. ChemBioChem 2014, 15, 223-227.
[5] Jing, Q., Okrasa, K., Kazlauskas, R. J., Manganese-Substituted Alpha-Carbonic Anhydrase as an Enantioselective Peroxidase. Top. Organomet. Chem. 2009, 25, 45-61.
[6] Coelho, P. S., Brustad, E. M., Kannan, A., Arnold, F. H., Olefin Cyclopropanation Via Carbene Transfer Catalyzed by Engineered Cytochrome P450 Enzymes. Science 2013, 339, 307-310.
[7] Renata, H., Wang, Z. J., Arnold, F. H., Expanding the Enzyme Universe: Accessing Non-Natural Reactions by Mechanism-Guided Directed Evolution. Angew. Chem., Int. Ed. 2015, 54, 3351-3367.
[8] Bordeaux, M., Tyagi, V., Fasan, R., Highly Diastereoselective and Enantioselective Olefin Cyclopropanation Using Engineered Myoglobin-Based Catalysts. Angew. Chem., Int. Ed. 2015, 54, 1744-1748.
[9] Srivastava, P., Yang, H., Ellis-Guardiola, K., Lewis, J. C., Engineering a dirhodium artificial metalloenzyme for selective olefin cyclopropanation. Nat. Commun. 2015, 6, 7789
[10] Yang, H., Swartz, A., Park, H. J., Srivastava, P., Ellis-Guardiola, K., Upp, D. M., Lee, G., Belsare, K., Gu, Y., Zhang, Moellering, R. E., Lewis, J. C., Evolving artificial metalloenzymes via random mutagenesis. Nature Chem. 2018, 10, 318–324
[11] Harris, M. N., Madura, J. D., Ming, L.-J., Harwood, V. J., Kinetic and mechanistic studies of prolyl oligopeptidase from the hyperthermophile Pyrococcus furiosus. J. Biol. Chem. 2001, 276, 19310–19317
[12] Belsare, K. D., Andorfer, M. C., Cardenas, F. C., Chael, J. R., Park, H. J., Lewis, J. C., A Simple Combinatorial Codon Mutagenesis Method for Targeted Protein Engineering, ACS syn. Bio. 2017, 6, 416-420
[13] Schwizer, F., Okamoto, Y., Heinisch, T., Gu, Y., Pellizzoni, M. M., Lebrun, V., Reuter, R., Köhler, V., Lewis, J. C., Ward, T. R., Artificial Metalloenzymes: Reaction Scope and Optimization Strategies. Chem Rev. 2018, 118, 142-231
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update