| DNA 이중나선을 이용한 단분자력 측정과 세포역학 연구 | |
|---|---|
| Date 2020-10-21 01:57:36 |
|

연구원 / 교수
인천대학교 생명공학부
kyungah220@gmail.com / introbc@gmail.com
1. 서론
세포는 살아있는 고감도 센서로서, 외부로부터 들어오는 자극을 지속적으로 인지하고, 상황에 맞게 반응한다. 최근 연구 결과, 생화학적 자극 (biochemical cue) 뿐만 아니라, 기계적 자극 (mechanical cue) 역시 세포활동에 큰 영향을 미치는 것으로 밝혀졌다. 즉, 세포 외부의 기계적 자극이 세포 내에서 생화학적 신호로 변환되어, 세포의 부착, 퍼짐, 움직임 등 세포활동에 영향을 미치는 것으로, 이러한 과정을 기계적 신호전달(mechano-transduction)이라고 한다 [1]. 어떻게 세포에서 외부의 기계적 자극이 인지되고, 이것을 생화학적 신호로 변환시키는지에 대한 많은 연구가 수행되어 왔다 [2,3]. 그 결과, 세포막 수용체 (receptor)와 세포 밖에 존재하는 리간드 (ligand)가 결합을 형성하고, 이것을 매개로 외부 기계적 힘을 인지하거나, 거꾸로 세포 안에서 밖으로 힘을 전달하는 것이 밝혀졌다. 이러한 현상의 한 예로, 세포 주위 세포외기질(Extracellular matrix, ECM)과 세포막 수용체 (receptor)인 인테그린 (integrin)의 결합과 힘의 전달과정을 들 수 있다 (그림 1A). 이 결합에 물리적·기계적 힘이 가해지게 되면 휴면 상태 (inactive form)인 접힘 구조 (folded structure)의 인테그린이, 풀림 상태(unfolded state) 로 구조변화 (conformational change) 를 일으켜 활성화되어, 리간드와 더욱 강한 결합 (bond)을 이루게 된다 (그림 1B). 이후, 인테그린-리간드 결합은 빈큘린 (vinculin), 탈린 (talin) 등 연결 단백질 (adaptor protein)들을 통해 세포 골격 (actin cytoskeleton)과 연결되고, 미오신 (myosin)의 작용에 의해 발생하는 세포 견인력 (traction force)을 외부로 전달한다. 결과적으로 인테그린-리간드 결합에 가해진 외부 물리력과 내부 물리력이 상호작용하여, 작게는 세포 부착, 퍼짐, 움직임에 영향을 미치고, 크게는 세포의 분화, 생장, 사멸 등 세포의 운명을 결정짓게 된다. 그러므로 비정상적인 기계적 신호전달은 세포의 기능 유지를 저해하고, 암과 같은 심각한 질병을 일으키기도 한다. 이 밖에 기계민감성 수용체 (mechano-sensitive receptor)와 다른 세포에서 발현하는 리간드의 결합을 통한 세포와 세포 사이의 신호전달 과정에서, 수용체에 전달되는 물리적 힘은 신호전달과정을 진행시키거나 막는 스위치 역할을 한다. 따라서, 기계민감성 수용체의 활성화에 필요한 힘의 크기, 수용체-리간드 결합에 전달되는 힘의 크기를 측정함으로써, 기계적 자극에 따른 세포 반응을 연구하려는 시도가 이루어지고 있으며, 이 과정에서 단분자 수준 (single molecule level)에서 수용체에 가해진 힘을 측정하는 기술들이 활용되고 있다. 본 기고에서는, 많은 단분자 측정기술 (single molecule force spectroscopy (SMFS)) 중, DNA를 응용한 단분자력 측정 기술인 tension gauge tether (TGT)의 원리와 활용에 대하여 알아보고자 한다.
그림 1. 외부 기계적 자극이 세포 내 생화학적 신호로 바뀌는 기계적 신호전달과정. (A) 세포막 단백질인 인테그린과 세포외기질 분자와의 결합을 통한 외부 자극인지. (B) 외부 힘에 의해 활성화된 인테그린의 변화와 이후 진행되는 세포 내 신호전달.
2. 본론
2.1 기존 세포의 기계적 힘 (mechanical force) 측정기술들과 한계
세포 견인력 (traction force) 측정은 세포가 접하고 있는 기질표면에 가하는 힘을 측정하는 방법으로 기계생물학 (mechanobiology) 연구에 널리 활용되어오고 있다. 대표적인 방법으로, 견인력 현미경 (Traction force microscopy) 과 마 이크로 기둥 배열 (micropillar array)이 있다. 견인력 현미경 방법은 형광 비드 (bead)가 들어있는 하이드로젤 (hydrogel) 에 세포가 부착한 후, 세포가 표면에 가하는 물리력을 젤의 변형에 따른 형광 비드의 이동을 계산하여 측정하는 방법이 다 [4]. 마이크로 기둥 배열 (Micropillar array) 방법은 마이크로 미터 크기의 탄성 중합체로 이루어진 마이크로 기둥 위 에 세포를 부착시킨 후, 세포의 견인력에 의해 생기는 기둥의 구부러짐 정도를 계산하여 세포 수준의 견인력을 측정하 는 방법이다 [5]. 하지만 이 방법들의 힘 분해능 (force resolution)은 nN 크기로, 분자수준의 물리력 측정에는 적합하지 않다. 이러한 문제를 해결하기 위해, 단분자 측정법들이 개발되었고. 보다 pN 크기의 높은 분해능과 정밀한 조절 기능 으로 인해, 세포 수용체에 가해진 분자수준의 힘을 측정 및 조절이 가능하게 되었다 [6]. 단분자측정방법의 예로는, 캔 틸레버 (cantilever)를 이용해 세포의 위상과 수용체 -리간드 결합력을 측정하는 원자현미경 (atomic force microscopy (AFM)) [7], 그리고 빛이나 자기력을 활용하여 수용체 -리간드 결합을 측정하는 광 핀셋 (optical tweezer)/자기력 핀셋 (magnetic tweezer) 기술 등이 있다. 이 기술들은 공통적으로 pN 단위의 물리력 측정이 가능해, 생물물리 연구에 널리 활용되어 왔지만, ‘한 번에 하나 측정 ’ 원리 상, 세포의 표면에 발현하는 다수의 수용체와 이들과 결합하는 리간드 사이 의 결합력 혹은 수용체 -리간드 결합에 가해진 힘의 측정이 제한되는 단점이 있다. 이러한 한계점을 극복하기 위하여, 최근 많은 수의 결합에 가해진 물리력을 한 번에 측정할 수 있는 기술의 필요성이 대두되었다.
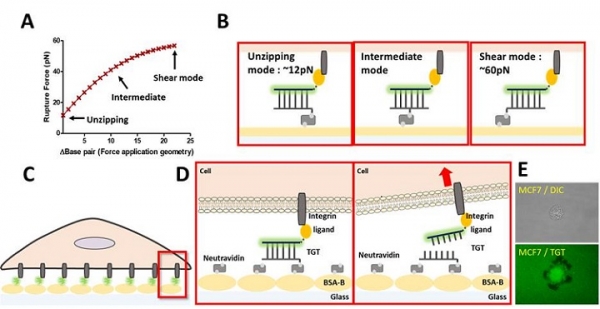
그림 2. DNA 이중나선을 활용한 단분자력 센서 개발. (A) 힘이 가해진 두 지점 사이 거리에 따른 DNA 이중 가닥 분리에 필요한 힘의 크기 변화와 (B) 분리 모드. (C) Tension gauge tether (TGT)를 매개로 한 세포 부착. (D) TGT 분자력 측정원리. (E) 단분자력의 시각적 맵핑.
2.2 DNA 이중나선을 활용한 단분자력 센서 개발
2.2.1 DNA 이중나선을 활용한 단분자력 측정 원리
DNA는 뉴클레오타이드로 구성되어 있는 긴 고분자 사슬이다. DNA의 염기는 다른 상보적인 염기와 수소 결합을 통해 쌍을 이루어, DNA 이중 나선구조를 형성하는 데 큰 역할을 한다. 예를 들어, 구아닌 (guanine)은 사이토신 (cytosine) 과 세 개의 수소결합을 통해, 아데닌 (adenine)과 티민 (thymine)은 두 개의 수소 결합을 형성하며 쌍을 이룬다. 앞서 설명한 단분자 측정기술을 이용하여 DNA 이중 가닥을 풀기 위해 필요한 힘 (tension tolerance, Ttol)은 힘이 가해진 위치에 따라 4 ~ 60 pN의 장력이 필요한 것이 de Gennes의 이론과 자기성 핀셋 실험을 통해 증명되었다 (그림 2A) [8,9]. 이때 DNA 이중 가닥을 분리를 위한 Ttol (4 ~ 60 pN)는 단분자 수준에서 기계적 신호변환을 일으키는 일반적인 장력의 크기와 일치한다. 따라서, DNA 의 화학적 구조와 물리적인 특성은 DNA를 기반으로 세포에서 일어나는 힘을 측정하기 위한 단분자력 센서 개발에 이론적 토대가 된다 (그림 2B).
2.2.2 DNA 이중나선 기반 단분자력 센서 개발
단분자력 센서가 세포에 가해진 장력 (tension)을 성공적으로 측정하기 위해서는 다음 기준들을 충족하여야 한다. 1) pN 수준의 장력 측정, 2) 표적 분자에 대한 높은 특이적 결합, 3) 측정 방식의 용이성, 4) 고속 대량 스크리닝.
존스홉킨스대학 하택집 교수 연구팀은 DNA를 활용하여 위 4개 기준을 모두 충족하는 DNA 단분자력 센서 (tension gauge tether, TGT) 를 개발하였다 (그림 2C-E) [10]. TGT 는 DNA 이중가닥의 Ttol 보다 큰 힘이 가해질 경우, 비가역적으 로 분리되어, DNA의 분리 유무를 확인하여 가해진 장력의 크기를 측정할 수 있도록 설계되었다. 또한, 화학적 변형 (chemical modification)을 통해, 윗가닥 (upper strand)의 한 쪽 끝에 특정 수용체 단백질과 결합 가능한 리간드를 붙여, 표적 수용체 -리간드 특이적 결합을 유도할 수 있다. 더불어, 윗가닥의 다른 부분에 형광 분자를 달아주어, 윗가닥이 세 포의 장력에 의해 뜯겨져 나갈 때, 형광의 손실을 유도하여, 결과적으로 세포 내 장력의 시공간적 변화 및 분포를 형광 이미지를 통해 쉽게 측정할 수 있다. 이후, 세포력 (cell force)을 시각화 맵핑 (mapping)하여 표현될 수 있다 (그림 2E). 마지막으로 기질 (substrate) 표면에 리간드가 결합된 TGT를 고루 분포시키고, 세포에서 발현하는 다수의 수용체와 동시 다발적 결합을 유도하여, 많은 수의 수용체 -리간드 결합에 가해진 장력의 변화를 한 번에 측정할 수 있다. 그 결과 세 포 활동에 따른 수용체 -리간드 결합에 가해지는 장력을 형광 신호를 통해 측정하고, 무수히 많은 형광신호의 분석을 통 하여, 세포가 경험하거나 발현하는 힘을 단분자 수준에서 세포수준까지 측정이 가능하게 되었다.
2.3 DNA 이중 나선을 활용한 세포 역학 연구의 응용
2.3.1 인테그린 활성화와 작용에 장력이 미치는 영향 측정
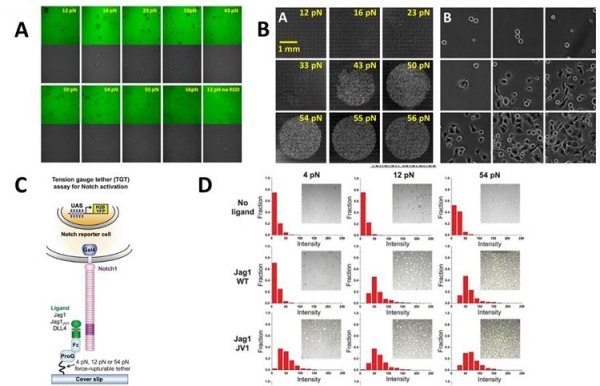
DNA 이중나선 기반 단분자력 센서 (TGT)는 인테그린이 세포외기질 분자와 결합하여 활성화되는 과정에서 가해진 장력이 미치는 영향을 측정하는 데 사용되었다 [10]. αv β3 인테그린과 결합 가능한 cyclic-RGDfK 리간드를 TGT의 윗 가닥에 공유결합을 통해 붙여 준 후, 세포에서 발현하는 인테그린과 리간드가 결합할 수 있게 디자인되었다 (그림 3A-B). TGT를 이용하여 세포 부착 시, 인테그린-리간드 결합에 가해지는 힘의 크기를 조절하여 분석한 결과, 약 40 pN 이상의 분자력이 가해질 경우에만 인테그린이 활성화되어, 안정적인 세포 부착으로 이어지는 것을 밝혔다 [10]. 또한, 이후 56 pN 이상의 Ttol를 갖는 TGT에서만 인테그린 -리간드 결합을 매개로 한 초점접합복합체 (focal adhesion complex) 형성이 유도되고, 더 나아가 미오신의 활성 시 액틴 -초점접합-인테그린 복합체에 전달되는 물리력이 56pN 이상임을 알 수 있었다 [11]. 이러한 특성은 다양한 세포에서 공통적으로 관찰되는 현상으로, 인테그린 활성화에 40pN 이상의 ‘보편 적’ 인테그린 장력이 필요함을 보여준다.
2.3.2 Notch 신호전달 활성화에 필요한 장력 측정
TGT는 Notch 신호 전달체계 활성화에 필요한 물리력 측정에도 사용되었다. 세포와 세포 사이 상호작용으로 일어나 는 Notch 신호전달체계는 Notch 수용체와 DLL (delta-like protein) 혹은 Jagged 등 리간드와 결합을 통해 시작된다. 이 과정에서 Notch 수용체에 pN 크기의 물리력이 가해지면 구조변화가 일어나게 되고, 막 주변에 존재하는 γ-secretase 효 소가 구조가 바뀐 수용체를 인지하여 특정 부분을 잘라주며 신호전달이 시작되게 된다. 각기 다른 Ttol를 갖는 TGT에 Notch 리간드를 결합한 후, Notch 신호의 활성화를 형광으로 확인할 수 있는 리포터세포 (Reporter cell)를 넣고 반응을 관찰한 결과, 4 ~ 12 pN의 장력으로도 충분히 Notch 신호 활성화를 할 수 있음을 밝혔으며, 후속 연구를 통하여 Notch 리간드의 종류와 결합 친화력 (binding affinity) 에 따라 Notch 활성화에 필요한 물리력이 달라짐을 확인할 수 있었다 (그 림 3C-D) [10,12,13].
그림 3. TGT 단분자력 센서의 활용 예시. (A-B) 정밀하게 조절된 인테그린 장력 (integrin tension)에 따른 세포 부착. (C-D) Notch 신호 활성화에 필요한 물리력 측정. 본 그림은 참고문헌 [10,13]에서 발췌함.
2.3.3 기타 응용 사례
기존 TGT에 쉽고 빠르게 다양한 종류의 리간드를 붙이기 위하여, protein G를 기반으로 한 TGT (proG-TGT) 가 개발 되었다 [14]. 이 방법은 리간드를 TGT에 직접적으로 공유결합을 통해 결합시키는 방법 대신, ProG를 붙여주고, 이것과 결합 가능한 Fc-tag을 가진 리간드를 선택적으로 붙일 수 있도록 고안되었다. 이 기술을 활용하여, E-cadherin을 통한 세포 부착 시, 12 pN 이하의 장력에서도 충분히 세포 부착이 될 수 있음을 밝혔으며, 내피세포 (endothelial cell) 에서 발현하는 P-selectin과 이와 결합하는 백혈구 (leukocyte)에서 발현하는 P-selectin glycoprotein ligand-1 (PSGL-1)의 결합으로 진행되는 혈관 내 백혈구 굴림 (rolling) 현상을 생물물리적으로 해석할 수 있었다 [15]. 이 밖에 TGT 시스템을 이용하여 암세포, 면역세포에서 일어나는 수용체를 매개로 진행되는 다양한 기계신호전달 현상에 대한 연구 역시 활발히 진행되 고 있다.
3. 결론 및 전망
물리적·기계적 자극이 세포에 미치는 영향을 DNA 이중나선을 활용하여 단분자 수준으로 측정하는 기술에 대하여 알아보았다. 본문에 소개된 내용 이외에도, 1) 수용체 특이적 활성에 따른 기계적 신호변환을 유도하기 위한 다양한 리 간드 결합, 2) DNA를 대체할 수 있는 펩타이드, 단백질, 폴리머 등을 활용하여, 더욱 다양한 범위의 힘의 측정, 3) 공간 분해능을 높이기 위해 초고해상도 현미경과 결합 등 기존 시스템을 향상시키기 위한 많은 연구가 진행되고 있다. 이 단 분자력 측정 기술의 활용으로 세포 내, 기계적신호전달 과정에 대한 더욱 폭 넓은 분자생물적 ·생물물리적 이해가 가능 하며, 해당분야의 학술적 발전에 큰 기여를 할 것으로 기대가 되고 있다. 나아가, 여러 가지 질병과 관련된 기계적 자극에 대한 이해를 더해 줌으로써 더욱 효과적인 치료법 및 진단법 개발에 대한 실마리를 제공할 것으로 예상된다.
참고문헌
1. Katsumi, A., et al., Integrins in mechanotransduction. Journal of Biological Chemistry, 2004. 279(13): p. 12001-12004.
2. Palazzo, A.F., et al., Localized stabilization of microtubules by integrin-and FAK-facilitated Rho signaling. Science, 2004. 303(5659):
p. 836-839.
3. Chicurel, M.E., C.S. Chen, and D.E. Ingber, Cellular control lies in the balance of forces. Current opinion in cell biology, 1998. 10(2):
p. 232-239.
4. Schwarz, U.S. and J.R. Soine, Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta, 2015. 1853(11 Pt B): p. 3095-104.
5. Rahmouni, S., et al., Hydrogel micropillars with integrin selective peptidomimetic functionalized nanopatterned tops: a new tool for the measurement of cell traction forces transmitted through alphavbeta3- or alpha5beta1-integrins. Adv Mater, 2013. 25(41): p. 5869-74.
6. Fuhrmann, A. and R. Ros, Single-molecule force spectroscopy: a method for quantitative analysis of ligand-receptor interactions. Nanomedicine, 2010. 5(4): p. 657-666.
7. Panorchan, P., et al., Single-molecule analysis of cadherin-mediated cell-cell adhesion. J Cell Sci, 2006. 119(Pt 1): p. 66-74.
8. de Gennes, P.-G., Maximum pull out force on DNA hybrids. arXiv preprint physics/0110011, 2001.
9. Hatch, K., et al., Demonstration that the shear force required to separate short double-stranded DNA does not increase significantly with sequence length for sequences longer than 25 base pairs. Physical Review E, 2008. 78(1): p. 011920.
10. Wang, X. and T. Ha, Defining Single Molecular Forces Required to Activate Integrin and Notch Signaling. Science, 2013. 340(6135): p. 991-994.
11. Wang, X., et al., Integrin molecular tension within motile focal adhesions. Biophysical journal, 2015. 109(11): p. 2259-2267.
12. Chowdhury, F., et al., Defining Single Molecular Forces Required for Notch Activation Using Nano Yoyo. Nano Letters, 2016. 16(6):
p. 3892-3897.
13. Luca, V.C., et al., Notch-Jagged Complex Structure Implicates a Catch Bond in Tuning Ligand Sensitivity. Science, 2017. 355: p. 1320-1324.
14. Wang, X., et al., Constructing Modular and Universal Single Molecule Tension Sensor Using Protein G to Study Mechano-Sensitive Receptors. Scientific Reports, 2016. 6: p. e21584.
15. Li, I.T.S., T. Ha, and Y.R. Chemla, Mapping Cell Surface Adhesion by Rotation Tracking and Adhesion Footprinting. Scientific Reports, 2017. 7: p. 44502.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update