| 합성생물학을 활용한 원위치(in situ) 장 마이크로바이옴 조절 기술 | |
|---|---|
| Date 2021-04-22 10:44:49 |
|

교수 / 박사후 연구원
인천대학교 생명공학부 / 인천대학교 생명공학부
sjang@inu.ac.kr / leehj8496@inu.ac.kr
1.원위치 장 마이크로바이옴 조절 기술의 필요성
마이크로바이옴 (microbiome)은 특정 서식지에 존재하는 모든 미생물들과 그로부터 유래한 유전물질을 통틀어 부르는 용어다. 지구 어디에나 미생물이 존재하며, 인체 내에도 인간의 세포 개수와 비슷하거나 오히려 더 많은 수의 미생물이 공존하고 있다[1]. 특히, 인간의 장(腸, gut)에는 굉장히 많은 수의 미생물이 서식하고 있으며, 이 미생물들이 구성하는 장 마이크로바이옴은 면역 조절, 물질 대사, 병원균과의 경쟁 등 다양한 메커니즘을 통해 인간의 건강에 지대한 영향을 미친다 [2]. 물론, 미생물이 인간의 건강 또는 질병 상태에 영향을 미치는 가장 잘 알려진 예는 병원균들에 의한 직접적인 감염일 것이다. 하지만, 최근에는 암, 대사질환, 자가면역질환, 심지어 신경퇴행성질환에 이르기까지 매우 다양한 질병들이 장 마이크로바이옴의 상태나 조성과 상관관계가 있다는 연구 결과들이 보고되고 있다.
마이크로바이옴 조성을 조절하여 이들이 인체에 미치는 영향을 변화시킴으로써 질병을 치료하는 몇 가지 방법들이 알려져 있지만, 대부분의 방법들은 특이성 (specificity)이 떨어지거나 조절 가능한 미생물종의 범위가 제한적이다. 먼저, 박테리아 감염을 제어하기 위해서 가장 전통적이고 광범위하게 사용되는 방법은 항생제 투여다. 하지만 항생제는 특이성이 매우 떨어지는 방법으로서 병원성 박테리아뿐만 아니라 장 마이크로바이옴을 구성하는 다양한 미생물들에 광범위하게 작용한다. 프리바이오틱스(prebiotics)는 식품에 첨가되는 물질로써 특정 미생물종의 성장을 돕고 그에 따라 질병을 예방하거나 증상을 완화한다고 알려져 있다. 예를 들면, 식이 섬유를 섭취하면 장 마이크로바이옴을 조절하여 제2형 당뇨병을 완화할 수 있다는 것이 밝혀진 바 있다[3]. 하지만, 프리바이오틱스로 성장을 조절할 수 있는 미생물 종의 범위가 제한적이며, 프리바이오틱스가 정확히 어떤 미생물종에 어떤 영향을 미치는지는 불분명하다. 장 마이크로바이옴을 조절하는 좀 더 급진적인 방법으로 분변 미생물총 이식 (fecal microbiota transplantation, FMT)이 있다. 예를 들어, 건강한 사람의 분변으로부터 분리해낸 마이크로바이옴을 환자에 이식하면 만성적이고 재발하는 Clostridioides difficile 감염을 치료할 수 있다 [4]. 하지만, 이 방법은 마이크로바이옴을 통째로 이식하는 방법이므로 조성을 정교하게 조절할 수 없으며, 공여자 (donor)에 따라 조성이 달라지므로 재현성이 떨어지고, 마이크로바이옴에 포함된 병원균이 함께 이식될 위험성이 있다.
현재의 마이크로바이옴 조절 기술들은 질병 치료뿐만 아니라 장 마이크로바이옴의 구성과 인간의 건강 사이의 인과관계를 밝혀내는 기초 연구에 활용되기에도 제한점이 많다. 단순히 상관관계를 규명하는 것을 넘어서 마이크로바이옴 조성과 인간의 건강 사이의 인과관계를 밝혀내려면 독립변인(마이크로바이옴 조성)을 조절하고 그에 따른 효과(건강상태)를 측정하는 실험을 수행해야만 한다. 하지만, 앞서 설명한 바와 같이 현재의 마이크로바이옴 조성을 조절하는 기술들은 특이성이나 조절 가능한 미생물종의 범위가 제한적이다. 마이크로바이옴을 대표하는 몇 가지 미생물 종을 개별적으로 배양한 뒤에 정해진 비율로 다시 섞어 인공 마이크로바이옴을 제작하는 bottom-up 접근법들은 [5] 마이크로바이옴의 복잡성을 단순화해줄 수 있지만, 자연 상태의 마이크로바이옴이 지니는 풍부한 다양성과 숙주와의 상호작용을 그대로 반영하기는 매우 어렵다. 마이크로바이옴 분야가 인과관계를 밝혀내고 발병 기전을 규명하는 수준으로 발전해가고 있긴 하지만 [6], 마이크로바이옴의 다양성을 보존하면서도 그 안에 포함된 개별 종을 정밀하게 조절할 수 있는 실험 기술은 여전히 부족한 실정이다.
마이크로바이옴은 매우 다양한 미생물들로 구성되어 있고 이 미생물들의 다양성과 상호작용이 어떤 방식으로 인간의 건강에 영향을 미치는지 아직 그 이해가 부족한 실정이기 때문에 가급적이면 자연 상태의 마이크로바이옴을 원위치(in situ)에서 조절하는 기술이 필요하다 [7]. 최근에는 합성생물학을 활용한 원위치 마이크로바이옴 조절 기술들이 활발히 연구되고 있다. 이 기술들은 전통적인 수평적 유전자 전달 (horizontal gene transfer)을 재설계한 기술들로서 목표 미생물의 성장을 조절하거나 여러 기능을 가지는 유전자 화물 (genetic cargo)을 전달할 수 있다. 본 기고문에서는 수평적 유전자 전달의 일종인 형질도입 (transduction)과 접합 (conjugation)을 재설계한 합성생물학 기술들이 어떻게 원위치 마이크로바이옴 조절에 활용될 수 있는지 소개하고자 한다.
2. 형질도입 (transduction) 기반 마이크로바이옴 조절 기술
형질도입은 박테리아를 감염시키는 바이러스인 박테리오파지 (bacteriophage)를 통한 유전 물질 전달 방법이다. 박테리오파지는 목표 박테리아의 성장을 억제하기 때문에, 전통적으로 박테리아 감염을 제어하는 파지 테라피 (phage therapy) 용도로 활용되어왔다 [8]. 최근에는 박테리오파지 유전체에 합성 유전자 회로 (synthetic genetic circuit)를 도입함으로써 다양한 박테리아를 높은 특이성으로 제어할 수 있는 기술들이 보고되고 있다.
박테리오파지는 목표 박테리아 세포에 특이적으로 결합해 유전 물질을 주입하지만, 목표 박테리아가 보유한 유전자 서열까지는 감지할 수 없기 때문에 동일한 박테리아 종 중에서 병원성과 비병원성 균주까지 구별하기는 어렵다. 연구자들은 CRISPR-Cas (clustered regularly interspaced short palindromic repeats – CRISPR-associated protein) 시스템을 박테리오파지에 넣어준 뒤 형질도입을 통해 목표 박테리아에 주입시킴으로써, 특정 염기서열을 보유한 박테리아만 사멸하도록 만드는 서열 특이적 박테리오파지를 제작했다 [9–11]. CRISPR-Cas 시스템은 목표 염기 서열을 인식해 핵산을 자르는 서열 특이적 핵산 분해 시스템이다. Cas 단백질은 crRNA (CRISPR RNA)에 상보적인 서열을 갖는 핵산에 결합하여 그 핵산을 분해한다. 따라서, crRNA의 서열을 적절히 설계하면 특정 서열을 가지는 핵산을 선택적으로 분해할 수 있고, 이렇게 유전체가 분해된 박테리아는 사멸하게 된다.
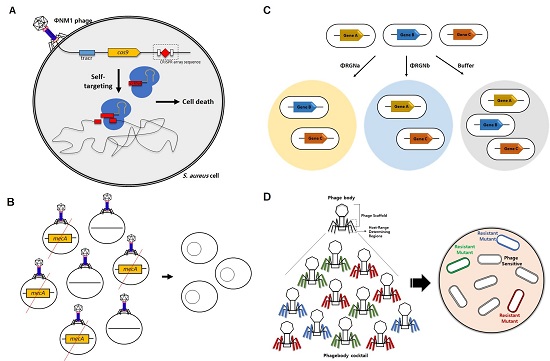
그림 1. 형질도입 기반 마이크로바이옴 조절 기술. (A) CRISPR-Cas를 포함한 박테리오파지를 활용한 유전자 서열 특이적 세포 사멸 기술 개념도. (B) CRISPR-Cas를 포함한 박테리오파지를 활용하면 특정 유전자 (mecA)를 포함한 균주만 선택적으로 사멸시킬 수 있다. (C) CRISPR-Cas를 포함한 박테리오파지를 이용해 인공적으로 조성한 미생물 군집에서 특정 미생물만을 선택적으로 사멸시킬 수 있다. (D) T3 박테리오파지의 숙주 범위 결정 구역 서열 다양화를 통한 숙주 특이성 엔지니어링.
Bikard 등은 Staphylococcus aureus를 감염시키는 ΦNM1 박테리오파지에 methicillin 내성 유전자인 mecA를 감지하는 CRISPR-Cas9 시스템을 설계하여 집어넣었다 [9] (그림 1A). 이들은 mecA 유전자를 보유하지 않은 S. aureus RNΦ와 mecA 유전자를 보유한 S. aureus USA300Φ를 섞은 뒤 mecA 특이적 박테리오파지를 첨가해주었다. 그 결과, S.aureus USA300Φ만 선택적으로 사멸된 것을 확인할 수 있었다 (그림 1B). Citorik 등은 대장균을 감염시키는 M13 박테리오파지에 CRISPR-Cas9 시스템을 집어넣고, 이를 활용해 카바페넴 내성 장내세균 (carbapenem-resistant Enterobacteriaceae)이나 장출혈성 대장균 (enterohemorrhagic Escherichia coli)을 선택적으로 사멸시켰다 [10]. 이들은 재설계된 박테리오파지를 활용하면 서로 다른 세 가지 E. coli strain들이 섞여 있는 인공 컨소시움에서도 목표로 하는 strain만을 선택적으로 제거할 수 있음을 보였다 (그림 1C). Kiga 등은 DNA를 자르는 Cas9 대신 RNA를 감지하여 자르는 Cas13a를 이용해 목표 RNA가 발현되면 해당 박테리아를 사멸시키는 박테리오파지를 개발했다 [11]. 이 기술을 활용하면 목표 유전자가 염색체상에 위치하지 않고 플라스미드에 위치하더라도 이로부터 발현된 RNA가 감지되기만 하면 목표 박테리아를 사멸시킬 수 있다.
한편, 박테리오파지의 목표 특이성은 일차적으로 박테리오파지와 목표 박테리아 세포의 결합에 의해 결정되므로, 박테리오파지 표면의 결합 단백질을 개량함으로써 목표 특이성을 조절할 수도 있다. Yehl 등은 E. coli B를 감염시키는 T3 phage의 tail fiber 단백질에서 숙주 범위를 결정하는 구역을 규명했다 [12] (그림 1D). 이들은 이 구역의 아미노산 서열을 다양화한 박테리오파지 라이브러리를 제작한 뒤 고처리능 스크리닝 (high-throughput screening)을 통해 숙주 특이성이 변화한 변종 박테리오파지들을 선별했다. 이렇게 개량된 박테리오파지들은 숙주 특이성이 변화했을 뿐만 아니라, 목표 박테리아가 세포 표면의 lipopolysaccharide 구조를 변화시켜서 기존의 박테리오파지를 회피하는 경우에도 여전히 목표 박테리아를 사멸시킬 수 있었다. 비록 해당 연구는 특정한 목표 박테리아의 변종들에 대해서만 제한적으로 특이성을 조절했지만, 향후에 숙주 결정 구역을 대규모로 조절하면 다양한 박테리아를 사멸시키는 새로운 박테리오파지를 개발하는 데에도 활용될 수 있을 것이라 기대한다.
3. 접합 (conjugation) 기반 마이크로바이옴 조절 기술
접합은 유전 물질을 제공하는 공여자 (donor)와 수용자 (recipient) 사이에 직접적인 물리적 통로가 형성되고, 이를 통해 유전 물질이 전달되는 방법이다. 접합은 수용자에 대한 특이성이 낮아서 매우 다양한 미생물들에 유전 물질을 전달하는 데 활용될 수 있다. 장 마이크로바이옴에는 굉장히 다양한 미생물 종이 포함되어 있다. 이 미생물들 중 대부분은 길들여지지 않은 (undomesticated) 미생물로서, 이들의 성장을 제어하거나 합성 유전자 회로를 도입해서 엔지니어링할 수 있는 마땅한 방법이 알려져 있지 않다. 따라서, 연구자들은 접합을 활용해 다양한 미생물들에 유전자 회로를 전달하는 방법을 연구하고 있다.
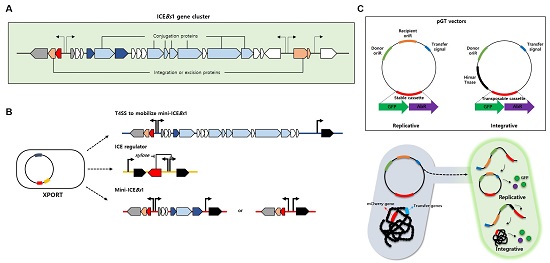
그림 2. 접합 기반 마이크로바이옴 조절 기술. (A) 야생형 ICEBs1 유전자군. (B) XPORT 균주 내부의 재설계된 ICEBs1 유전자군. (C) IncP α-family RP4 접합 시스템을 활용한 원위치 마이크로바이옴 엔지니어링 기술.
접합을 활용한 전통적인 유전자 전달법은 원위치 마이크로바이옴 조절이라는 목표를 달성하는 데 한계가 있었다. 먼저, 전달된 DNA가 플라스미드 형태로 존재하므로 적절한 선택압 (selection pressure)이 가해지지 않으면 쉽게 소실될 수 있다. 또한, 전달된 DNA 자체에 전달에 관여하는 유전자들이 포함되어 있으므로, 원치 않는 미생물들에 2차적으로 DNA를 전달할 가능성이 있다. 이런 문제를 해결하기 위해 Brophy 등은 Bacillus subtilis의 integrative and conjugative element (ICEBs1, 그림 2A)를 재설계하여 3개의 모듈로 나눈 뒤, B. subtilis 염색체상의 서로 다른 위치에 각 모듈을 도입하여 XPORT라는 공여자 균주를 개발했다 [13] (그림 2B). XPORT에 들어있는 3개의 모듈은 다음과 같다: 수용자 박테리아의 염색체에 외래 DNA를 삽입하는 데 필요한 유전자들이 들어있는 mini-ICEBs1; mini-ICEBs1을 전달하는 데 필요한 type IV secretion system (T4SS); inducer를 첨가하여 접합을 유도하기 위한 regulator. 연구자들은 mini-ICEBs1 내부에 전달시키고자 하는 외래 유전자들을 삽입하고 접합을 유도하여, 토양과 인간에서 분리된 마이크로바이옴에 외래 유전자들을 전달시키고 수용자 박테리아의 염색체에 성공적으로 삽입시켰다. XPORT 균주를 통한 접합을 활용하면, 외래 유전자들이 수용자의 염색체에 삽입되므로 별도의 선택압이 가해지지 않더라도 삽입된 유전자들이 안정적으로 유지될 수 있고, 외래 유전자가 T4SS와 분리되어 있으므로 추가적인 접합을 통해 원치 않는 미생물에 유전자들을 추가적으로 전달할 가능성이 없다는 장점이 있다.
접합은 다양한 수용자 박테리아에 유전자를 전달할 수 있다는 장점이 있지만, 마이크로바이옴을 구성하는 다양한 박테리아 중에서 어떤 박테리아가 유전자를 전달받아 엔지니어링될 수 있는지 확인하기 어려웠다. Ronda 등은 Grampositive와 Gram-negative 박테리아 모두에 유전자를 전달할 수 있는 IncPα-family RP4 접합 시스템을 이용해 마이크로바이옴을 구성하는 다양한 미생물들에 유전자 화물을 전달하고, 이 유전자 화물에 들어 있는 형광 단백질 유전자의 발현을 통해 유전자를 전달받은 수용자 박테리아를 분리해내는 MAGIC(metagenomic alteration of gut microbiome by in situ conjugation) 기술을 보고했다 [14] (그림 2C). 이 기술의 또 다른 특징은 유전자 화물을 수용자 박테리아 내에서 복제 가능한 플라스미드 형태 또는 수용자 박테리아 염색체에 끼어들어간 형태로 자유롭게 설계해 전달 가능하다는 점이다. 연구자들은 이 기술을 이용해 4개의 phyla와 19개의 genera에 속하는 297가지의 수용자 박테리아에 유전자 화물을 전달했다.
4. 결론 및 전망
마이크로바이옴은 지구 어디에나 존재하며, 그들이 속한 생태계에서 중요한 역할을 담당하고 있다. 장 마이크로바이옴은 인간의 건강에 다양한 영향을 미치는 것으로 알려져 있으며, 장 마이크로바이옴을 조절하여 질병을 치료하려는 다양한 연구들이 진행되고 있다. 마이크로바이옴과 인간의 건강 사이의 인과관계를 정확하게 밝혀내고, 새로운 치료법을 제시하려면 다양한 미생물들을 정교하게 조절하는 기술이 필요하다. 합성생물학은 유전자 부품들을 개발하고 이를 조립하여 특정 기능을 갖는 합성 유전자 회로를 제작함으로써 마이크로바이옴을 조절하는 새로운 기술 개발에 기여하고 있다. 특히 최근에는 수평적 유전자 전달 기법을 재설계한 형질도입 또는 접합 기반의 마이크로바이옴 조절 기술들이 소개되어 앞으로의 발전 가능성을 기대하게 만들고 있다.
앞으로 마이크로바이옴 조절 기술은 두 가지 측면에서 발전할 여지가 있다. 먼저, 현재의 기술은 매우 특이적이거나 또는 매우 범용적이다. 마이크로바이옴을 특이적으로 조절할 수 있는 기술은 다양한 미생물을 조절하기 어렵고, 다양한 미생물을 조절할 수 있는 기술은 특이성이 떨어진다. 따라서, 향후에는 마이크로바이옴에 포함된 다양한 미생물종 각각을 특이적으로 조절할 수 있는 기술이 개발되어야 한다. 두 번째로는, 현재의 마이크로바이옴 조절 기술은 대부분 박테리아에만 적용 가능하며, 마이크로바이옴을 구성하는 바이러스 (viruses)나 진균류 (fungi)를 조절하는 기술은 잘 알려져 있지 않다. 따라서, 앞으로 바이러스와 진균류를 조절하는 기술들이 개발되어야 마이크로바이옴과 인간의 건강 사이의 관계를 좀 더 완전하게 이해할 수 있으며, 새로운 치료법도 제시할 수 있을 것이다.
참고 문헌
1. Sender R, Fuchs S, Milo R. Revised estimates for the number of human and bacteria cells in the body. PLoS Biol. 2016,
14(8):e1002533.
2. Clemente JC, Ursell LK, Parfrey LW, Knight R. The impact of the gut microbiota on human health: an integrative view. Cell. 2012,
148(6):1258–1270.
3. Zhao L, Zhang F, Ding X, Wu G, Lam YY, Wang X, et al. Gut bacteria selectively promoted by dietary fibers alleviate type 2
diabetes. Science. 2018, 359(6380):1151–1156.
4. van Nood E, Vrieze A, Nieuwdorp M, Fuentes S, Zoetendal EG, de Vos WM, et al. Duodenal infusion of donor feces for recurrent
Clostridium difficile. N Engl J Med. 2013, 368(5):407–415.
5. Venturelli OS, Carr AC, Fisher G, Hsu RH, Lau R, Bowen BP, et al. Deciphering microbial interactions in synthetic human gut
microbiome communities. Mol Syst Biol. 2018, 14(6):e8157.
6. Zmora N, Soffer E, Elinav E. Transforming medicine with the microbiome. Sci Transl Med. 2019, 11(477).
7. Sheth RU, Cabral V, Chen SP, Wang HH. Manipulating Bacterial Communities by in situ Microbiome Engineering. Trends Genet.
2016, 32(4):189–200.
8. Merril CR, Scholl D, Adhya SL. The prospect for bacteriophage therapy in Western medicine. Nat Rev Drug Discov. 2003,
2(6):489–497.
9. Bikard D, Euler CW, Jiang W, Nussenzweig PM, Goldberg GW, Duportet X, et al. Exploiting CRISPR-Cas nucleases to produce
sequence-specific antimicrobials. Nat Biotechnol. 2014, 32(11):1146–1150.
10. Citorik RJ, Mimee M, Lu TK. Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases. Nat Biotechnol.
2014, 32(11):1141–1145.
11. Kiga K, Tan X-E, Ibarra-Chávez R, Watanabe S, Aiba Y, Sato’o Y, et al. Development of CRISPR-Cas13a-based antimicrobials
capable of sequence-specific killing of target bacteria. Nat Commun. 2020, 11(1):2934.
12. Yehl K, Lemire S, Yang AC, Ando H, Mimee M, Torres MDT, et al. Engineering Phage Host-Range and Suppressing Bacterial
Resistance through Phage Tail Fiber Mutagenesis. Cell. 2019, 179(2):459–469.e9.
13. Brophy JAN, Triassi AJ, Adams BL, Renberg RL, Stratis-Cullum DN, Grossman AD, et al. Engineered integrative and conjugative
elements for efficient and inducible DNA transfer to undomesticated bacteria. Nat Microbiol. 2018, 3(9):1043–1053.
14. Ronda C, Chen SP, Cabral V, Yaung SJ, Wang HH. Metagenomic engineering of the mammalian gut microbiome in situ. Nat
Methods. 2019, 16(2):167–170.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update 
