| 신약개발을 위한 인공지능 기술 동향 | |
|---|---|
| Date 2021-10-03 18:35:48 |
|

교수
덕성여자대학교 바이오공학전공
jyryu@duksung.ac.kr
1. 서론
하나의 약물이 미국 식품의약국(FDA)으로부터 품목허가를 받기까지 평균적으로 10년이 걸리고 1조원 이상의 비용이 소모된다. 이렇게 막대한 시간과 비용이 신약개발에 들어가지만, 신약개발의 성공 가능성은 매우 낮다. 신약후보물질이 임상시험에 들어가기도 어렵지만, 임상 1상 이후 FDA의 품목허가 승인을 받을 확률은 10% 정도로 알려져 있다 [1]. 즉, 임상 1상에 진입한 10개의 신약후보물질 중 최종적으로 품목허가 승인을 받는 약물은 하나에 불과한 것이다. 이처럼 신약개발은 대표적인 고위험 고수익 (high risk high return) 분야이다.
신약개발 과정은 크게 발견단계 (discovery)와 개발단계 (development)로 나뉜다. 발견단계는 표적 발굴, 유효물질 발굴, 선도물질 발굴 및 최적화 과정을 포함하며, 이를 통해 신약후보물질을 도출하는 단계이다. 개발단계는 동물과 사람에게서 신약후보물질의 안전성과 유효성을 확인하는 단계로 전임상시험과 임상시험을 실시한다. 이 과정에서 수많은 시행착오가 발생할 수 있으며 막대한 시간과 비용이 든다. 또한, 신약 후보물질이 전임상시험 또는 임상시험 단계에서 안전성, 유효성 등에 문제가 발생하여 실패하는 경우 신약개발에 투자된 막대한 연구 비용의 회수가 어렵다는 점도 신약 개발이 어려운 이유 중 하나이다.
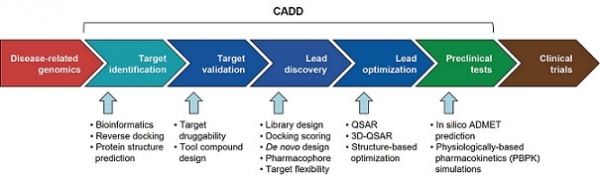
연구자들은 이렇게 신약개발에 드는 막대한 시간과 비용을 절감하고, 신약개발의 성공 가능성을 높이기 위한 다양한 시도를 해왔다. 대표적인 예가 1990년대 중반부터 시작된 컴퓨터를 이용한 신약설계 (Computer Aided Drug Design; CADD)이다 [2]. 그림 1에서 볼 수 있듯이, CADD는 표적 발굴 및 검증, 선도물질 발굴, 전임상시험에 이르기까지 다양한 신약개발 과정에 활용될 수 있다. 이때, 다양한 기계 학습 방법론들이 사용되어 약물의 약효뿐 아니라 물리화학적 특성과 ADME/Tox (흡수, 분포, 대사, 배설, 독성)를 예측하는 모델이 구축되어 활용된다.
최근 다른 산업 분야와 마찬가지로 빅데이터, 인공지능과 같은 새로운 기술들이 신약개발 분야에 적극적으로 도입되고 활용되고 있다. 특히, 인공신경망 기반의 딥러닝 예측 모델은 전통적인 기계 학습 방법론이 효과적으로 적용되기 어려운 비정형 데이터의 처리 및 학습에 특화된 성능을 보여주고 있다. 또한, 변형 자동 인코더 (Variational Autoencoder; VAE)나 생성적 적대 신경망 (Generative Adversarial Network; GAN)과 같은 딥러닝 기반의 새로운 형태의 생성모델은 학습 데이터의 분포를 학습하여 원하는 특성을 갖는 새로운 데이터를 생성할 수 있다. 이는 초기 신약개발 단계에서 시간이 많이 소요되는 선도물질 최적화 단계에 화합물의 유도체를 생성하는 목적으로 활용되고 있다. 본 기고문에서는 초기 신약개발 과정에서 활용되고 있는 인공지능 기술과 관련된 연구 결과를 소개하고자 한다.
2. 본론
2.1. 표적 발굴
신약개발의 첫 단계는 특정 질병을 치료할 수 있는 약물의 표적을 발굴하는 단계이다. 예를 들어, 암 치료 목적의 표적 발굴에서는 암세포의 성장을 억제하고, 암세포를 사멸시킬 수 있는 표적 유전자를 인공지능 기술을 적용하여 예측할 수 있다 [3]. 이때, 약물 부작용을 최소화하기 위하여 암세포의 성장은 억제하고 정상 세포에는 영향이 적은 표적을 발굴하는 것이 중요하다. 항암 표적 유전자를 실험적으로 찾아내기 위한 방법으로는 특정 암세포에서 특정 유전자의 발현을 실험적으로 감소시킨 후 암세포의 생존능력을 측정하는 방법이 있다. 이때 특정 유전자의 발현을 감소시키기 위해 siRNA나 CRISPR interference (CRISPRi)와 같은 실험 기법을 사용할 수 있다. 하지만 시간과 비용의 문제로 실험적으로 모든 유전자를 대상으로 평가하기 어려울 뿐 아니라, 정상 세포를 대상으로 실험을 할 수 없는 한계가 있다.
최근 이러한 어려움을 극복할 수 있는 in silico siRNA 기술이 개발되었다 [4]. In silico siRNA 기술은 특정 암세포의 유전자 조절 네트워크와 유전자 발현 정보를 사용하여 특정 유전자 녹다운 (knock-down) 후 세포 내 유전자 발현 변화를 계산하고, 이를 통해 암세포의 성장 여부를 판단하는 모델이다. 이 모델을 이용하면 특정 암세포에서의 모든 유전자의 개별 녹다운 후 암세포의 성장 여부를 예측해 실험의 양을 획기적으로 줄여줄 수 있다. 또한, 실험적으로는 확인할 수 없는 정상 세포에서도 적용해볼 수 있고, 이를 통해 암세포의 성장만을 특이적으로 억제하여 부작용이 적고, 치료 효과가 높은 표적 유전자를 발굴하는 데 활용될 수 있을 것이다.
신약개발을 위한 약물의 표적을 발굴하는 전통적인 방법은 문헌 조사였으며 현재도 이를 통해 관심 있는 질병의 표적을 찾아내고 있다. 예를 들어, Nature, Cell, Science와 같은 저명한 학술지에 관심 있는 유전자의 기능과 질병에서의 역할이 규명된 논문이 출판되면 해당 유전자를 표적으로 선정하고 신약개발을 진행하는 것이다. 문헌 조사를 통한 약물의 표적 발굴에도 인공지능 기술이 활용될 수 있다. 인공지능 분야 중 컴퓨터 비전과 함께 가장 발전이 빠른 분야가 자연어 처리 분야이다. 최근 개발된 인공지능 모델은 세계 최대의 생물 의학 문헌 데이터베이스인 PubMed에 존재하는 약 1,800만 건의 문헌을 인공지능을 통해 학습하고, 이를 통해 관심 있는 질병을 치료하기 위한 표적 유전자를 예측할 수 있다 [5]. 이처럼, 인공지능 기술을 이용하면 사람이 파악하기 힘든 양의 문헌 분석을 통해 보다 성공률 높은 약물의 표적을 제안할 수 있을 것으로 기대한다.
2.2. 유효물질 발굴
표적 단백질이 정해진 후 다음 단계는 표적 단백질을 저해하는 유효물질을 탐색하는 과정이다. 일반적으로 이 과정에서 적게는 수천 개에서 많게는 수백만 개의 화합물로 구성된 화합물 라이브러리를 이용하여 약물가상탐색 (virtual screening)을 수행한다 [6]. 이때 표적 단백질의 구조가 밝혀진 경우는 분자 도킹 (molecular docking) 방법을 이용하여 표적 단백질에 가장 결합력이 좋을 것으로 계산된 화합물을 선별한다. 표적 단백질의 구조가 밝혀지지 않은 경우에는 표적 단백질을 저해한다고 알려진 화합물들과의 물리화학적 특성을 비교하는 방법을 통해 화합물을 선별하게 된다. 이렇게 예측된 화합물들은 in vitro 생화학적 분석을 통해 표적 단백질에 실제 결합하는지 실험적으로 확인하는 과정을 통해 유효물질을 발굴할 수 있다.
약물가상탐색 과정에서 중요한 것은 약효가 있을 것으로 예측된 화합물 중 실제 약효가 있는 화합물의 비율인 유효물질 도출 비율 (hit rate)을 높이는 것이다. 유효물질 도출 비율을 높이기 위한 다양한 인공지능 기반 방법론들이 활용되고 있고, 그 중 대표적인 방법이 분자 도킹의 정확도를 향상하는 전략이다. 일반적으로 표적 단백질의 구조가 밝혀진 경우 사용되는 분자 도킹은 약물가상탐색에서 가장 널리 사용되는 방법론이지만, 예측 결과의 위양성 (false positive) 비율이 높은 것이 단점이다. 즉, 화합물과 표적 단백질의 결합력이 높게 계산되어 유효물질로 예측되지만, 실제는 약효가 없는 경우이다. 이러한 분자 도킹의 위양성 비율을 낮추기 위하여 분자 도킹 결과로 얻어진 화합물과 표적 단백질의 결합형태를 분석하여 약효를 예측한다. 이때 인공지능 모델은 화합물과 표적 단백질의 결합 에너지를 계산하지 않고, PDBbind 데이터베이스에 존재하는 화합물과 단백질의 결합 형태와 약효 간의 관계를 학습하여 약효를 예측한다 [7]. 분자 도킹 방법론과 함께 이러한 예측 모델을 동시에 사용하여 위양성 비율을 낮출 수 있다.
분자 도킹은 표적 단백질의 구조가 밝혀진 경우 약물가상탐색에 사용되는 매우 유용한 기술이지만, 표적 단백질의 구조가 밝혀지지 않은 경우 활용할 수 없다는 단점이 있다. 단백질의 서열 정보로 단백질 구조를 예측하는 방법론들이 오래전부터 사용되기는 하였으나 예측 정확도가 만족스럽지 못하였다. 최근 Google의 자회사인 DeepMind에서 개발한 단백질 구조 예측 인공지능 모델인 AlphaFold2는 단백질 구조를 매우 정확하게 예측한다는 평가를 받고 있고, 실제로 단백질 구조예측 학술대회 (CASP)에서 압도적인 성적으로 1위를 차지하였다 [8]. AlphaFold2로 인간을 비롯한 20개 모델 생물체에서 35만 개 이상의 구조를 예측해 결과를 제공하고 있다. AlphaFold2를 통해 구축된 단백질의 구조 정보는 신약개발 과정에서 매우 크게 기여할 것으로 기대한다.
2.3. 선도물질 발굴
약물가상탐색 등을 통해 발굴한 유효물질은 약효, 독성, 약물성 등 여러 가지 측면에서 부족함이 있을 수 있다. 무엇보다도 유효물질은 이미 누군가가 합성하여 라이브러리를 통해 제공되는 화합물이기에 물질특허를 확보할 수 없다. 따라서, 유효물질의 구조를 변형하여 약효는 보다 증대시키고, 물질특허 확보가 가능한 새로운 구조의 화합물을 도출하는 단계가 필요하다. 이 단계를 선도물질 발굴 단계라고 하며, 초기 신약개발에서 많은 시간이 소요되는 단계이다.
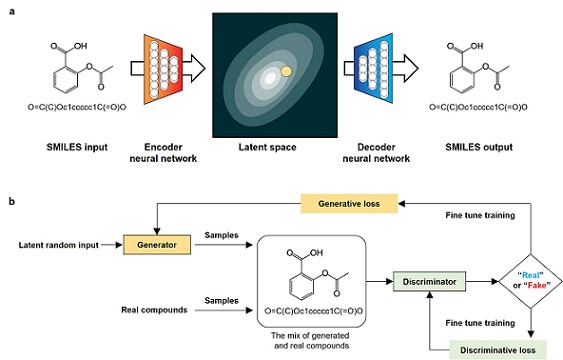
선도물질 발굴 단계는 최신 인공지능 기술이 가장 활발하게 적용되고 있는 분야이다. 특히, 변형 자동 인코더 (VAE) [9]나 생성적 적대 신경망 (GAN) [10]과 같은 딥러닝 기반의 생성 모델이 적용된다. 이는 인공지능 기술이 가장 활발하게 연구되는 컴퓨터 비전 분야와 매우 유사한 목적을 갖기 때문이다. 최근 컴퓨터 비전 분야에서 초상권이 없는 사람들의 얼굴을 현실과 가깝게 만들어내는 연구를 수행하고 있고, 나아가 원하는 특징을 갖는 얼굴을 생성해낼 수 있다. 이는 선도물질 발굴의 목적과 유사하다. 즉, 물질특허를 확보할 수 있는 새로운 화합물을 설계해야 하며 약효와 같은 원하는 특성을 갖는 화합물을 생성해야 한다.
대표적인 연구 결과는 2019년 Nature Biotechnology에 게재된 Insilico Medicine 사의 연구 결과이다. Insilico Medicine 사는 신약 후보물질을 설계하는 인공지능 시스템인 GENTRL을 개발하여 공개하였다 [11]. 인공지능 시스템을 이용하여 DDR1 kinase 저해제를 설계, 합성, 검증까지 46일 이내에 완료하여 전 세계적으로 큰 주목을 받았다.
선도물질 발굴 단계에서 다양한 생성 모델이 개발되고 있고, 선도물질 최적화를 위해 사용되고 있다. 이러한 생성 모델은 추후 선도물질 최적화 단계에서 매우 유용하게 사용될 것으로 전망하지만, 해결해야 할 문제가 존재한다. 대표적인 문제는 생성 모델이 제안하는 화합물의 구조가 실제 유기합성을 하기 어렵다는 것이다. 이러한 문제를 해결하기 위해 생성 모델은 약효가 좋고 물질특허를 확보할 수 있을 뿐 아니라 실제로 합성도 가능한 화합물의 구조를 제안해야 할 것이다.
3. 결론 및 전망
치료제가 필요한 질병의 수는 희귀 질환을 포함하면 8,000종 이상이지만 [12], 이 중 95%는 마땅한 치료법이 없는 실정이다. 또한 코로나19 (COVID-19)와 같은 감염병이 인류를 위협하고 있다. 이러한 상황에서, 더욱 빠르고 효율적인 신약개발 전략이 필요하고 인공지능이 핵심적인 역할을 할 것으로 기대한다.
앞서 소개한 것처럼 인공지능 기반 신약개발은 미래 제약 산업에서 매우 중요한 역할을 할 것으로 전망된다. 최근 국제적으로 인공지능 기반 신약개발 회사들이 설립되고 있으며 전통적인 제약사들은 인공지능 신약개발 회사와의 협력을 통해 신약개발을 시도하고 있다. 국내에서도 인공지능 기반 신약개발 연구자가 늘어나고 있으며, 인공지능 기반 신약개발 기술을 핵심 기술로 하는 벤처 회사들이 설립되어 성과를 내고 있다. 국내 연구자 및 기업이 개발한 인공지능 기반 신약의 개발을 기대하며, 이를 통해 국내 제약 산업의 경쟁력 제고를 기대한다.
참고문헌
1. Mullard, A., Parsing clinical success rates. Nat. Rev. Drug Discov., 15(7): p. 447, 2016.
2. Tang, Y., et al., New technologies in computer-aided drug design: Toward target identification and new chemical entity discovery. Drug Discov. Today Technol., 3(3): p. 307-13, 2006.
3. Ho, D., Artificial intelligence in cancer therapy. Science, 367(6481): p. 982-983, 2020.
4. Jang, K., et al., Computational inference of cancer-specific vulnerabilities in clinical samples. Genome Biol., 21(1): p. 155, 2020.
5. Lee, J., et al., BioBERT: a pre-trained biomedical language representation model for biomedical text mining. Bioinformatics, 36(4): p. 1234-1240, 2020.
6. Shoichet, B.K., Virtual screening of chemical libraries. Nature, 432(7019): p. 862-5, 2004.
7. Lim, J., et al., Predicting Drug-Target Interaction Using a Novel Graph Neural Network with 3D Structure-Embedded Graph Representation. J. Chem. Inf. Model., 59(9): p. 3981-3988, 2019.
8. Jumper, J., et al., Highly accurate protein structure prediction with AlphaFold. Nature, 2021.
9. Gomez-Bombarelli, R., et al., Automatic Chemical Design Using a Data-Driven Continuous Representation of Molecules. ACS Cent. Sci., 4(2): p. 268-276, 2018.
10. Bian, Y. and X.Q. Xie, Generative chemistry: drug discovery with deep learning generative models. J. Mol. Model., 27(3): p. 71, 2021.
11. Zhavoronkov, A., et al., Deep learning enables rapid identification of potent DDR1 kinase inhibitors. Nat. Biotechnol., 37(9): p. 1038-1040, 2019.
12. Haendel, M., et al., How many rare diseases are there? Nat. Rev. Drug Discov., 19(2): p. 77-78, 2020.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update