| 효과적인 바이오의약품 생산을 위한 CHO 세포주 불안정성의 원인 및 극복 방안 | |
|---|---|
| Date 2022-09-26 16:11:57 |
|

1. 서론
CHO (Chinese hamster ovary) 세포는 바이오의약품, 특히 항체의약품의 생산에 가장 널리 사용되는 동물세포이다 [1]. CHO 세포는 (1) 외부의 유전자를 도입, 증폭하기 용이하고 (2) 다양한 환경에 비교적 쉽게 적응 배양이 가능하며 (3) 사람과 유사한 번역 후 변형
(posttranslational modification)이 가능하면서도 (4) 인체 감염 바이러스에 대해 안전하다는 장점이 있다. CHO 세포는 이렇듯 바이오의약품의 생산에 있어 많은 장점을 보유한 반면 한계점 또한 가지고 있는데, 그중 대표적인 것이 세포주 불안정성이다. 세포주 불안정성은 세포의 장기 배양중 발생하는 예기치 않은 배양성능의 변화– 세포 성장속도, 최대 세포농도, 바이오의약품 농도 및 품질, 생산성, 배양기간 등 –를 의미하며 하며, 특히 생산성의 감소는 상품 비용 (cost of goods)을 증가시키고 바이오의약품 품질 변화는 약효에 직접적인 영향을 미치는 등 중대한 문제를 야기한다 [2].
이러한 문제를 방지하기위해 현재로서는 개발된 세포주들의 장기배양을 통해 불안정성을 시험하는 과정을 거치는데, 약 6개월 정도의 긴 기간이 소요된다. 따라서 세포주 불안정성의 원인을 이해하고 이를 방지하는 것이 바이오의약품의 개발기간을 단축시키는데 매우 중요하다. 이에 본 기고문에서는 CHO 세포주 불안정성의 원인 규명 및 극복 방안에 대한 최신 연구 동향을 소개하고자 한다.
2. CHO 세포주 불안정성의 주요 원인
2.1. 유전체 불안정성으로 인한 유전자 소실
CHO 세포의 유전체는 Chinese hamster의 이배체 유전체(2n = 22)와 비교하여 염색체의 수, 크기 및 구조 등에서 뚜렷한 차이가 나타나는 특징을 갖고 있다 [3]. 대부분의 CHO 세포에서 염색체 이수성 (aneuploidy)이 나타나며 크고 작은 구조적 변이 (structural variation)들이 발생하여 염색체의 구조가 원래의 상태와 상당히 다르게 존재한다 [4]. 이러한 특성은 CHO 세포의 고유한 유전적 가소성 (plasticity)과 관련이 있으며, 계속되는 배양으로 세대를 거듭하면서 단일 염기 변이 (Single Nucleotide Variation, SNV)와 함께 염색체 재배열
(chromosomal rearrangement) 등의 구조적 변이가 발생하게 된다 [5]. 특히, 특정 염색체 부위가 재배열됨으로 인해 장기배양에 따른 재조합 단백질 생산량이 크게 감소하는 등 염색체의 구조적 변이 자체만으로도 세포주 불안정성의 직접적인 요인이 될 수 있다는 연구가 보고되었다 [6].
이와 같이 다양한 DNA의 변이 양상은 바이오의약품 생산 세포주가 되기 이전 단계의 숙주세포 (host cell) 상태에서도 유사하게 나타나며, 목적 단백질의 발현을 위한 외래 유전자의 삽입 및 생산성 증대를 위한 유전자 증폭 과정을 거치면서 더욱 심화된다 [7]. 목적 단백질을 암호화하고 있는 외래 유전자가 변형되거나 소실되는 현상은 위에서 언급한 세포주 고유 특성, DNA 복제 스트레스, 배양환경 변화 등의 요인으로 DNA의 이중 가닥 절단 (Double Strand Break,DSB)이 발생하고 이것이 불완전하게 수리되는 과정에서 일어나게 된다 [8]. 이러한 유전자 소실이 CHO 세포주의 생산성 감소와 의미 있는 상관관계를 보이며, 따라서 세포주 불안정성의 직접적인 원인으로 꼽힌다 [9].
2.2. 후성유전적 변형 (epigenetic modification)에 따른 유전자 침묵 (gene silencing)
최근 연구들이 주목하고 있는 후성유전적 변형은 DNA 메틸화 (DNA methylation)와 히스톤 변형 (histonemodification), 그리고 비암호화 RNA (non-coding RNA, ncRNA)와 주로 관련되어 있다. DNA 메틸화는 주로 목적 유전자를 포함한 프로모터 (promoter)나 인핸서
(enhancer) 부위에 발생하며, 메틸화가 진행되면 전사체 (transcriptionalfactor)와의 결합을 막아 유전자의 전사 및 발현이 감소하게 된다 [10]. 한편 히스톤 변형은 염색질을 형성하는 기본단위인 뉴클레오솜 (nucleosome)을 형성하는 히스톤 단백질에 변화가 생겨 염색질의 구조가 변하는 현상을 말한다. 느슨한 양상을 보이는 진정염색질 (euchromatin)에서는 전사가 활발하게 일어나지만 이와 반대로 촘촘하게 응집된 형태의 이질염색질 (heterochromatin)에서는 전사가 잘 진행되지 않는다 [11]. ncRNA는 DNA로부터 전사되어 RNA 자체로 존재하지만 단백질로 번역되지는 않는 기능적인 분자를 말하며, 일반적으로 전사 및 이후 과정을 조절하는 기능을 한다 [12]. 이들은 공통적으로 외부로부터 도입된 DNA 서열이나 유전체 본래의 염기서열 자체는 변화시키지 않지만, 유전자의 전사 수준을 조절하여 장기적으로 생산성을 감소시킨다 [13]. 이러한 후성유전적인 특징 또한 CHO 세포 자체의 유전체 불안정성과 간접적인 연관이 있다고 볼 수 있으나, 유전자 사본 수가 더 많음에도 불구하고 목적 단백질의 생산량이 유전자 사본 수가 더 적은 세포보다 크게 감소한 사례가 있어 세포 내의 후성유전적 조절 패턴이 세포주 불안정성의 또 다른 주요 원인으로 여겨진다 [14,15].
어떠한 이유로든 – 즉, 유전자 소실 혹은 침묵 – 세포가 목적 단백질을 덜 생산하게 되면 (혹은 생산하지 않게 되면)소모하지 않은 에너지
(ATP)와 자원(아미노산)을 바이오매스를 늘리는 데 사용할 수 있게 된다. 이는 결과적으로 생산성이 낮은 세포의 성장을 빨라지게 만들어 곧 전체 세포 중 다수를 차지하게 하고 배양 전체의 생산성을 감소시키게 된다.
3. 안정한 CHO 세포주 개발을 위한 전략
3.1. 세포의 DNA 복구 기능 향상을 통한 안정성 개선
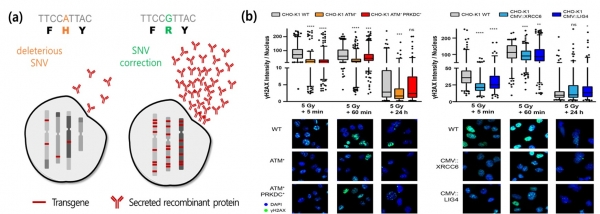
CHO 세포의 유전체 불안정성에 대한 이해 및 이를 극복하기 위한 DNA 복구경로를 확인하는 연구는 이전부터 이루어져 왔다 [16]. 하지만 DNA 복구 관련 유전자에 영향을 주는 다양한 돌연변이 사례를 제시하고 다양한 연구에 이용되고 있는 CRISPR/Cas9 기반 유전자 편집 기술을 통해 CHO 세포의 DNA 복구 능력을 회복시키고자 하는 연구는 최근 에서야 보고되었다. 이 연구에 따르면 DNA – 특히 DSB – 복구에 영향을 주는 단일 유전자를 복원하는 것만으로도 장기적인 유전체 안정성을 증가시키고 목적 단백질의 높은 생산성을 유지할 수 있었다.
(그림 1(a)) [17].
그림 1. (a) 변이된 DNA 복구 유전자의 교정을 통한 외래 유전자 사본 수 및 재조합 단백질 생산량 증가에 대한 모식도. (b) CHO-K1세포의 DNA 복구 유전자의 교정(좌) 또는 과발현(우)에 따른 DSB 양상 [17]. DSB는 X-선을 통해 유도되었으며, γH2AX는 DSB에 대한 바이오마커이다.
저자들은 Chinese hamster fibroblast와의 DNA 복구 능력 비교를 통해 CHO 세포에서 DNA 복구 능력이 손상되어 있다는 것을 확인했으며 11개의 CHO 세포주의 WGS (whole genome sequencing) 데이터를 분석하여 DNA 복구 관련 유전자에서 단일 염기 변이체를 발견했다. 파악된 변이의 양상을 분석하여 DSB 수리에 관여하는 경로인 비상동 말단 연결 (nonhomologous end-joining, NHEJ)과 상동 재조합 (homology-directed repair, HDR) 중 NHEJ의 복원이 CHO 세포에서 염색체 불안정성과 목적 유전자 손실을 완화하는 데 더 중요하게 기여할 것으로 예측하였다. 이후 4가지 후보 유전자 (ATM, PRKDC, XRCC6, LIG4)에 대하여 과발현하거나 또는 돌연변이를 교정하였을 때 DNA 복구 속도 및 정확도에 영향을 주는지 확인하였다. 그 결과 전반적인 복구 능력이 향상되었으며, 핵형 및 유전체의 안정성도 개선되었다(그림 1(b)).
3.2. 표적 후성유전 편집 (Targeted epigenetic editing)을 통한 세포주 안정성 개선
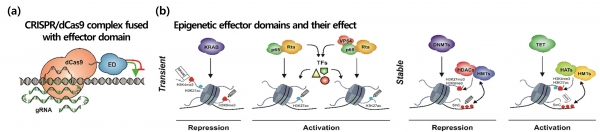
CHO 세포를 대상으로 표적 후성유전 편집을 시도한 연구는 최근에 보고되고 있는데, 이는 CHO 세포 및 Chinese hamster의 유전체 데이터의 완전성 및 가용성이 개선되어 CRISPR 기반 기술을 적용하기에 용이해졌기 때문이다 [18,19]. 플라스미드의 유전체 내 도입 없이 목적 유전자의 발현을 선택적으로 조절할 수 있다는 장점을 보유하고 있어 표적 후성유전 편집은 세포주 개발에서 유용한 도구로 이용될 수 있다. CRISPR 기반 후성유전 조절 방식은 효과기 도메인 (effector domain, ED)과 DNA 타게팅 도메인을 융합하는 것으로, 주로 gRNA와
nuclease 활성이 없는 dCas9을 활용하여 의도하는 유전체 상의 위치에 효과기를 표적화 시킴으로써 유전자 발현을 조절한다 (그림 2(a)) [20,21].
그림 2. (a) 동물세포의 표적 후성유전 편집에 이용되는 CRISPR/dCas9-효과기 도메인 복합체. (b) 후성유전적 효과기 도메인의 예시 및 효과 [22].
CHO 세포에 적용된 후성유전 효과기 도메인은 KRAB (Krüppel Associated Box), PR (p65, Rta), VPR (VP64, p65,Rta) 등이 있다 (그림 2(b)). 여기서 KRAB은 전사를 억제하는 도메인이며 PR과 VPR은 반대로 활성화시키는 도메인이다. 이 도메인들을 적용한 연구의 대부분은 표적 유전자의 일시적인 활성화 또는 침묵만을 유도하였는데, 이는 이러한 유전자 발현 조절 도구들을 지속적으로 발현하는 경우 세포에 과발현 스트레스와 이로 인한 단백질 생산 능력의 저하를 야기할 수 있기 때문이다. 따라서 이러한 전략은 유전자 과발현이나 knock-out와 같은 영구적인 유전자 발현 조절기술을 적용하기 이전 단계의 유전자 스크리닝 목적으로 활용되기에 더 적합하다 [24,25].
한편 표적 후성유전 편집을 통해 CHO 세포 내 유전자의 발현을 안정적으로 조절한 연구도 보고되었다. Marx와 그동료들은 dCas9을 기반으로 DNA 탈메틸화 또는 메틸화를 적용하여 CHO 세포주에서 β-Galactoside α-2,6-Sialyltransferase 1 (st6gal1) 유전자를 안정적으로 활성화시키고 다시 침묵시키는 과정을 진행하였다. Ten-Eleven Translocation methylcytosine dioxygenase 1 (TET1)의 촉매 도메인을 dCas9와 결합시키고 이 복합체를 휴면 중인 st6gal1 유전자의 프로모터에 위치시켜 전사체 및 N -글리칸 수준을 분석하였다. 또한 DNA methyltransferase 3 alpha (DNMT3A)의 촉매 도메인과 dCas9의 복합체를 이용한 프로모터의 연속적인 재메틸화를 통해 st6gal1의 효과적인 하향조절을 확인했다. 성장 능력 및 재조합 단백질 생산성에는 별다른 영향이 없었으며, dCas9-TET1 시스템을 활용한 일시적인 형질감염 (transfection)은 80일 이상 동안 st6gal1 유전자가 안정적으로 발현되도록 만들었다 [26].
이를 통해 DNA 메틸화가 장기간 유지되는 과정이라는 것을 확인하였고 표적 후성유전 편집 기술로도 목적 유전자와 조절 전략에 따라 CHO 세포주의 생산 안정성 개선을 꾀할 수 있다는 것을 보였다.
4. 결론 및 전망
바이오의약품 생산 세포주의 개발기간 단축은 바이오기업의 이익뿐만 아니라 바이오의약품을 필요로 하는 환자를 위해서도 꼭 필요하다. 특히, 최근의 COVID-19 팬데믹을 겪으면서 감염병 출현의 상황에서 단기간에 치료제를 개발,생산하는 것이 매우 중요하다는 것을 깨닫게 되었다. 세포주 개발기간의 절반가량을 차지하는 세포주 불안정성 시험과정을 간소화하거나 기간을 단축할 수 있다면 바이오의약품 개발 기간 및 비용 감소, 나아가 국민 건강 증진으로 이어질 것으로 기대한다.
참고문헌
1. Walsh, G. (2018). Biopharmaceutical benchmarks 2018. Nature biotechnology, 36(12), 1136-1145.
2. Dahodwala, H., & Lee, K. H. (2019). The fickle CHO: a review of the causes, implications, and potential alleviation of the CHO cell line instability problem. Current opinion in biotechnology, 60, 128-137.
3. Wurm, F. M., & Hacker, D. (2011). First CHO genome. Nature biotechnology, 29(8), 718-720.
4. Vcelar, S., Jadhav, V., Melcher, M., Auer, N., Hrdina, A., Sagmeister, R., ... & Borth, N. (2018). Karyotype variation of CHO host cell lines over time in culture characterized by chromosome counting and chromosome painting. Biotechnology and bioengineering, 115(1), 165-173.
5. Wurm, M. J., & Wurm, F. M. (2021). Naming CHO cells for bio‐manufacturing: Genome plasticity and variant phenotypes of cell populations in bioreactors question the relevance of old names. Biotechnology Journal, 16(7), 2100165.
6 Baik, J. Y., & Lee, K. H. (2017). A framework to quantify karyotype variation associated with CHO cell line instability at a single‐cell level. Biotechnology and Bioengineering, 114(5), 1045-1053.
7. Vcelar, S., Melcher, M., Auer, N., Hrdina, A., Puklowski, A., Leisch, F., ... & Borth, N. (2018). Changes in chromosome counts and patterns in CHO cell lines upon generation of recombinant cell lines and subcloning. Biotechnology Journal, 13(3), 1700495.
8. Qian, Y., Sowa, S. W., Aron, K. L., Xu, P., Langsdorf, E., Warrack, B., ... & Li, Z. J. (2020). New insights into genetic instability of an industrial CHO cell line by orthogonal omics. Biochemical Engineering Journal, 164, 107799.
9. Bandyopadhyay, A. A., O’Brien, S. A., Zhao, L., Fu, H. Y., Vishwanathan, N., & Hu, W. S. (2019). Recurring genomic structural variation leads to clonal instability and loss of productivity. Biotechnology and bioengineering, 116(1), 41-53.
10. Yang, Y., Chusainow, J., & Yap, M. G. (2010). DNA methylation contributes to loss in productivity of monoclonal antibodyproducing CHO cell lines. Journal of biotechnology, 147(3-4), 180-185.
11. Clapier, C. R., Iwasa, J., Cairns, B. R., & Peterson, C. L. (2017). Mechanisms of action and regulation of ATP-dependent chromatin-remodelling complexes. Nature reviews Molecular cell biology, 18(7), 407-422.
12. Kaikkonen, M. U., Lam, M. T., & Glass, C. K. (2011). Non-coding RNAs as regulators of gene expression and epigenetics. Cardiovascular research, 90(3), 430-440.
13. Ho, S. C., Koh, E. Y., Soo, B. P., Chao, S. H., & Yang, Y. (2016). Evaluating the use of a CpG free promoter for long-term recombinant protein expression stability in Chinese hamster ovary cells. BMC biotechnology, 16(1), 1-13.
14. Boyko, A., & Kovalchuk, I. (2011). Genome instability and epigenetic modification—heritable responses to environmental stress?. Current opinion in plant biology, 14(3), 260-266.
15. Veith, N., Ziehr, H., MacLeod, R. A., & Reamon-Buettner, S. M. (2016). Mechanisms underlying epigenetic and transcriptional heterogeneity in Chinese hamster ovary (CHO) cell lines. BMC biotechnology, 16(1), 1-16.
16. Shen, M. R., Mohrenweiser, H., Thompson, L. H., Thelen, M. P., & Zdzienicka, M. Z. (1998). Mutations in hamster single-strand break repair gene XRCC1 causing defective DNA repair. Nucleic acids research, 26(4), 1032-1037.
17. Spahn, P. N., Zhang, X., Hu, Q., Lu, H., Hamaker, N. K., Hefzi, H., ... & Lewis, N. E. (2022). Restoration of DNA repair mitigates genome instability and increases productivity of Chinese hamster ovary cells. Biotechnology and Bioengineering, 119(3), 963-982.
18. Lewis, N. E., Liu, X., Li, Y., Nagarajan, H., Yerganian, G., O'brien, E., ... & Palsson, B. O. (2013). Genomic landscapes of Chinese hamster ovary cell lines as revealed by the Cricetulus griseus draft genome. Nature biotechnology, 31(8), 759-765.
19. Rupp, O., MacDonald, M. L., Li, S., Dhiman, H., Polson, S., Griep, S., ... & Lee, K. H. (2018). A reference genome of the Chinese hamster based on a hybrid assembly strategy. Biotechnology and bioengineering, 115(8), 2087-2100.
20. Kungulovski, G., & Jeltsch, A. (2016). Epigenome editing: state of the art, concepts, and perspectives. Trends in Genetics, 32(2), 101-113.
21. Shen, C. C., Sung, L. Y., Lin, S. Y., Lin, M. W., & Hu, Y. C. (2017). Enhancing protein production yield from chinese hamster ovary cells by CRISPR interference. ACS synthetic biology, 6(8), 1509-1519.
22. Marx, N., Eisenhut, P., Weinguny, M., Klanert, G., & Borth, N. (2022). How to train your cell-Towards controlling phenotypes by harnessing the epigenome of Chinese hamster ovary production cell lines. Biotechnology Advances, 107924.
23. Pulecio, J., Verma, N., Mejía-Ramírez, E., Huangfu, D., & Raya, A. (2017). CRISPR/Cas9-based engineering of the epigenome. Cell Stem Cell, 21(4), 431-447.
24. Shen, C. C., Sung, L. Y., Lin, S. Y., Lin, M. W., & Hu, Y. C. (2017). Enhancing protein production yield from chinese hamster ovary cells by CRISPR interference. ACS synthetic biology, 6(8), 1509-1519.
25. Karottki, K. J. L. C., Hefzi, H., Xiong, K., Shamie, I., Hansen, A. H., Li, S., ... & Lewis, N. E. (2020). Awakening dormant glycosyltransferases in CHO cells with CRISPRa. Biotechnology and bioengineering, 117(2), 593-598.
26. Marx, N., Dhiman, H., Schmieder, V., Freire, C. M., Nguyen, L. N., Klanert, G., & Borth, N. (2021). Enhanced targeted DNA methylation of the CMV and endogenous promoters with dCas9-DNMT3A3L entails distinct subsequent histone modification changes in CHO cells. Metabolic engineering, 66, 268-282.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update