| 무세포(cell-free) 시스템을 이용한 펩타이드모방분자 (peptidomimetics) 합성 동향 | |
|---|---|
| Date 2022-09-26 16:40:59 |
|

1. 서론
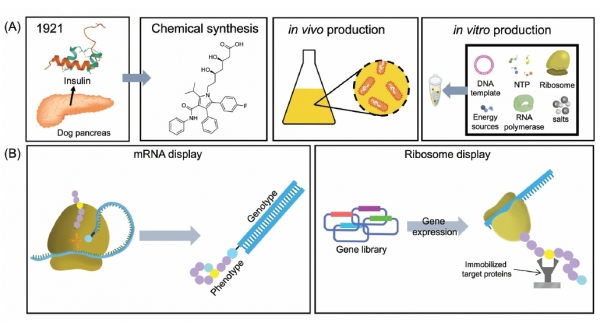
펩타이드는 서로 다른 아미노산들의 조합을 통해 원자 수준에서 분자의 생화학적 특성을 조절하여 약물로서 사용될 수 있는 가능성 때문에 오랫동안 다양한 방식으로 생산되어왔다.
초기 펩타이드 의약품 생산은 생명체에서 합성된 단백질을 직접 취하는 방식으로 이루어졌다. 예를 들어, 1920년대에 최초로 생산된 인슐린(51개 아미노산)은 개의 췌장에서 직접 추출하여 사용하였다. 하지만, 이후 생명공학의 발전과 함께 1970-80년대를 지나면서 재조합
DNA 기술을 이용한 방식으로 단백질기반 의약품의 생산방식이 변화하였고, 이와 더불어 대량생산도 가능하게 되었다(그림 1A).하지만, 단백질보다 짧은 구조를 지닌 선형 펩타이드는 다음과 같은 이유로 인하여일반적으로 약리학적 물질로 사용되기 어렵다. 첫째, 펩타이드는 구조적으로 너무 유연하여 표적 분자와 효과적인 상호 작용을 하지 못한다. 둘째, 펩타이드는 세포막을 통과하기 어렵고, 세포막을 투과하여 세포 내로 진입하더라도 세포 내에 존재하는 단백질 분해효소에 대한 저항성이 낮아 약리 효능을 발휘하기도 전에 분해될 가능성이 높다. 셋째, 비록 20가지의 천연아미노산을 이용하여 다양한 조합의 펩타이드 생성이 가능하더라도, 세포막을 효율적으로 투과하는 펩타이드의 길이가 약 5-15개 내외의 아미노산으로 한정된다는 점을 감안하면 다양한 생화학적 특성을 표현하기에 그 범위가 제한되어 있다.
이러한 문제점을 해결하기 위한 방법으로 펩타이드모방분자(peptidomimetics)가 제시되었다. 첫째, 펩타이드모방분자는 펩타이드와 달리 구조적 차이를 보인다. 단순 선형 구조에서 벗어나 부분 또는 전체에 고리(원형)구조를 포함한다는 것이다. 이 고리 구조는 펩타이드 분자 내에 강직도(rigidity)를 제공하고 엔트로피 손실을 최소화하여 표적분자와의 상호작용을 높이는 효과를 가져온다. 둘째, 펩타이드모방분자는 펩타이드와 달리 비천연아미노산을 포함하여 합성할 수 있는데, 이는 생화학적 특성과 구조의 범위가 사실상 무한하다는 의미이다. 이로 인해, 펩타이드모방분자는 기존의 펩타이드에 비해 세포벽 투과능력과 세포 내 단백질 분해효소에 의한내성을 동시에 향상시킬 수 있어 더 나은 약물 효능을 제공할 수 있다.
따라서 본론에서는 펩타이드모방 물질의 합성 및 선별을 위해 최근에 사용되고 있는 무세포단백질합성(cell-free protein synthesis, CFPS) 시스템에 대해 이야기할 것이다. 세부적으로, CFPS 시스템의 특징, 펩타이드모방물질 선별을 위한 다양한 in vitro 디스플레이 기술, 그리고 CFPS 플랫폼을 통하여 비천연아미노산을 포함하도록 생산된 여러가지 펩타이드모방 물질의 특징에 대해 기술하고자 한다(그림 1 B).
2. 본론
2.1. CFPS의 활용 1) 비정규 단량체를 포함한 펩타이드모방 분자의 형성
현재까지 신약개발은 대부분 화학적으로 합성된 소분자(small molecule, 분자량: 100-1,000Da) 형태의 분자를 선별함 으로써 이루어졌다. 이렇게 합성된 소분자약물(small molecule drug)은 일반적으로 수동확산(passive diffusion)을 통해 세포벽을 투과하는 데 우수하여 세포 내 약물 전달에 탁월한 능력을 가지고 있지만, 약물의 작은 표면적으로 인해 표적 단백질과 제한된 영역에서만 상호작용을 하게 되어 종종 그 효과가 극대화되지 못한다고 알려져 있다. 의약품 중 가장 많은 부분을 차지하고 있던 소분자약물의 승인 수가 매년 일정하거나 감소하는 추이를 보이는 최근의 데이터는 [1,2],
이러한 과학적 사실을 반증하는 것이라 생각한다.
소분자약물을 대체하기 위한 신약개발대상 물질로 위에서 언급한 펩타이드가 언급된다. 이러한 고분자들은 우선 표적 단백질과 더 큰 접촉 영역을 제공하며, 표적 물질에 대한 뛰어난 반응 특이성을 보인다, 또한 복잡한 생물학적 반응조건에서도 표적 반응이 정확하고 신속하다. 여러가지 화학 작용기를 가진 비천연 단량체를 위치 특이적으로 포함함으로써 고분자의 생화학적 특성 또한 다양화 될 수 있다.
세포 내에서 단백질 합성에 관여하는 효소를 시험관으로 옮겨 펩타이드 또는 단백질을 합성하는 CFPS 플랫폼 [3,4]은 다양한 유형의 바이오고분자 합성을 위한 이상적인 기술로 제시된다. 이 시스템은 리보솜(ribosome)의 단백질합성효율(99.999%의 정확도로 1초에 20개 아미노산을 펩타이드로 연결)을 이용한 시스템으로서, 고체상합성법을 이용한 펩타이드 합성법보다 정확하고 빠르게 다양한 펩타이드를 합성할 수 있다는 장점이 있다. 또한 이 시스템은 새로운 물질의 합성을 위해 비천연물질을 첨가하여도 세포 독성으로 인한 세포 사멸을 고려할 필요가 없기 때문에 세포 내에서 합성이 불가능한 다양한 형태의 비천연 아미노산을 단백질 내에 도입시킬 수 있다. 하지만 비천연아미노산이 기존의 단백질 번역시스템에 의해 단백질 내로 삽입되기 위해서는 반드시 tRNA (transfer RNA)에 결합하는 반응이 선행되어야 한다. 본래 이 과정은 세포 내에서 aminoacyl-tRNA synthetase (aaRS)라는 촉매에 의해 발생하지만, 이 효소는 20개 아미노산에 대해 높은 기질특이성(substrate specificity)을 가지고 있기 때문에 비천연 아미노산을 인식하지 못하여 아미노산을 tRNA와 결합시키지 못한다. 최근 비천연 아미노산을 tRNA와 연결시키는 많은 방법 중 flexizyme (Fx)이라는 리보자임(ribozyme)을 이용한 방법이 주목을 받고 있다. 이 리보자임은 SELEX (systematic evolution of ligands by exponential enrichment)라는 시스템 [5]에서 선별진화한 RNA 촉매로,
carboxylic acid를 포함하는 기질에 대하여 아실화반응(acylation)을 진행하여 tRNA의 3’-말단에 결합시키는 생화학적 반응특성이 최적화된 촉매이다 [6]. 이는 기질 내에 Fx가 인식할 수 있는 적절한 방향족이 존재하기만 하면 수많은 형태의 비천연 아미노산도 tRNA에 결합될 수 있다는 것을 의미한다.
동경대의 Hiroaki Suga는 이러한 Fx을 이용하여 다양한 형태의 비천연 아미노산을 tRNA에 결합시키고 무세포시스템에서 펩타이드 혹은 단백질에 비천연아미노산을 삽입하는 연구를 진행해왔다. 최근 우리 연구실에서도 기질의 분자구조에 따라 tRNA의 결합 수율을 예측할 수 있는 디자인 규칙을 개발하여 더욱더 넓은 범위의 비천연 아미노산을 단백질의 구성요소로 사용할 수 있는 기반을 마련하였다 [7]. 20개의 아미노산에 국한되어 있던 펩타이드를 생화학적 특성을 무한대로 확장하여 합성할 수 있는 기회가 마련된 것이다.
2.2. CFPS의 활용 2) 디스플레이 기술과 통합하여 약물특성을 갖는 펩타이드모방분자 발굴
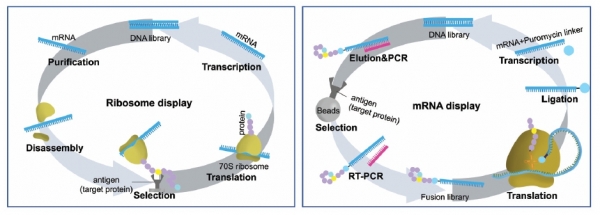
디스플레이 기술(그림 2)은 고정된 표적 단백질에 대해 높은 친화도와 특이성을 가진 후보 분자를 선별하는 기술이다.
리보솜(ribosome) 디스플레이 또는 mRNA 디스플레이와 같은 기술은 일반적으로 약 1012-1014에 달하는 펩타이드 중에서 표적 단백질과 반응하는 ‘최적’의 분자를 선별할 수 있는 기술이다 [8]. 리보솜 디스플레이(그림 2)는 표적단백질에 결합하는 펩타이드를 발견하기 위하여 펩타이드-리보솜-mRNA의 3차 착물(ternary complex) 형성 반응을 응용하는 것이 핵심이다. In vivo 단백질 번역 과정에서는 리보솜이 mRNA의 정지코돈(stop codon)에 도달했을 시 release factor가 리보솜에 결합하여 리보솜을 해체시켜 단백질 번역과정을 완료하지만,
in vitro에서는 release factor가 제거된 환경을구축하면 리보솜이 정지코돈에서 해체되지 못해 mRNA와 초기 펩타이드 사슬에 결합되어 있는 상태로 정지하게 된다.
따라서, 각 변이체의 유전형(mRNA)과 표적에 결합된 표현형(펩타이드)이 리보솜을 중심으로 결합된 형태를 생산할 수 있다. 리보솜 밖으로 빠져나온 펩타이드를 이용하여 선별된 유전자를 포획(capture)하고, 이를 RT-PCR 및 DNA로 증폭하는 과정을 반복적으로 시행하면 우세 선별군을 확보할 수 있다.
mRNA 디스플레이(그림 2)는 리보솜 디스플레이로 생성된 3차 착물에서 펩타이드와 유전자를 공유 결합으로 연결하는 기술이다. 최근
Suga가 개발한 RaPID (Random non-canonical Peptide Integrated Discovery)는 이 mRNA 디스플레이 기술을 기반으로 한 시스템으로 비천연아미노산을 삽입한 펩타이드모방물질의 선별을 위해 설계되었다. 즉, RaPID시스템은 in vitro에서 Fx 시스템과 mRNA 디스플레이 플랫폼을 통합하여 비천연아미노산을 펩타이드모방 라이브러리에 포함시켜 선별하는 것이 핵심이다. 이를 통해 일반적인 post-
transcriptional modification으로 생성되지 않는 다양한 작용기를 펩타이드 내에 도입하고, 거대고리(macrocycle) 형태의 펩타이드 또는 원형모티프들을 생성하여, 약물로서의 효과를 극대화시킨다. 실제 RaPID를 이용하여 개발된 펩타이드모방분자들은 다양한 표적 단백질을 대상으로 현재 약물로 개발되고 있다 [9].
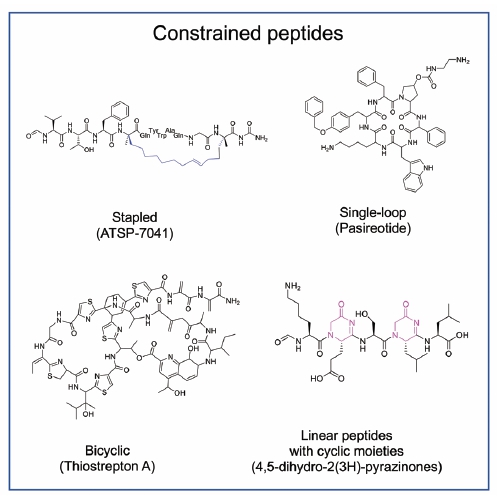
2.3. CFPS의 활용 3) 치료물질로 사용되는 고리형 펩타이드
일반적으로 약 4-20개의 단량체를 포함하도록 디자인된 고리형 펩타이드모방분자들은 면역원성(immunogenicity)의 위험이 낮고, 높은 세포벽 투과성을 유지하며, 표적 선별성(target selectivity)을 부여한다고 알려져있다[10,11]. 이를 위해 다양한 형태의 고리화작업이 이루어진다. 가장 단순한 고리 구조는 단일고리(monocycle)의 형태이며, 또 다른 구조는 안정적인 2차 구조를 형성하도록 중추구의 회전을 고정시킨 형태의 스테이플(staple) 구조이다. 추가적으로 개선된 형태는 이중 고리(bicycle) 형태인데, 이 구조는 펩타이드 분자의 중심에 3방향 접합을 포함하는 2개의 루프를 이용하여 합성된다. 이중고리 구조는 단일 고리형 구조보다 더 큰 구조적 경직성과 단백질 분해 안정성을 나타내어 펩타이드의 결합 친화도와 분해 반감기를 증가시킬 수 있다고 알려진다 [12].
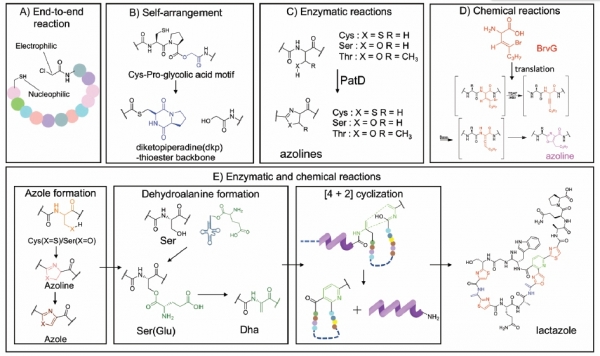
2.4. CFPS의 활용 4) 비천연물질을 이용한 새로운 고분자 중추구조 합성
위에 언급한 다양한 고리구조를 생성하기 위한 연구들은 주로 Suga 그룹에서 진행되고 있고 그 발전 동향을 파악하기 용이하다. 대표적인 고리형태로 서로 반응성이 있는 두 개의 비천연 아미노산이 선형 펩타이드의 N-말단과 C-말단에 삽입된 형태이다(그림 4A).
이들은 intramolecular reaction을 통해 쉽게 공유 결합으로 연결된 고리구조의 펩타이드를 형성한다. 예를 들어 chloroacetyl 작용기와
Cysteine을 펩타이드 내에 포함시키면 thioether 결합을 형성한다 [13,14].
이후, 이러한 발전을 기반으로 Suga 그룹은 분자 스스로 고리를 형성하는 방식을 이용하여 두 개의 고리로 구성된 중추 구조를 구현하였다. 즉, 펩타이드의 중간에 삽입된 Cys-Pro-Glycolic acid의 모티프가 자기 재배열(self-rearrangement)을 통해 diketopiperadine (dkp)-
thioester 중추구조를 변환한다 [15] (그림 4B). Azoline은 5개의 아미노산으로 이루어진 헤테로고리(heterocycle) 형태를 가지는 화합물로, cyanobactin과 같은 항암, 항바이러스 분자는 펩타이드 내에 이러한 azoline을 중추구조를 4개나 포함한다 (그림 4C). Cyanobactin은 세포에서 리보솜에 의해 시스테인, 세린 및 트레오닌을 포함하는 전구체 펩타이드로 합성된 후 azoline 생성 효소인 PatD에 의해 변형되는데, 최근 연구에서 Suga 그룹은 전구체와 PatD 효소가 보완된 CFPS 플랫폼을 이용하여 효율적으로 azoline 그룹을 생산할 수 있는
‘one-pot’ 시스템을 구축했다. 후속 연구에서는, 펩타이드 사슬에 4-bromovinylglycine 기를 포함한 비천연아미노산을 이용하여(dehydrobromination을 거친 azoline 형성) azole 고리를 추가 효소의 첨가없이 바로 합성할 수 있는 시스템으로 발전시켰다 (그림 4D).
이어진 후속 연구에서는 위에서 기술한 분자 내 고리화 생성 전략들을 조합하여 하나의 펩타이드 내에 서로 다른 여러가지의 고리형태를 생성하는 기술을 개발하였다. 즉, azole과 pyridine을 형성하는 두 개의 이종고리를 함께 도입하여 lactazole과 같은 thiopeptide 계열의 펩타이드모방분자를 합성하였다 [16] (그림 4E). 이처럼 CFPS는 다양한 화합물을 특정 위치에 삽입시켜 고리화시킬 수 있는 장점이 있어 펩타이드 모방물질의 합성범위를 대폭 확장시키는 중요한 플랫폼이라 할 수 있다.
3. 결론 및 전망
CFPS가 펩타이드모방분자를 효율적으로 개발해내는 데, 획기적인 역할을 해 온 것은 틀림없지만, 여전히 해결해야 할 숙제들이 존재한다. 첫째, in vitro 환경에서 선별된 펩타이드모방분자가 세포 내에 진입했을 때 미치는 영향에 대한 정보(단백분해저항성 및 숙주 세포에 대한 독성 등)가 여전히 부족하여 in vivo에 실험이 반드시 수반되어야 한다. 둘째, 라이브러리에서 펩타이드모방 물질이 선별되는 것은 주로 표적단백질에 대한 분자 친화력(affinity)에 의해 결정되는 것이므로, 실제 세포 내에서 표적분자에 대한 높은 친화력이 반드시 표적분자 활성도저하로 귀결될지에 대해서는 정확하게 예측할 수 없다. 셋째, 라이브러리의 크기는 1012-1014로 일반적인 in vivo 디스플레이 시스템
(109-1010) 라이브러리보다 그 크기가 크지만, 단순히 라이브러리의 크기가 크다고 약물 발견의 확률도 높아지는 것은 아니다. 이는 비천연아미노산이 무작위로 삽입될 경우 오히려 표적 단백질과의 결합친화도가 감소하는 라이브러리가 생성될 가능성도 존재하기 때문이다. 마지막으로, CFPS로 생산되는 생산물의 수율은 일반적으로 μg mL-1 [17]에서 ~1 mg mL-1 [18] 정도로낮다. 이렇게 낮은 생산량은 시장의 수요를 충족하기 어렵기 때문에 대량 생산을 위한 화학 합성의 개발이 수반되어야한다.
이러한 어려운 점에도 불구하고 비천연 아미노산의 도입이 자유로운 CFPS 플랫폼은 약리학적으로 이용될 수 있는 펩타이드모방 분자의 개발 및 생산을 가속화하는 데 중요한 역할을 할 것으로 기대된다. 특히, 비천연 중추구조(펩타이드 결합제외)가 내재된 펩티드모방분자의 합성모델로 매우 적절할 것이며, 최근 부각되고 있는 컴퓨터 모델링 기술들과 통합될 경우 보다 정밀한 펩타이드모방물질의 선별에 기여할 것이다.
참고문헌
[1] A. R. Spiegelman(1964) Synthetic Vasopressin and Diabetes Insipidus. JAMA. 187: 1035.
[2] I. S. Johnson(1983) Human insulin from recombinant DNA technology. Science. 219: 632-7.
[3] Y. Shimizu, A. Inoue, Y. Tomari, T. Suzuki, T. Yokogawa, K. Nishikawa and T. Ueda(2001) Cell-free translation
reconstituted with purified components. Nat Biotechnol. 19: 751-5.
[4] S. S. Yim, N. I. Johns, J. Park, A. L. Gomes, R. M. McBee, M. Richardson, C. Ronda, S. P. Chen, D. Garenne, V. Noireaux
and H.H. Wang(2019) Multiplex transcriptional characterizations across diverse bacterial species using cell-free systems.
Mol Syst Biol.15: e8875.
[5] J. Morimoto, Y. Hayashi, K. Iwasaki and H. Suga(2011) Flexizymes: their evolutionary history and the origin of catalytic
function. Acc Chem Res. 44: 1359-68.
[6] H. Murakami, A. Ohta, H. Ashigai and H. Suga(2006) A highly flexible tRNA acylation method for non-natural polypeptide synthesis.Nat Methods. 3: 357-9.
[7] J. Lee, K. E. Schwieter, A. M. Watkins, D. S. Kim, H. Yu, K. J. Schwarz, J. Lim, J. Coronado, M. Byrom, E. V. Anslyn, A. D.
Ellington, J. S. Moore and M. C. Jewett(2019) Expanding the limits of the second genetic code with ribozymes. Nat Commun. 10: 5097.
[8] M. J. Hammerling, B. R. Fritz, D. J. Yoesep, D. S. Kim, E. D. Carlson and M. C. Jewett(2020) In vitro ribosome synthesis and evolution through ribosome display. Nat Commun. 11: 1108.
[9] Y. Goto and H. Suga(2021) The RaPID Platform for the Discovery of Pseudo-Natural Macrocyclic Peptides. Acc Chem Res. 54:3604-3617.
[10] H. Peacock and H. Suga(2021) Discovery of De Novo Macrocyclic Peptides by Messenger RNA Display. Trends Pharmacol Sci.42: 385-397.
[11] L. Plais and J. Scheuermann(2022) Macrocyclic DNA-encoded chemical libraries: a historical perspective. RSC Chem Biol. 3:7-17.
[12] C. A. Rhodes and D. Pei(2017) Bicyclic Peptides as Next-Generation Therapeutics. Chemistry. 23: 12690-12703.
[13] C. Heinis, T. Rutherford, S. Freund and G. Winter(2009) Phage-encoded combinatorial chemical libraries based on bicyclic peptides. Nat Chem Biol. 5: 502-7.
[14] S. W. Millward, T. T. Takahashi and R. W. Roberts(2005) A general route for post-translational cyclization of mRNA display libraries. J Am Chem Soc. 127: 14142-3.
[15] T. Kawakami, A. Ohta, M. Ohuchi, H. Ashigai, H. Murakami and H. Suga(2009) Diverse backbone-cyclized peptides via
codon reprogramming. Nat Chem Biol. 5: 888-90.
[16] A. A. Vinogradov, M. Shimomura, Y. Goto, T. Ozaki, S. Asamizu, Y. Sugai, H. Suga and H. Onaka(2020) Minimal
lactazole scaffold for in vitro thiopeptide bioengineering. Nat Commun. 11: 2272.
[17] E. G. Worst, M. P. Exner, A. De Simone, M. Schenkelberger, V. Noireaux, N. Budisa and A. Ott(2015) Cell-free expression with the toxic amino acid canavanine. Bioorg Med Chem Lett. 25: 3658-60.
[18] C. Albayrak and J. R. Swartz(2013) Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific nonnatural amino acid incorporation. Nucleic Acids Res. 41: 5949-63.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update