| Region-of-interest 기반 공간 오믹스 기술 | |
|---|---|
| Date 2023-04-13 01:00:49 |
|

1. 서론
공간 오믹스(Spatial omics)는 조직 내 세포 및 소세포 수준에서 분자 정보를 식별하고 분석할 수 있는 최첨단 기술이다 [1]. 이 기술은 발달, 질병 진행, 치료에 대한 반응 등 다양한 생물학적 과정에 관여하는 분자 메커니즘을 포괄적으로 이해할 수 있다는 점에서 과학계에서 큰 주목을 받고 있다. 공간 오믹스 접근법에는 생물학적 샘플을 공간적으로 분해하여 분석할 수 있는 다양한 기술이 사용된다. 현재 공간 오믹스에서 가장 널리 이용되고 있는 기술은 공간 전사체학(Spatial transcriptomics) 으로, 고처리량 RNA 시퀀싱(RNA-seq)과 공간 분해 캡처 기술 등을 활용하여 공간 분해 전사체 데이터를 생성하는 기술이다 [2]. Spatial transcriptomics는 조직의 특정 영역에서 유전자의 발현을 식별하여 조직 내에서 일어나는 분자 과정에 대한 고해상도 뷰를 제공한다.
2020년도 Nature Methods 지에서 “Method of the year”로 선정되며 spatial transcriptomics가 공간 오믹스 분야에 크게 기여했음이 강조되었다 [3]. 공간 전사체학은 조직 내에서 일어나는 분자 과정을 고해상도로 볼 수 있고 다양한 질병에 대한 새로운 바이오마커와 치료 표적을 식별할 수 있어 다양한 분야의 연구자들에게 유용한 도구로 활용되고 있다. 이처럼 차세대 염기 서열 분석(Next Generation Sequencing, NGS)을 활용하여 Spatial genomics, spatial epigenomics, spatial epitranscriptomics 등 공간정보를 유지하며 오믹스 분석을 수행할 수 있는 기술들은 생물학적 샘플에 대한 새로운 정보를 제공할 수 있다. 공간 단백질체학(Spatial proteomics) 은 공간 오믹스의 또 다른 새로운 분야로, 조직 내 단백질의 분포와 발현량을 정량화하는 것이다 [4].
Spatial proteomics 기술은 이미징과 질량 분석법을 결합하여 공간적으로 분해된 proteomics 데이터를 생성한다. 이러한 데이터는 세포와 조직 내에서 일어나는 복잡한 분자 상호작용에 대한 통찰을 제공하여 다양한 질병에 대한 새로운 바이오마커와 치료 표적을 식별할 수 있게 해준다. 공간 대사체학(Spatial metabolomics) 은 비교적 새로이 개발된 공간 오믹스 분야로, 조직 내 대사산물의 식별과 정량화를 포함한다 [5]. Spatial metabolomics 기술은 이미징 질량 분석법이나 항체-DNA 바코드 결합체를 사용하여 공간적으로 분해된 대사체 데이터를 생성한다. 이러한 데이터는 세포와 조직 내에서 일어나는 대사 과정을 분석하여 다양한 생물학적 과정에서 중요한 역할을 하는 대사 경로를 식별할 수 있게 해준다. 공간 오믹스 기술들은 Region-of-interest (ROI) 기반 그리고 Non-ROI 기반 공간 오믹스 기술들로 분류될 수 있다. 기술들의 장단점을 이해한다면 연구에 활용될 수 있는 잠재력이 매우 높다. 본 최신 연구동향에서는 여러 공간 오믹스 기술들 중 ROI-기반 공간 오믹스 기술, 특히 SLACS (Spatially-resolved Laser Activated Cell Sorting)을 활용한 연구동향을 소개하고 이의 잠재적 활용법을 소개한다.
2. 본론
2.1. Non-ROI 기반 공간 오믹스
대량 공간 오믹스라고도 하는 non-ROI 기반 공간 오믹스는 세포나 아세포 구조의 공간적 위치를 고려하지 않고 전체 조직의 분자 데이터를 분석하는 방식이다. 이 접근 방식은 2020년도 Nature Methods에 의해 Method of the year로 선정된 이후 유전체학 및 단백질체학 분야에서 상당한 발전을 가져왔다. 그러나 non-ROI 기반 공간 오믹스는 조직과 세포의 공간적 이질성을 포착하는 데 한계가 있다. non-ROI 기반 공간 오믹스는 전체 조직 넓은 영역의 분자적 특성에 대한 평균적인 뷰를 제공하기 때문에 조직의 다른 영역에 있는 세포 간 분자 패턴의 중요한 차이를 모호하게 만들 수 있기 때문이다. 특히 non-ROI 기반 공간 오믹스는 각 기술별로 전사체만 분석할 수 있거나 [2] 후성 유전체만 분석이 가능한 [6] single modality 분석만 가능하기 때문에 다중 오믹스가 가능한 ROI 기반 접근 방식에 비해 동일 기술로 다각도로 생물학적 샘플을 분석하는 것이 불가능하다. 따라서 다른 modality를 더할 수 있는 별개의 기술을 추가적으로 도입해야하는 단점이 존재한다 [7].
2.2. ROI 기반 공간 오믹스
조직 내 특정 위치에 대하여 심화 분석을 할 수 있는 기술인 ROI 기반 공간 오믹스 기술의 발전은 연구자들이 생물학적 시스템의 공간적 조직을 더 잘 이해할 수 있게 해줌으로써 생물학 분야에 큰 변화를 가져왔다. ROI 기반 공간 오믹스를 사용하면 조직의 특정 영역 내에서 세포 및 아세포 수준의 분자 정보를 식별하고 분석할 수 있다. 이 접근 방식은 조직 내에서 일어나는 분자 과정에 대한 고해상도 뷰를 제공하며, 조직의 특정 영역 내에서 유전자 발현, 단백질 위치, 대사 물질 분포를 식별할 수 있게 해준다. ROI 기반 공간 오믹스는 조직과 세포의 공간적 이질성을 포착함으로써 세포와 조직 내에서 일어나는 복잡한 분자 상호작용에 대한 통찰력을 제공하고, 다양한 질병에 대한 새로운 바이오마커와 치료 표적을 식별할 수 있게 해준다.
ROI 기반 공간 오믹스의 주요 장점 중 하나는 생물학적 시스템의 공간적 조직 중 원하는 영역들만을 포착할 수 있다는 점이다. 조직의 특정 영역 내에서 발현한 분자 정보를 식별하고 분석함으로써 조직과 세포의 분자 이질성을 더욱 정확하게 표현하고, 특정 세포 기능 또는 질병 상태와 관련된 분자 패턴을 식별할 수 있다. 또한 ROI 기반 공간 오믹스를 사용하면 조직 내에서 희귀한 세포 유형을 식별할 수 있어 다양한 질병의 발병과 진행을 이해하는 데 중요하다.
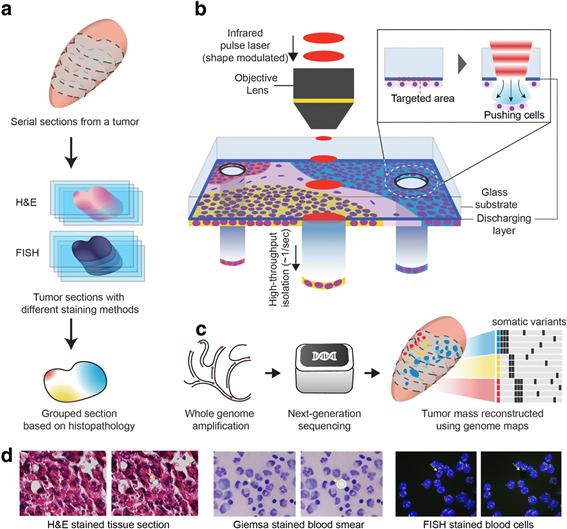
ROI 기반 공간 오믹스의 또 다른 장점은 동일한 조직 영역에서 다중 오믹스 데이터를 생성할 수 있다는 점이다. 예를들어, 연구자들은 같은 ROI 기반 기술로 공간 전사체학과 공간 단백질체학을 결합하여 조직의 특정 영역 내에서 유전자 발현과 단백질 국소화 사이의 관계를 파악할 수 있다 [8]. 이를 통해 다양한 생물학적 과정에 관여하는 새로운 조절 메커니즘과 신호 경로를 파악할 수 있다.
ROI 기반 공간 오믹스의 장점에도 불구하고 이 접근 방식에는 몇 가지 한계가 존재한다. ROI 기반 공간 오믹스를 위한 샘플 준비는 관심 있는 조직 영역을 세심하게 해부해야 하는데 [9], 이는 일부 조직 유형이나 실험 시스템에서는 어려울 수 있다. 또한 ROI 기반 공간 오믹스를 위한 이미징 기술에는 높은 공간 해상도와 감도가 필요하므로 비용과 시간이 많이 소요될 수 있다.
이러한 문제를 해결하기 위해 연구자들은 현재 공간 오믹스 데이터 분석을 위한 새로운 컴퓨팅 방식과 알고리즘을 개발하고 있다. 예를 들어, 머신러닝 접근법을 사용하면 조직 샘플 내에서 관심 영역을 자동으로 식별하여 수동 해부의 필요성을 줄일 수 있다 [10]. 또한 딥러닝 접근법을 사용하여 조직 고정 및 처리 프로토콜, 이미징 조건, 데이터 정규화 방법의 다양성을 고려하여 공간 오믹스 데이터 분석의 정확성과 재현성을 향상시킬 수 있다.
위와 같이 ROI 기반 공간 오믹스는 주로 레이저 미세절개 (laser microdissection) 기술들을 위주로 발달되어 왔으며, 최근 들어 광학을 이용한 GeoMx 등과 같이 ROI를 선정하여 오믹스를 수행하는 기술들이 등장하고 있다. 특히, Spatially-resolved Laser Activated Cell Sorting (SLACS) 기술은 미세절개 절차 없이 원적외선 펄스레이저를 ROI에 조사하여 원하는 세포들을 빠르게 분리할 수 있는 기술이다. 본 기술을 사용하여 공간 유전체 (Spatial genomics) [11,12], 공간 전사체 (Spatial transcriptomics) [13] 등 진단 및 제약에 활용될 수 있는 ROI 기반 중개의학 연구들이 보고되고 있다.
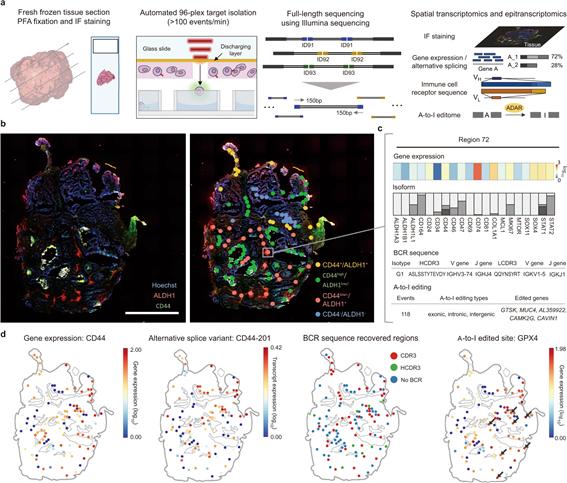
특히 ROI 기반의 방식은 조직 내 특정 영역에서 심화 분석의 수행이 가능하기 때문에, 기존 기술이 분석하지 못하는 새로운 분야의 분석이 가능하다. 공간 후성전사체학(Spatial epitranscriptomics) 은 조직의 특정 영역 내에서 RNA 변형을 식별하고 분석하는 공간 오믹스의 새로운 분야이다 [13,14]. 암 연구에서 공간 후성 전사체학을 적용한 최근 사례로는 2022년 Nature Communications에 발표된 연구가 있다. 이 연구에서 연구자들은 유방암 조직 내 암 줄기세포 마이크로니치(microniches) 에서 A-to-I RNA 편집을 조사하기 위해 공간 전사체학을 사용하였다. 연구진은 암 줄기세포 마이크로니치 내의 후성전사체를 분석하여 줄기세포 유지 및 분화와 관련된 특정 A-to-I editing 현상을 확인할 수 있었다.
이러한 발견은 유방암 줄기세포 기능에 관여하는 분자 메커니즘에 대한 새로운 통찰력을 제공하며, 유방암에 대한 새로운 표적 치료법 개발에 중요한 영향을 미칠 수 있다. 이의 중요성이 강조되어 2023년도에 Nature Reviews Cancer지에서 공간 후성전사체학의 주요성을 알리며 기술을 “Tools of the trade”로 선정하였다 [14].
공간 오믹스 기술은 암 연구를 포함한 다양한 생물학 분야에서도 널리 사용되고 있다. 예를 들어, 신경과학에서 공간 전사체학은 뇌의 특정 영역 내에서 유전자 발현 패턴을 조사하는 데 사용되어 뇌 발달과 기능에 관련된 분자 메커니즘에 대한 통찰력을 제공한다. 마찬가지로 발달 생물학에서도 공간 오믹스 기술은 다양한 조직과 기관의 발달에 관여하는 분자 메커니즘을 조사하는 데 사용되어 기관 발생의 분자적 기초에 대한 새로운 통찰력을 제공한다.
공간 오믹스 기술을 다른 새로운 기술과 결합하여 건강과 질병의 분자적 기초에 대한 보다 포괄적인 이해를 제공하기 위해 지속적인 연구 노력이 이루어지고 있다. 공간 오믹스 기술과 단일 세포 유전체학 및 이미징과 같은 다른 첨단기술의 통합은 다양한 생물학적 과정에 관여하는 분자 메커니즘에 대한 이해를 크게 발전시키고 광범위한 질병에 대한 새로운 진단 및 치료 접근법 개발에 중요한 영향을 미칠 것으로 예상된다.
3. 결론
결론적으로, ROI 기반 공간 오믹스 기술은 조직 내 세포 및 조직 수준에서 분자 정보를 식별하고 분석할 수 있는 강력한 접근 방식이다. 이러한 기술은 다양한 생물학적 과정에 관여하는 분자 메커니즘에 대한 이해를 증진하고 광범위한 질병에 대한 새로운 진단 및 치료 접근법의 개발을 촉진할 수 있는 잠재력을 지니고 있다. 현재 공간 오믹스 정보를 분석할 수 있는 다양한 기술들이 끊임없이 개발되고 있으며, 다양한 분야에 적용되어 기존 연구 방식으로는 분석하기 힘들었던 세포의 공간적 맥락에 대한 연구를 수행하고 있다. 현재까지 공간 오믹스 연구들이 가진 한계들을 극복하고 공간 오믹스 데이터를 다른 새로운 기술과 결합하여 건강과 질병의 분자적 기초에 대한 보다 포괄적인 이해를 제공하기 위해 지속적인 연구 노력이 이루어지고 있다.
참고문헌
[1] M. Asp, J. Bergenstråhle, and J. Lundeberg, “Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration,”BioEssays, p. 1900221, May 2020, doi: 10.1002/bies.201900221.
[2] P. L. Ståhl et al., “Visualization and analysis of gene expression in tissue sections by spatial transcriptomics,” Science, vol. 353, no.
6294. American Association for the Advancement of Science, pp. 78–82, Jul. 01, 2016, doi: 10.1126/science.aaf2403.
[3] “Method of the Year 2020: spatially resolved transcriptomics,” Nat. Methods 2020 181, vol. 18, no. 1, pp. 1–1, Jan. 2021, doi:10.1038/s41592-020-01042-x.
[4] E. Lundberg and G. H. H. Borner, “Spatial proteomics: a powerful discovery tool for cell biology,” Nat. Rev. Mol. Cell Biol., vol. 20,no. 5, pp. 285–302, May 2019, doi: 10.1038/s41580-018-0094-y.
[5] B. Geier et al., “Spatial metabolomics of in situ host–microbe interactions at the micrometre scale,” Nat. Microbiol. 2020 53, vol. 5,no. 3, pp. 498–510, Feb. 2020, doi: 10.1038/s41564-019-0664-6.
[6] Y. Deng et al., “Spatial profiling of chromatin accessibility in mouse and human tissues,” Nat. 2022, pp. 1–9, Aug. 2022, doi:10.1038/s41586-022-05094-1.
[7] S. M. Lewis et al., “Spatial omics and multiplexed imaging to explore cancer biology,” Nat. Methods 2021, pp. 1–16, Aug. 2021,doi: 10.1038/s41592-021-01203-6.
[8] J. L. G. Niño et al., “Effect of the intratumoral microbiota on spatial and cellular heterogeneity in cancer,” Nat. 2022, pp. 1–8, Nov.2022, doi: 10.1038/s41586-022-05435-0.
[9] V. Espina et al., “Laser-capture microdissection,” Nat. Protoc., vol. 1, no. 2, pp. 586–603, 2006, doi: 10.1038/nprot.2006.85.
[10] Y. Lee et al., “Derivation of prognostic contextual histopathological features from whole-slide images of tumours via graph deep learning,” Nat. Biomed. Eng. 2022, pp. 1–15, Aug. 2022, doi: 10.1038/s41551-022-00923-0.
[11] S. Kim et al., “PHLI-seq: constructing and visualizing cancer genomic maps in 3D by phenotype-based high-throughput laseraided isolation and sequencing,” Genome Biol., vol. 19, no. 1, p. 158, Dec. 2018, doi: 10.1186/s13059-018-1543-9.
[12] O. Kim et al., “Whole Genome Sequencing of Single Circulating Tumor Cells Isolated by Applying a Pulsed Laser to Cell-Capturing Microstructures,” Small, p. 1902607, Jun. 2019, doi: 10.1002/smll.201902607.
[13] A. C. Lee et al., “Spatial epitranscriptomics reveals A-to-I editome specific to cancer stem cell microniches,” Nat. Commun. 2022 131, vol. 13, no. 1, pp. 1–12, May 2022, doi: 10.1038/s41467-022-30299-3.
[14] A. C. Lee, “Epitranscriptomics of cancer microniches,” Nat. Rev. Cancer 2023, pp. 1–1, Feb. 2023, doi: 10.1038/s41568-023-00552-y.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update 
