| 미생물 전사 조절 네트워크 분석을 위한 전사체 대량 데이터 분석 | |
|---|---|
| Date 2023-10-17 12:43:16 |
|

교수
인하대학교 생명공학과
hyungyu.lim@inha.ac.kr
1.서론
미생물 내 유전자들은 주변 환경에 따라 최적 발현되도록 다양한 전사 조절 단백질 (transcription factor, TF)들에 의해 조절되고 있으며, 유전체 레벨에서의 전사 조절 네트워크(transcriptional regulatory network)의 규명은 개별 미생물의 특성을 이해하는 데 필수적이다.
유전 서열 해독 기술은 Next generation sequencing (NGS) 기술의 등장으로 비약적으로 발전하였고, 비용도 약 88만 배 (2001년 $5292/Mb에서 2022년 $0.006/Mb, https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data) 줄어듦의 따라 대중적인 방법으로 자리 잡았다. NGS을 통해 유전 정보를 얻고, 분석함
으로써 다양한 생명체에 대한 이해를 높이고 있다. 이러한 ‘오믹스’ 데이터의 종류로는 관찰하려고 하는 현상에 따라 생산 및 분석 방법이 다르며, 대표적으로 DNA-seq, RNA-seq, ChIP(chromatin Immunoprecipitation)-seq, Tn(transposon)-seq, Ribo(ribosome)-seq 등이 있다.
최근 이들 데이터 중에서, 다양한 환경에서 수집된 대량 전사체 데이터를 활용하여 미생물 내 전사 조절 네트워크(Transcriptional regulatory network, TRN)를 규명하고자 하는 연구가 보고되고 있다. RNA-seq을 활용하여 미생물의 전체 유전자의 발현량을 쉽게 측정할 수 있게 되었기에 가능해진 전략이다. TRN 규명뿐만 아니라 전사체 변화를 개별 유전자 단위가 아닌 전사 조절 단백질(transcription factor, TF)의 조절 단위로 파악하여 생명 현상을 이해할 수 있다는 장점도 지니고 있다. 본 글에서는 이 기술을 간략하게나마 소개하고자 한다.
2.본론
전통적으로 연구자들은 미생물 내 유전자의 발현 조절 기작을 밝히기 위하여, TF의 작동 조건 및 타겟 유전자군 (regulon)을 개별적으로 연구한다. 이때 DNA와 TF 단백질의 상호 작용을 볼 수 있는 DNA electrophoretic mobility shift assay (EMSA), DNA pull-down assays, ChIP-seq, Reporter assay 등 직간접적인 방법들이 이용된다. 이러한 실험 방법들로 TF의 타겟 유전자를 특정하여 레귤론(regulon)을 밝히고, 개별 실험 결과들을 종합하는 상향식(bottom-up) 방법으로 규명되고 있다.
앞서 언급된 개별적인 연구 결과의 종합이라는 상향식 접근 방법과 반대로, 하향식(top-down)으로 게놈 규모의 전사조절 네트워크를 규명할 수 있다 (Sastry et al., 2019). 동일 regulator에 의해 발현이 조절되는 유전자들은 외부 환경이 변하더라도 비슷한 패턴으로 발현량이 변화할 것이기 때문에, 이러한 유전자 그룹을 대규모 전사체 데이터 종합 분석을 통해 특정하는 것을 목표로 한다.
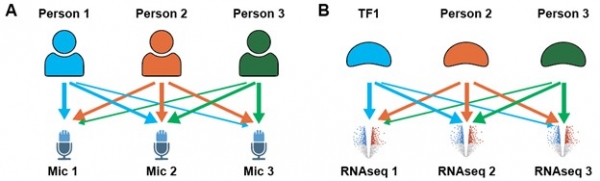
이에 대표적인 방법으로서 기계 학습 알고리즘 중 독립성분분석(Independent component analysis, ICA)이 활용되고 있다. ICA는 혼합된 신호로부터 독립적인 본래의 신호(independent signal)를 식별할 수 있는 blind source separation에 최적화된 기계학습 알고리즘의 하나이다 (Comon, 1994). 이해를 돕기 위하여 여러 사람이 참여하고 있는 행사장 안의 상황을 떠올려 보자 (그림 1B). 파티장안에는 여러 사람이 각자 대화를 나누고 있기에, 여러 사람의 섞인 목소리가 측정된다. 이때 파티장 안에 마이크가 하나 밖에 설치되어 있지 않다면 여러 목소리로부터 개별 사람들의 목소리를 추론하는 것은 매우 어렵다. 하지만 마이크가 여러 개가 설치되어 사람들의 목소리가 다른 세기로 혼합된 여러 신호가 존재할 경우, ICA 알고리즘을 통해 분석, 개별 사람들의 목소리를 분리해 낼 수 있다. 이러한 장점을 가지는 ICA는 실제 통화품질을 증가시키기 위하여 마이크에 같이 잡히는 노이즈 제거에도 활용되고 있다.
앞선 예시를 미생물 유전자 발현에 적용해 보면 (그림 1B), 미생물의 transcriptome은 파티장, TF들을 파티장안의 사람이라고 생각할 수 있으며, TF가 유전자들을 개별적으로 조절하는 세기는 사람들의 목소리에 비유할 수 있을 것이다. 여러 대의 마이크에 서로 다른 세기로 녹음된 원래 목소리를 분리하듯이 유전자 발현 데이터가 충분히 다양한 환경에서 수집되어 있다면, 각 TF들이 개별 타겟 유전자들을 조절하고 전체 유전자 발현 프로파일로부터 개별 조절 시그널을 추출할 수 있다. 이 ICA 알고리즘은 게놈 규모의TRN을 규명하기 위한 다양한 알고리즘(예, K-means, hierarchical clustering, DBSCAN 등) 중 가장 우수한 성능을 보이는 것으로 평가되었다 (Saelens et al., 2018).
그림 1. ICA알고리즘의 적용. (A) ICA는 파티장 안의 목소리의 서로 다른 세기 조합으로부터 개별 목소리 및 각 조합에서의 목소리 크기를 추출한다. (B) ICA는 유전자 발현 프로파일로부터 각 TF가 조절하는 유전자를 특정하고, 조절 세기 정보를 추출한다.
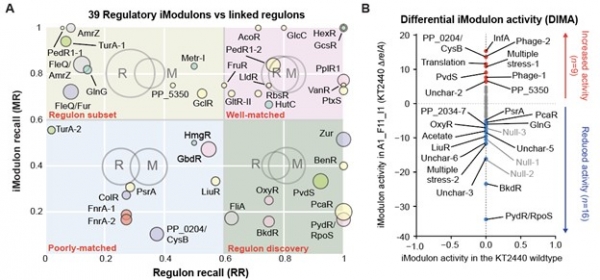
ICA를 통하여 그룹화된 유전자 그룹은 기존의 regulon과 구분하기 위하여 iModulon으로 불리고 있다. 현재까지 ICA는 대장균(Sastry et al., 2019)과 같은 모델 미생물뿐만 아니라 Bacillus subtilis (Rychel et al., 2020), Staphylococcus aureus (Poudel et al., 2020), Pseudomonas putida (Lim et al., 2022) 등 비 모델 미생물에 대해서 축적된 microarray 및 RNA 시퀀싱 데이터에 적용이 되었다. 미생물마다 수집된 데이터 규모의 차이로 iModulon들의 개수가 차이가 있긴 하지만, 각 미생물에서 성공적으로 유전자 발현 패턴이 비슷한 유전자들 그룹을 특정할 수 있었다. 이렇게 특정된 일부 iModulon들의 유전자 membership을 기존regulon과 비교하였을 때, 매우 유사함을 알 수 있었다 (그림 2A). 이는 생물학적 정보에 의존하지 않고도 여러 TF의 기능을 특정하고, 유전체 수준에서의 TRN을 밝히는 것이 가능해짐을 시사한다.
ICA를 통한 co-regulated 유전자 그룹화는 TRN을 밝히는 것뿐만 아니라 복잡한 전사체를 해석하는데 도움을 줄 수 있다. 흔히 전사체의 변화를 파악하기 위하여 differentially expressed genes(DEGs) 분석을 수행하는데, 보통 수백 개 이상의 DEG가 특정되어 복합적인 이해를 하기에 어려운 점이 있다. 하지만 ICA를 통해 미생물 내 발현 패턴이 유사한 유전자들을 이미 그룹화 하였기 때문에, 개별 유전자들의 발현량을 보기보다, 이들 그룹인 iModulon의 activity 변화를 파악함으로써 전체 전사체의 변화를 이해할 수 있다 (그림 2B).
그림 2. P. putida 에서 정의된 iModulon의 비교 분석 및 iModulon을 활용한 전사체 변화 이해. (A) P. putida전사체의 ICA 결과로부터 발굴된 39개의 iModulons 및 이들과 관련된 TF regulon과의 관계. iModulon과 regulon들이 많은 overlap을 가지고 있는 것을 확인할 수 있었으며, 두 유전자 그룹의 차이는 데이터 및 실험 기반 클러스터링의 간의 inconsistency로서 후속 연구가 필요함을 시사함. (B) 전체 전사체 변화를 이해하기 쉽도록, 개별 유전자의 발현 변화가 아닌 iModulon의 활성변화로써 나타낼 수 있음. 이미지 출처: Lim et al., Metab. Eng., 2022, doi: 10.1016/j.ymben.2022.04.004.
3. 결론
최근 NCBI에서 운영하는 GEO (Gene expression omnibus) 에 2023년 8월 기준으로 약 6백만 개의 바이오 샘플들이 등록되어 있으며, 지속적으로 증가하고 있다. 앞으로 바이오파운드리 등 실험실 자동화를 기반으로 생명체의 디자인, 제작, 검증, 분석이 대규모로 가능해 짐에 따라, 오믹스 데이터의 생산은 더욱더 가속화될 것으로 예상된다. 이 과정에서 생산되는 다양한 생물 데이터들을 ICA와 같이 효과적으로 분석할 수 있는 기술에 대한 필요성이 증대되고 있기에, 앞으로의 관련 분야의 연구가 집중적으로 진행되어야 한다. 이런 연구를 통해 궁극적으로 미생물을 포함한 생명체에 대한 이해를 높이고, 다양한 생명공학 산업에 적용하여 인류가 맞닥뜨리고 있는 다양한 문제들을 해결하길 기대해 본다.
참고문헌
Comon, P., 1994. Independent component analysis, A new concept? Signal Processing 36, 287–314.
Lim, H.G., Rychel, K., Sastry, A.V., Bentley, G.J., Mueller, J., Schindel, H.S., Larsen, P.E., Laible, P.D., Guss, A.M., Niu, W., Johnson, C.W., Beckham, G.T., Feist, A.M., Palsson, B.O., 2022. Machine-learning from Pseudomonas putida KT2440 transcriptomes reveals its transcriptional regulatory network. Metab. Eng. 72, 297–310.
Poudel, S., Tsunemoto, H., Seif, Y., Sastry, A.V., Szubin, R., Xu, S., Machado, H., Olson, C.A., Anand, A., Pogliano, J., Nizet, V., Palsson, B.O., 2020. Revealing 29 sets of independently modulated genes in Staphylococcus aureus, their regulators, and role in key physiological response. Proc. Natl. Acad. Sci. U. S. A. 117, 17228–17239.
Rychel, K., Sastry, A.V., Palsson, B.O., 2020. Machine learning uncovers independently regulated modules in the Bacillus subtilis transcriptome. Nat. Commun. 11, 6338.
Saelens, W., Cannoodt, R., Saeys, Y., 2018. A comprehensive evaluation of module detection methods for gene expression data. Nat. Commun. 9, 1090.
Sastry, A.V., Gao, Y., Szubin, R., Hefner, Y., Xu, S., Kim, D., Choudhary, K.S., Yang, L., King, Z.A., Palsson, B.O., 2019. The Escherichia coli transcriptome mostly consists of independently regulated modules. Nat. Commun. 10, 5536.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update