| 차세대 염기서열 분석 (Next-Generation Sequencing)과 암 유전체 분석 | |
|---|---|
| Date 2017-10-09 15:23:09 |
|

교수
삼성서울병원 혈액종양내과/성균관대학교 의과대학
silkahn@skku.edu
1. 서론
차세대 염기서열 분석(Next-Generation Sequencing, NGS)은 기존의 DNA 분석 방법인 생거 염기서열 분석(sanger sequencing)과 달리 대량의 병렬 데이터 생산(Massive parallel sequencing)으로 유전체의 염기서열을 고속으로 분석(highthroughput sequencing)하는 기술을 통칭한다. 2003년 human genome project (HGP)의 완료 이후, 차세대 염기서열 분석 기술은 분석 용량, 속도, 가격 등 양과 질적인 면에서 비약적으로 발전하였다. 이와 더불어 지난 수년간 전장 유전체 연관성 분석(Genome-Wide Association Study, GWAS)을 통해 인간 유전체 전체에 걸쳐 다양한 질환과의 연관성을 나타내는 유전자변이들이 기하급수적으로 발굴되고 있다. 이러한 차세대 염기서열 분석 (NGS) 기술의 발전과 분석 비용의 절감은 의생명 분야에서 차세대 염기서열 분석을 이용한 질병의 원인 규명 및 맞춤의학(Personalized medicine)의 발전으로 이어졌다.
현재 차세대 염기서열 분석(NGS) 기술은 암(cancer)의 연구 및 치료에서 가장 활발히 이용되고 있다. 2010년 이후 the Cancer Genome Atlas (TCGA)와 The International Cancer Genome Consortium (ICGC)같은 대형 염기서열 분석 프로젝트들이 차세대 염기서열 분석(NGS)을 기반으로 다양한 암 종에서 암 유전체(Cancer genomic) 빅데이터를 구축하고 있다. 또한 최근 미국에서는 백 만 명의 환자의 유전체 정보를 분석하여 개인별 맞춤의학 구현을 위한 정보를 수집하는 Precision Medicine Initiative라는 수백만 달러 규모의 장기적인 집단 연구를 진행 중이다[1]. 미국뿐 아니라 전세계적으로 유전체 정보를 이용한 맞춤치료에 대한 연구는 활발히 진행되어 영국에서는 The 100,000 Genomes Project가 진행 중이며, 중국은 베이징 유전체 연구소(Beijing Genomics Institute, BGI)를 중심으로 지금은 전세계 유전체데이터의 20% 이상을 생산하며 유전체 분석 산업에 전폭적인 지원을 아끼지 않고 있다[2, 3]. 우리나라도 맞춤의학 구현을 위한 국제적 수준의 유전체 연구 자원·정보 확보 및 맞춤형 예방·진단·치료기술 개발을 지원하기 위해 2014년~2021년까지 약 5,788억 원을 투입하는 ‘포스트게놈 다부처 유전체사업’을 진행 중이며, 이 사업을 통해 생산된 유전체 데이터를 KOBIC을 통해 통합 관리하여 한국인 유전체 정보를 바탕으로 한 맞춤의학 구현의 근거 기반을 마련하고 있다[4].
차세대 염기서열 분석(NGS) 기술의 발달에 따른 암 유전체 정보의 축적을 통해 암의 진단, 치료 등에 사용될 수 있는 종양 유전자(driver oncogene)가 알려지면서, 이를 빠르게 확인 할 수 있는 차세대 유전체 분석 패널(panel)들이 개발되어 사용되고 있다. 암의 진단 및 치료에 있어 차세대 유전체 분석 패널을 이용한 검사의 임상적 유용성이 폐암, 유방암, 대장암 등에서 밝혀지면서, 유전자 진단법은 암 환자의 조기 진단과 맞춤의학을 위한 검사법으로 대두되고 있다. 우리나라에서도 2017년부터 차세대 염기서열 분석(NGS) 기반 유전자 패널 검사에 건강보험이 적용되어 암의 진단 및 치료에 그 이용이 더욱 증가할 것으로 예상된다. 본고에서는 차세대 염기서열 분석(NGS) 기술에 대해 소개하고, 암 연구에 있어서 차세대 염기서열 분석(NGS) 기술의 사용에 대해 살펴보고자 한다.
2. 본론
2.1 차세대 염기서열 분석(NGS) 기술
현재 사용되는 차세대 염기서열 분석(NGS)은 이전의 생거 방식의 자동화 시퀀싱 기기와 구분하기 위해 사용되는 명칭이다. 이전 방식의 자동화 시퀀싱 기기는 기술적인 진보에도 불구하고 개인의 유전체 분석을 위해 막대한 시간과 비용이 요구되었다. 이러한 한계점을 극복하고 개인의 유전체를 저렴한 비용으로 분석하기 위해 기존 분석 방법에서 병목이 되는 복잡한 과정을 생략하고, 시간이 많이 소요되는 일련의 과정을 한번에 대량으로 처리할 수 있도록 개발된 것이 차세대 염기서열 분석(NGS)이다. 이러한 차세대 염기서열 분석(NGS) 기술은 적은 양의 검체로도 염기서열 분석이 가능하고, 수십 만개의 반응을 동시에 진행할 수 있는 다중화(multiplexing) 능력을 갖추고 있다[5].
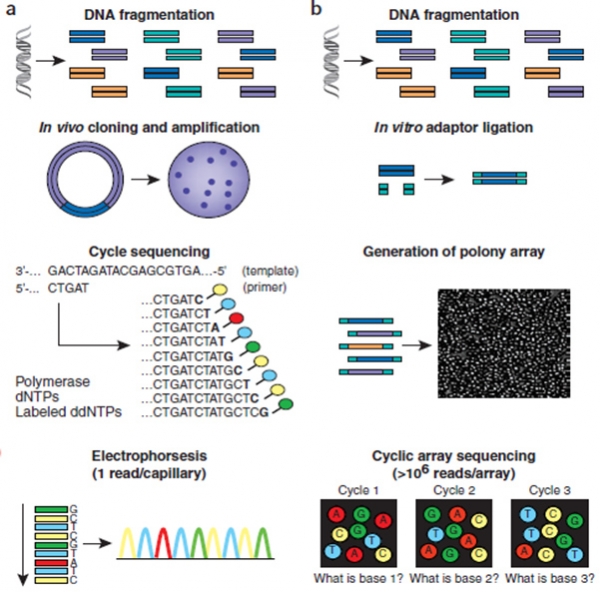
그림 1. 기존 방식 (a) 대 차세대 염기서열 분석 (NGS) (b) 방식의 비교 [5]
2006년 첫 상용화 이후 현재까지 다양한 차세대 염기서열 분석(NGS) 플랫폼이 출시되고 있지만, 현재 일루미나(Illumina), 로슈(Roche), 라이프 테크놀로지(Life Technologies) 등이 전체 차세대 염기서열 분석(NGS) 시장의 대부분을 차지하고 있다. 차세대 염기서열 분석(NGS)은 회사별 기종에 따라 구동방식에 약간의 차이는 있지만 상용화 되어있는 차세대 염기서열 분석(NGS) 장비들은 기본적으로 DNA 서열에 대한 증폭을 한 후, 형광 표식 등의 표지(tag)를 카메라로 찍어 이미지를 처리하는 방식으로 염기서열을 읽어낸다. 차세대 염기서열 분석(NGS) 실험은 크게 1) 클론 증폭(Clonal amplification), 2) 대량병렬법(massively parallel sequencing), 3) 염기서열 결정법(Cyclic sequencing)의 3가지 기술로 이루어진다[6].
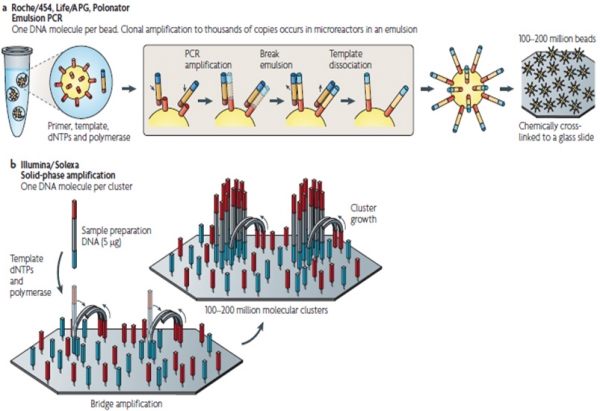
첫 번째 클론 증폭은 주형 클론(clone)을 얻는 과정을 단순화하여 번거로운 library구축과 클로닝(cloning)과정을 없애고 DNA를 짧은 단편으로 직접 자른 다음 PCR로 바로 증폭하여 주형 클론을 얻었다. PCR 방식에 따라 로슈와 라이프 테크놀로지스의 emulsion PCR방식과 일루미나의 solid-phase amplification으로 이루어진다[6].
그림 2. Emulsion PCR (a) 대 solid-phase amplification (b) 방식의 비교 [6]
두 번째 대량병렬법은 수십만 개의 단일 DNA 단편(single DNA fragment)을 공간적으로 분리한 다음 그 자리에서클론을 바로 증폭하거나 염기서열 결정반응을 진행하는 기술이다. 이 경우 로슈와 라이프 테크놀로지스는 emulsion PCR로 증폭된 산물을 대량병렬법으로 정렬하여 염기서열 분석을 진행하나 일루미나의 경우 단일DNA 단편을adaptor와 결합시켜 대량병렬 상태를 갖춘 후 증폭과 염기서열 분석이 이루어진다. 마지막으로 염기서열 결정을 위해차세대 염기서열 분석에서는 생거 염기서열 분석을 탈피하여 작용기전이 다른 염기서열 분석 기법을 사용하는데, 라이프 테크놀로지스는 ligation, 일루미나는 형광 표지 DNA 합성법, 로슈는 pyrosequencing 방법을 이용한다.
2.2 차세대 염기서열 분석(NGS) 기술의 종류
차세대 염기서열 분석(NGS) 기술은 유전자(DNA level), 전사체(RNA level), 후성유전체(Epigenetic level)에 걸친 유전체 전 분야에 대한 분석이 가능하다. 이를 위해 전장 유전체 염기서열 분석(Whole Genome Sequencing, WGS),엑솜 염기서열 분석(Whole Exome Sequencing, WES), 전사체 염기서열 분석(Whole transcriptome sequencing,WTS)등 다양한 분석 플랫폼을 포함한다. DNA상에서 단일 염기서열 변이(Single Nucleotide Variants, SNVs), 삽입/결손(insertion/deletion), 복제수 변이(copy number alteration)등은 전장 유전체 염기서열 분석이나 엑솜 염기서열분석으로, RNA상에서 mRNA의 발현량 변화, 융합 유전자(gene fusion), 선택적 접합(alternative splicing)등은 전사체 염기서열 분석으로 확인할 수 있다. 이러한 분석 방법은 대량 유전체 정보 생산과 새로운 유전체 변이의 발굴을 위해 유용하나 비용적인 부담과 방대한 분량의 데이터 생산으로 인한 데이터 분석과 적용의 어려움, 긴 분석 시간 등의 한계점을 지니고 있어 실제적인 진단용 검사법으로 사용되기에는 적합하지 않다.
이러한 한계점을 극복하기 위한 플랫폼으로 표적 염기서열 분석(targeted NGS)이 대두되었다. 표적 염기서열 분석(targeted NGS)은 전장 유전체 염기서열 분석이나 엑솜 염기서열 분석과 달리 선정된 특정 유전자만을 표적으로 하여 분석하기 때문에 비용 효율성을 높일 수 있고 유전체 분석에 필요한 진단 시간을 줄일 수 있는 방법이다 [7].
이러한 표적 염기서열 분석은 한 염기를 더 많이 읽어 깊이(high depth) 분석하기 때문에 낮은 allele frequency를 더 정확하게(higher sensitivity) 분석할 수 있고, 단일염기서열 변이부터 큰 사이즈의 염색체 재배열까지 분석 가능하다. 또한 전장 유전체 염기서열 분석, 엑솜 염기서열 분석보다 더 많은 검체를 한 번에 분석 가능하고, 표적으로 하는 분석 대상이 명확하기 때문에 검체 효율성이 높아 암 등의 질환에서 소량의 검체를 이용하여 새로운 유전자 변이를 밝히고자 할 때 유리한 이점이 있다. 표적 염기서열 분석을 이용한 진단의 최적화를 위해 표적으로 하는 유전자(진단 및 치료의 바이오 마커 등)에 대한 정보를 잘 알고 목적에 적합한 패널을 디자인하고, 생산된 대량의 데이터를 효과적으로 분석할 수 있어야 할 것이다.
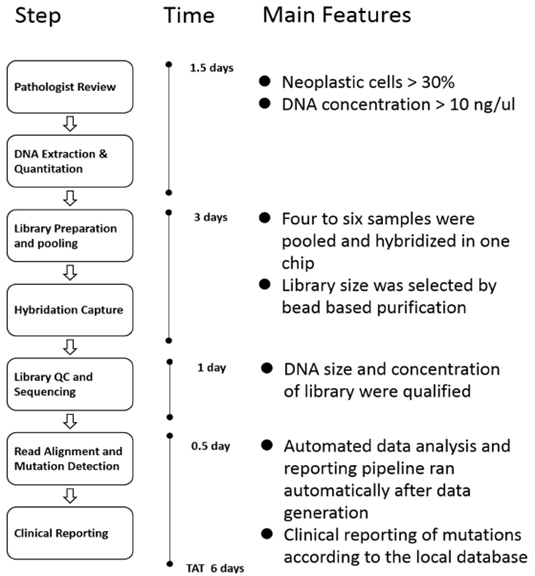
그림 4. 표적 염기서열 분석(targeted NGS)의 과정과 필요 시간 [7]
이러한 다양한 차세대 염기서열 분석(NGS)기술의 발전을 바탕으로 최근에는 하나 하나의 세포에서 유전체 분석을 시행하는 단일 세포 염기서열 분석(single cell sequencing)으로 기술의 발전이 이루어지고 있다. 2009년 단일세포 전사체 분석(single cell whole transcriptome sequencing) 기술이 개발된 이후, 2011년 단일세포 전장 유전체 염기서열 분석(single cell whole genome sequencing), 2012년 단일세포 엑솜 염기서열 분석(single cell whole exome sequencing), 2013년 단일세포 후성유전체 분석(single cell epigenomic sequencing) 기술이 차례로 개발되어 현재 암을 비롯한 다양한 분야에서 활발히 연구가 이루어지고 있다[8].
2.3 차세대 염기서열 분석(NGS)을 이용한 폐암 진단 및 치료
차세대 염기서열 분석(NGS)을 이용한 유전체 정보 기반의 진단 및 치료의 효과는 다양한 암 종에서 보고되고 있으나, 그 중 가장 대표적인 암 종인 폐암에서 그 유용성을 살펴보고자 한다.
폐암(lung cancer)은 우리나라뿐 아니라 전 세계적으로 높은 암 사망률을 보이는 암으로 치료 후의 5년 생존율도 상대적으로 낮은 편이다. 기존에는 폐암을 조직학적 분류(histologic subtype)에 따라 비소세포폐암(non-small cell lung cancer)과 소세포폐암(small cell lung cancer)으로 나누어 일괄적인 치료가 이루어졌다. 하지만 폐암은 비소세포폐암이라는 하나의 조직학적 분류 내에서도 매우 높은 수준의 유전적 비균질성(heterogeneity)을 나타내는 암으로 이러한 단순 분류에 의한 치료로는 그 효과에 한계가 있다. 차세대 염기서열 분석 기술의 발전으로 폐암의 분류는 조직학적 분류라는 기존의 관점에서 벗어나 암의 발병 원인이 되는 종양 유전자의 변이 여부에 따라 분자적 특성에 기반을 둔 아형(molecular subtype) 분류로 발전하였다. 특히 비소세포폐암 중 폐선암(lung adenocarcinoma)에서 이러한 종양 유전자의 변이가 가장 잘 밝혀져 있고 이에 따른 아형 분류가 현재는 진단 및 치료법 결정의 기본이 되고 있다. 1987년 폐선암에서 KRAS mutation이 알려진 이후 20여년의 세월이 흐른 2004년에서야 EGFR mutation이 밝혀져 폐선암의 진단 및 치료에서 EGFR 저해제(EGFR tyrosine kinase inhibitor, EGFR TKI)의 치료 유용성이 알려졌다. 이 후 차세대 염기서열 분석 기술의 발전으로 대규모 염기서열 분석이 이루어지면서 2014년 TCGA에서는 폐선암에서 15개 이상의 유전적 변이에 따른 아형을 보고하였고 이에 기반한 폐암의 진단 및 치료에 대한 연구가 진행되고 있다[9].
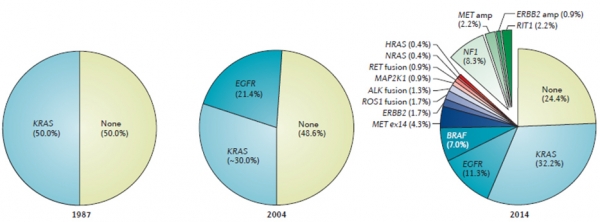
그림 5. 비소세포폐암 중 폐선암에서 다양한 유전적 변이형의 발견 역사 [9]
이러한 유전적 변이 중 임상적 유용성이 검증된 표적항암치료제가 있는 EGFR, ALK, ROS 등은 환자의 진단 시 표적항암치료제 선택을 위한 검사가 이루어지고 있다. 임상에서 사용되는 기존의 검사는 암 조직을 이용하여, EGFR mutation은 PNA clamp방법, ALK fusion과 ROS fusion은 FISH 방법으로 검사한다. 하지만 이러한 검사는 하나 하나의 유전자에 대한 검사가 순차적으로 이루어지기 때문에 많은 시간과 비용이 소요되어 빠른 치료 방법의 결정이 중요한 암 환자에게 불편을 초래하고 치료 효과를 낮추는 결과를 가져올 수 있다. 최근 차세대 염기서열 분석 기술을 활용한 다중(multiplex) 유전자 검사용 패널이 많이 개발되어 연구에 사용되고 있고, 임상 유용성에 대한 신뢰도 또한 높아지고 있다. 이러한 다양한 유전자 검사 패널 중 써모피셔(Thermo Fisher)의 차세대 염기서열 분석 기반 동반진단 ‘Oncomine Dx’가 2017년 6월 3가지(EGFR mutation, ROS1 fusion, BRAF mutation) 비소세포폐암 치료에 적합한 환자를 선별하는 동반진단으로 미국 FDA의 허가를 받았다. 우리나라에서도 2017년 3월부터 16종의 암(폐암, 유방암, 위암 등 고형암 10종, 혈액암 6종)에 대해 차세대 염기서열 분석(NGS) 기반 유전자패널 검사에 건강보험을 적용하기 시작했다. 이러한 차세대 염기서열 분석(NGS) 기술 이용 진단 검사의 발전은 유전자 패널과 진단 기술 자체의 고도화, 정밀화와 더불어, 현재는 조직을 넘어서 혈액이나 타액, 소변 등의 액체 생검을 이용한 진단 기술 개발로 그 방향이 확대되고 있다.
3. 결론
차세대 염기서열 분석(NGS)이 등장하면서부터 언급되었던 중요한 전환점은 $1,000의 인간 게놈 염기서열 분석이 가능하게 되는 시기였는데, 현재 급속한 기술의 발전으로 암 등의 질환 관련 유전자 검사에서 그 전환점에 이르렀고 유전자를 제외하고는 의학을 논할 수 없는 시대가 되었다. 최근 차세대 염기서열 분석(NGS) 기반 유전자패널들이 암에서 기존의 병리검사를 대체할 수 있는 표준검사법으로 대두되면서 차세대 염기서열 분석(NGS) 분야에 대한 연구 개발과 실용화는 더욱 가속화 될 것으로 예상된다. 현재까지는 표적 염기서열 분석(targeted sequencing)으로 수 십 개의 유전자에서 정해진 특정 표적 서열에 대한 분석만을 진행하지만 현재와 같은 기술의 발전 속도로 미루어 멀지 않은 미래에 모든 유전자에 대한 검사를 진행할 수 있는 기술 개발도 가능할 것으로 기대된다. 하지만 현재 차세대 염기서열 분석(NGS)에 사용되는 유전체 분석 장비와 패널, 분석 소프트웨어 등은 소수의 외국기업이 전세계 시장을 주도하고있고, 국내에서도 모두 이러한 회사의 제품에 의존해 차세대 염기서열 분석(NGS)이 이루어지고 있다. 향후 차세대 염기서열 분석(NGS) 기술의 높은 활용도가 기대되는 만큼 국산 기술의 개발을 위한 투자와 연구에 더 많은 관심과 전략이 필요할 것이다.
참고문헌
1. Collins FS, Varmus H. A new initiative on precision medicine. N Engl J Med. 2015 Feb 26;372(9):793-5.
2. Peplow M. The 100,000 genomes project. BMJ. 2016 Apr 13;353:i1757.
3. Cyranoski D. China’s bid to be a DNA superpower. Nature. 2016 Jun 23;534(7608):462-3.
4. 생명공학정책연구센터. 주요 국가별 유전체 분석 프로젝트 현황 (2015.10.2).
5. Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008 Oct;26(10):1135-45.
6. metzker ML. Sequencing technologies-the next generation. Nat Rev Genet. 2010 Jan;11(1):31-46.
7. Shao D et al. A targeted next0generation sequencing method for identifying clinically relevant mutation profiles in lung
adenocarcinoma. Sci Rep. 2016 Mr 3;6:22338.
8. Wang J, Song Y. Single cell sequencing: a distinct new field. Clin Transl Med. 2017 Dec;6(1):10.
9. Vargas AJ, Harris CC. Biomarker development in the precision medicine era: lung cancer as a case study. Nat Rev Cancer. 2016
Aug;16(8):525-37.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update