| 유전체 교정 시대의 도래와 전망 | |
|---|---|
| Date 2017-10-09 18:18:38 |
|

교수
한양대학교 화학과
sangsubae@hanyang.ac.kr
1. 서론
지난 1998년 에단 호크, 우마 서먼, 주드 로 주연의 ‘가타카(GATTACA)’ 라는 제목의 영화가 국내에 개봉된 적이 있다. 다소 이상한 영화제목은 DNA의 4가지 염기인 아데닌(A). 티민(T), 구아닌(G), 시토신(C)의 조합으로 만든 것인데, 유전공학 기술이 발달한 먼 미래를 배경으로 하고 있다. 영화는 자연임신으로 태어난 인간과 유전자 편집을 통해 우성인자들만 가지고 태어난 인간이 계층적으로 구분되어 살아가는 모습을 보여준다. 그런데 당시에는 예술인들의 상상력으로 구성한 내용들이 오늘날에 와서는 점차 현실이 되고 있다. 이는 바로 제3세대 유전자가위 기술로 일컬어 지는 ‘크리스퍼(CRISPR) 유전자가위’ 기술의 등장에 기인한다. 이미 2015년에 중국 연구진들에 의해 인간의 수정란인 배아세포에서 최초로 중증빈혈 질환과 관련한 유전자 교정이 성공한 바 있고, 올해 8월에는 한국 기초과학연구원(IBS)과 미국 오리건보건과학대(OHSU) 등이 공동연구를 통해 인간의 배아세포에서 심장질환을 유도한다고 알려진 MYBPC3 유전자의 변이를 성공적으로 교정한 연구내용이 학술지 <Nature>에서 발표되었다.[1] 이는 과학계 뿐 아니라, 사회, 법, 윤리 분야에 큰 충격을 주고 있는데, 특정 유전자의 변이에 기인한 유전질환에 대한 치료법으로서 기대감과, 정상적인 개체로 자랄 수 있는 인간 배아의 유전자를 교정하는 것에 대한 우려를 동시에 낳고 있다.
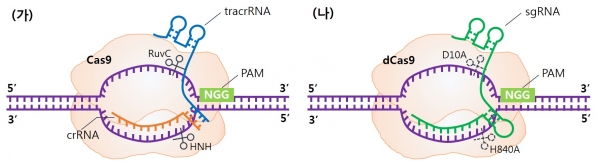
크리스퍼 유전자가위의 원리가 되는 CRISPR-Cas 시스템은 본래 원핵생물이 가지고 있는 후천적 면역체계(adaptive immunity)로서, 외부의 바이러스나 다른 원핵생물의 유전물질이 침투하면 이를 인지하고 잘라내는 역할을 한다. [2] 2012년에는 대표적인 시스템인 CRISPR-Cas9의 작동기작이 매우 자세히 밝혀졌는데, 그 특징은 표적유전자에 대한 상보적인 염기서열을 가지고 있는 crRNA와 crRNA에 상보적으로 결합하는 tracrRNA가 Cas9 단백질과 결합하여 Cas9이 표적 유전자를 인식하고 자르도록 유도한다는 점이다. [3] 즉, Cas9이라는 단백질과 이를 타겟 DNA에 인도해주는 가이드 RNA만 있으면, 특정 부위의 DNA를 인식해서 자를 수 있다는 내용으로, 그 구성인자들을 간단하고 쉽게 만들 수 있다. (그림 1)
그림 1. 크리스퍼 유전자가위의 모식도. (가) 자연계에서 발견된 크리스퍼 유전자가위의 구성인자. Cas9 단백질과 한 쌍의 가이드 RNA(crRNA and tracrRNA)로 구성되어 있다. (나) 개조된 크리스퍼 유전자가위의 구성 인자. 하나로 연결된 가이드 RNA (single guide RNA; sgRNA)로 작동 할 수 있고, 자르는 도메인을 망가뜨린 불활성 Cas9 단백질 (deactivated Cas9; dCas9)을 만들 수 있다.
그리고 1년도 채 안되어서 복수의 연구자들이 독립적으로 CRISPR-Cas9을 이용하여 인간세포를 포함한 동물세포에서 유전체 편집에 성공하였으며, 현재까지 미생물, 식물, 곤충 등 다양한 종에서 매우 폭넓게 이용되고 있다. [4-7] 본고에서는 최근까지 계속해서 개발되고 있는 크리스퍼 시스템을 이용한 유전체 교정 기술에 대해 설명하고, 그 의미에 대해 간단히 밝히고자 한다.
2. 본론
2.1 세포 본연의 DNA 수선 기작을 이용한 유전자 편집 크리스퍼 유전자가위는 기본적으로 특정 DNA 타겟을 자르는 것으로, 절단된 DNA는 이후 세포 스스로의 다양한 DNA 수선 기작(DNA repair pathway)에 의해 복구된다. 인간과 같은 고등세포에서는 DNA 수선은 주로 비상동 말단 부착(nonhomologous end joining; NHEJ)이라는 기작을 통해 이루어진다. 비상동 말단부착 기작은 종종 몇 개의 염기서열이 추가되거나 제거되는 삽입 및 결실(Indel)의 돌연변이를 유도하고, 이로 인해 유전자의 코돈 구성이 어긋나면 유전자의 기능이 망가지게 된다. 이를 이용하여 특정 유전자의 녹아웃(Knock-Out)을 유도할 수 있다. 한편, 크리스퍼 유전자가위에 의한 DNA 절단은 때때로 동형방식수선(homology directed repair; HDR) 기작을 통해 고쳐 지기도 한다. 이때는 동형 유전자(homologous DNA)조각을 주형으로 하여 돌연변이 없이 정확하게 DNA가 복구된다.이 과정에서, 외래의 원하는 염기서열의 DNA 공여체(donor DNA)를 다량으로 넣어주면 외래의 DNA 공여체를 기반으로 동형방식수선이 이루어진다. 이를 통해 유전자의 특정 돌연변이를 교정하거나 외래 유전자를 삽입하는 녹인(knock-in)을 유도할 수 있다.
2.2 다양한 종의 크리스퍼 유전자가위
현재 가장 널리 쓰이고 있는 크리스퍼 유전자가위는 Streptococcus pyogenes 종에서 유래한 CRISPR-Cas9 시스템으로, SpCas9이라는 단백질과 표적 유전자의 20개 염기서열을 인식하는 sgRNA로 구성된다. 이 유전자가위는 DNA에 있는 5’-NGG-3’의 PAM (protospacer adjacent motif)이라 불리는 염기서열을 인식해서 DNA에 결합하고 DNA를 자른다. 이를 이용하면 이론적으로 전체 유전체 내에서 1/8에 해당하는 부분을 특이적으로 자를 수 있게 된다.
SpCas9외에도 많은 종의 세균에서 유래한 다양한 변종들(orthologs)이 발견되고 있다. [8] 크게 분류하자면, 여러 단백질들이 모여 Cascade를 형성해야만 DNA를 인식하고 자르는 Class I과 하나의 단백질이 타겟 DNA를 인식하고 자를 수 있는 Class II 가 있다. 이 중에서 하나의 단백질로 작동하는 Class II가 유전체 편집에 이용하는 데 유리하다. Class II 크리스퍼 중에서 SpCas9은 Type II로 분류되는데, Type II는 DNA를 자르는 HNH domain과 RuvC domain이 있고, 평활 말단(Blunt end)을 만드는 특징이 있다. SpCas9 외에도 크기가 작아 Adeno-associated virus (AAV)벡터를 이용한 전달이 가능한 Staphylococcus aureus Cas9 (SaCas9)과 Campylobacter jejuni Cas9 (CjCas9)과, 다양한 PAM을 인식하는 Streptococcus thermophiles Cas9 (StCas9), Neisseria meningitides Cas9 (NmCas9),Treponema denticola Cas9 (TdCas9)등이 유전체 편집에 이용되고 있다.
한편, Class II 중에서 Cpf1 (Cas12a)은 Type II Cas9과 달리, tracrRNA없이 crRNA만으로도 타겟 DNA를 인식하고 자를 수 있고, 표적 DNA 타겟 염기서열의 시작부분(Upstream)에 있는 5’-TTTV-3’ 을 PAM으로 인식하는 특성이 있어, 이를 따로 Type V로 분류하였다. [9] 최근에는 Cpf1이 Cas9에 비해 더욱 표적에 특이적으로 작동하고, 표적이 아닌 곳(off-target site)에 작동하는 빈도가 적다는 연구결과가 보고되기도 했다. [10]
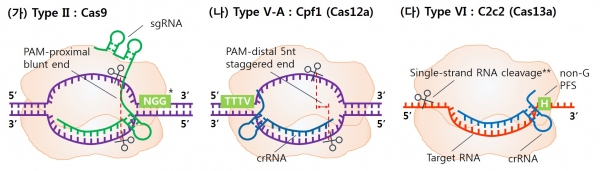
또한, 가장 최근에는 Class II 중에서 DNA 이중나선을 표적으로 하여 자르는 Cas9이나 Cpf1과 달리, 한 가닥 RNA를 표적으로 하여 자르는 C2c2 (Cas13a) 단백질이 발견되었는데 이를 새롭게 Type VI로 분류하였다. [11] C2c2는 C2c1 (Cas12b), C2c3 (Cas13c)와 함께 박테리아의 유전체 데이터 분석을 통해 예측되었는데, [12] Cpf1처럼tracrRNA를 필요로 하지 않는 특징이 있고, [13] 표적 RNA와 결합한 후에는 비 표적 RNA들까지 자르는 특성이 있어, 특정 바이러스를 검출하는 센서로 개발 되기도 하였다. [14] Class II 유전자가위의 여러 형태를 정리하면 위의 그림 2와 같다.
그림 2. Class II 크리스퍼 유전자 가위의 작동 모식도. (가) Cas9은 표적 DNA 염기서열 말단의 PAM을 인식하고, PAM과 가까운 위치의 DNA를 잘라서 평활 말단 (Blunt end)을 만든다. 종에 따라 PAM의 길이 및 서열이 다르다. *NGG 는 SpCas9의 PAM. (나)Cpf1은 표적 DNA 염기서열의 시작 부분 (Upstream)의 5’-TTTV-3’ PAM을 인식하고 PAM에서 먼 쪽의 DNA를 잘라 점착 말단(Staggered end)을 만든다. (다) C2c2는 표적 RNA와 결합하여 활성화되면 표적 RNA뿐 아니라 비표적 RNA도 자른다. **표적RNA가 잘리는 위치는 아직까지 명확하게 밝혀지지 않았다.
2.3 크리스퍼 유전자가위 변형을 통해 만든 새로운 유전자 교정 기술
크리스퍼 유전자가위는 기본적으로 가이드 RNA와 상보적인 타겟 DNA에 결합하여 이중 나선을 절단하는 방식을 일컫는다. 하지만, DNA를 절단하는 domain을 없애면, 특정 DNA에 결합할 수는 있지만 절단할 수는 없게 된다. 예를들어, SpCas9의 경우, RuvC domain과 HNH domain을 치환하여 망가뜨리면 DNA를 자르는 기능을 상실하고 표적유전자에 대한 특이적 부착 기능만 남은 deactivated Cas9 (dCas9)이 되어 새로운 형태의 유전체 편집 도구가 된다.(그림 1나) 이러한 dCas9에 특정 기능의 단백질을 결합하는 형태로 다양하게 응용 될 수 있다. [15]
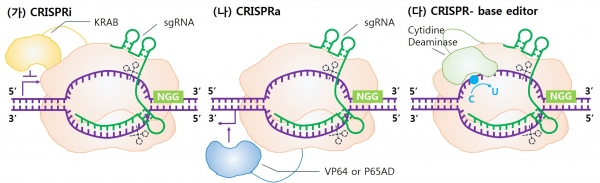
예를 들어, 그림 3가와 같이 dCas9에 kruppel-associated box (KRAB) 단백질을 부착하면 전사과정(Transcription)에서 RNA 중합 효소의 진행을 막아서 유전자의 발현을 억제 할 수 있는데, 이러한 CRISPR 기반의 유전자 발현 억제 기술을 CRISPR interference (CRISPRi)라고 부른다. [16] 반대로 그림 3나와 같이 dCas9에 VP64나 p65 activation domain (P65AD) 단백질을 결합하면 표적 유전자의 발현을 촉진 시킬 수 있는데, 이러한 기술을 CRISPR-mediated gene activation (CRISPRa)라고 부른다. 특히, CRIPSRa 기술은 다양한 분야에 응용할 수 있는데, Joshua B. Black 그룹에서는 Brn2, Ascl1, Myt1l 유전자(BAM factors)의 과발현을 통해 쥐의 배아 유래 섬유아세포(mouse embryonic fibroblast)를 신경세포로 직접 분화(direct reprograming)시키는데 성공하였다. [17]
그림 3. 크리스퍼 유전자가위 변형을 통해 만든 새로운 유전자 교정 기술 모식도. (가) 표적 유전자 발현을 억제하는 CRISPRi. (나) 유전자의 발현을 촉진시키는 CRISPRa. (다) DNA 절단 및 외래 DNA 공여체 없이 교정이 가능한 CRISP Rbase editing.
한편, 2.1절에서 서술한 바와 같이, 유전자의 특정 돌연변이를 원하는 염기서열로 정확하게 교정하기 위해서는 외래의 DNA 공여체를 넣어 동형방식수선(HDR) 기작을 유도해야 한다. 그러나 이는 효율이 매우 낮고(1% 내외), 외래의 DNA 공여체가 필요하다는 측면에서 어려움이 있다. CRISPR-base editor는 그림 3다와 같이 dCas9의 말단에Cytidine deaminase를 결합한 형태로 만들어 특정 위치의 C 염기를 T(또는 G 염기를 A)로 바꿀 수 있도록 제작되었다. [18,19] CRISPR-base editor는 DNA 두 가닥 절단을 일으키지 않아 위험성이 적을 뿐 아니라, 외래의 DNA 공여체의 추가 전달이 필요 없어 동물 실험에 유리하고, 작동 효율도 수 십 % 정도로 매우 높다는 장점이 있다. 앞으로CRISPR-base editor의 타겟 부위를 좀더 정밀하게 하거나, 교정할 수 있는 염기서열의 폭을 넓힌다면 더욱 강력한 유전체 편집 도구가 될 것으로 기대된다.
3. 결론
2013년 크리스퍼 유전자가위를 이용한 유전체 편집이 성공한 이후로 유전체 편집 관련 분야는 매우 빠르게 발전하고 있다. 또한 크리스퍼 유전자가위 기술의 높은 접근성은 유전체 편집 외의 다양한 분야에서 CRISPR 기술이 널리 쓰일 수 있도록 해주었다. 이러한 파급력을 바탕으로 CRISPR 시스템은 더욱 다양한 기술적 요구와 만나고, 그에 상응하는다양한 기술들이 개량되어 왔다. 이 분야가 하루가 다르게 발전하고 있는 만큼 본고에서 미처 다루지 못한 부분들이 있을 수 있다.
한편, 빠르게 발전한 유전체 교정 기술에 상응하여 법적, 제도적, 사회적 장치들이 아직 제대로 정립되지 못하고 있다. 유전체 교정 기술은 제대로 활용하면 유전자 치료, 새로운 식물 육종의 개발, 기초 유전자 연구 활용 등 그 가능성이 무궁무진 한 반면, 맞춤형 아기, 무분별한 편집 시도 등과 같은 부작용 또한 클 수 있다. 따라서 유전체 교정 기술을 올바르게 활용할 수 있도록 사회적 논의 및 합의가 필요할 것이다. 이는 시험관 아기 시술의 개발 및 적용과 유사하다. 시험관 아기 시술은 난자와 정자를 체외에서 수정시켜 자궁에 착상시키는 방법으로 일찍이 1978년 영국에서 최초로 시도되었고, 1985년 국내에 도입되었다. 현재에는 국내에서만 매년 수만 명의 아이가 이 시술로 태어나고 있는데, 처음 시도될 당시에는 여러 사회적, 윤리적 우려들이 있었던 것이 사실이다. 유전체 교정기술도 이와 마찬가지로, 그 장점 및 활용을 극대화 하여 사회에 기여할 수 있기를 기대한다.
참고문헌
1. H. Ma, N. Marti-Gutierrez, S.-W. Park, J. Wu, Y. Lee, K. Suzuki, A. Koski, D. Ji, T. Hayama, R. Ahmed, H. Darby, C. Van Dyken, Y.Li, E. Kang, A.R. Park, D. Kim, S.-T. Kim, J. Gong, Y. Gu, X. Xu, D. Battaglia, S.A. Krieg, D.M. Lee, D.H. Wu, D.P. Wolf, S.B.Heitner, J.C.I. Belmonte, P. Amato, J.-S. Kim, S. Kaul, S. Mitalipov, Correction of a pathogenic gene mutation in human embryos,Nature advance online publication (2017).
2. L.A. Marraffini, E.J. Sontheimer, CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea, Nat Rev Genet 11(3) (2010) 181-190.
3. M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, J.A. Doudna, E. Charpentier, A Programmable Dual-RNA.Guided DNA Endonuclease in Adaptive Bacterial Immunity, Science 337(6096) (2012) 816-821.
4. S.W. Cho, S. Kim, J.M. Kim, J.-S. Kim, Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease,Nat Biotech 31(3) (2013) 230-232.
5. L. Cong, F.A. Ran, D. Cox, S. Lin, R. Barretto, N. Habib, P.D. Hsu, X. Wu, W. Jiang, L.A. Marraffini, F. Zhang, Multiplex Genome Engineering Using CRISPR/Cas Systems, Science 339(6121) (2013) 819-823.
6. P. Mali, L. Yang, K.M. Esvelt, J. Aach, M. Guell, J.E. DiCarlo, J.E. Norville, G.M. Church, RNA-Guided Human Genome Engineering via Cas9, Science 339(6121) (2013) 823-826.
7. W. Jiang, D. Bikard, D. Cox, F. Zhang, L.A. Marraffini, RNA-guided editing of bacterial genomes using CRISPR-Cas systems, Nat Biotech 31(3) (2013) 233-239.
8. A. Cebrian-Serrano, B. Davies, CRISPR-Cas orthologues and variants: optimizing the repertoire, specificity and delivery of genome engineering tools, Mammalian Genome 28(7) (2017) 247-261.
9. B. Zetsche, Jonathan S. Gootenberg, Omar O. Abudayyeh, Ian M. Slaymaker, Kira S. Makarova, P. Essletzbichler, Sara E. Volz, J. Joung, J. van der Oost, A. Regev, Eugene V. Koonin, F. Zhang, Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System, Cell 163(3) (2015) 759-771.
10. D. Kim, J. Kim, J.K. Hur, K.W. Been, S.-h. Yoon, J.-S. Kim, Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells, Nat Biotech 34(8) (2016) 863-868.
11. S. Shmakov, A. Smargon, D. Scott, D. Cox, N. Pyzocha, W. Yan, O.O. Abudayyeh, J.S. Gootenberg, K.S. Makarova, Y.I. Wolf, K. Severinov, F. Zhang, E.V. Koonin, Diversity and evolution of class 2 CRISPR-Cas systems, Nat Rev Micro 15(3) (2017) 169-182.
12. S. Shmakov, O.O. Abudayyeh, K.S. Makarova, Y.I. Wolf, J.S. Gootenberg, E. Semenova, L. Minakhin, J. Joung, S. Konermann, K. Severinov, Discovery and functional characterization of diverse class 2 CRISPR-Cas systems, Molecular cell 60(3) (2015) 385-397.
13. O.O. Abudayyeh, J.S. Gootenberg, S. Konermann, J. Joung, I.M. Slaymaker, D.B.T. Cox, S. Shmakov, K.S. Makarova, E.Semenova, L. Minakhin, K. Severinov, A. Regev, E.S. Lander, E.V. Koonin, F. Zhang, C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector, Science 353(6299) (2016).
14. J.S. Gootenberg, O.O. Abudayyeh, J.W. Lee, P. Essletzbichler, A.J. Dy, J. Joung, V. Verdine, N. Donghia, N.M. Daringer, C.A. Freije, C. Myhrvold, R.P. Bhattacharyya, J. Livny, A. Regev, E.V. Koonin, D.T. Hung, P.C. Sabeti, J.J. Collins, F. Zhang, Nucleic acid detection with CRISPR-Cas13a/C2c2, Science 356(6336) (2017) 438-442.
15. A.A. Dominguez, W.A. Lim, L.S. Qi, Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation,Nat Rev Mol Cell Biol 17(1) (2016) 5-15.
16. Luke A. Gilbert, Max A. Horlbeck, B. Adamson, Jacqueline E. Villalta, Y. Chen, Evan H. Whitehead, C. Guimaraes, B. Panning,Hidde L. Ploegh, Michael C. Bassik, Lei S. Qi, M. Kampmann, Jonathan S. Weissman, Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation, Cell 159(3) (2014) 647-661.
17. Joshua B. Black, Andrew F. Adler, H.-G. Wang, Anthony M. D’Ippolito, Hunter A. Hutchinson, Timothy E. Reddy, Geoffrey S. Pitt,Kam W. Leong, Charles A. Gersbach, Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells, Cell Stem Cell 19(3) (2016) 406-414.
18. A.C. Komor, Y.B. Kim, M.S. Packer, J.A. Zuris, D.R. Liu, Programmable editing of a target base in genomic DNA without doublestranded DNA cleavage, Nature 533(7603) (2016) 420-424.
19. K. Nishida, T. Arazoe, N. Yachie, S. Banno, M. Kakimoto, M. Tabata, M. Mochizuki, A. Miyabe, M. Araki, K.Y. Hara, Z. Shimatani, A. Kondo, Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems, Science 353(6305) (2016).
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update